POJ-2778 DNA Sequence AC自动机+矩阵二分
题目链接:http://poj.org/problem?id=2778
[摘自Matrix67] 题目大意是,检测所有可能的n位DNA串有多少个DNA串中不含有指定的病毒片段。合法的DNA只能由ACTG四个字符构成。题目将给出10个以内的病毒片段,每个片段长度不超过10。数据规模n<=2 000 000 000。
下面的讲解中我们以ATC,AAA,GGC,CT这四个病毒片段为例,说明怎样像上面的题一样通过构图将问题转化为例题8。我们找出所有病毒片段的前缀,把n位DNA分为以下7类:以AT结尾、以AA结尾、以GG结尾、以?A结尾、以?G结尾、以?C结尾和以??结尾。其中问号表示“其它情况”,它可以是任一字母,只要这个字母不会让它所在的串成为某个病毒的前缀。显然,这些分类是全集的一个划分(交集为空,并集为全集)。现在,假如我们已经知道了长度为n-1的各类DNA中符合要求的DNA个数,我们需要求出长度为n时各类DNA的个数。我们可以根据各类型间的转移构造一个边上带权的有向图。例如,从AT不能转移到AA,从AT转移到??有4种方法(后面加任一字母),从?A转移到AA有1种方案(后面加个A),从?A转移到??有2种方案(后面加G或C),从GG到??有2种方案(后面加C将构成病毒片段,不合法,只能加A和T)等等。这个图的构造过程类似于用有限状态自动机做串匹配。然后,我们就把这个图转化成矩阵,让这个矩阵自乘n次即可。最后输出的是从??状态到所有其它状态的路径数总和。
题目中的数据规模保证前缀数不超过100,一次矩阵乘法是三方的,一共要乘log(n)次。因此这题总的复杂度是100^3 * log(n),AC了。
Example:
2 3
AG
CG
状态转移图:
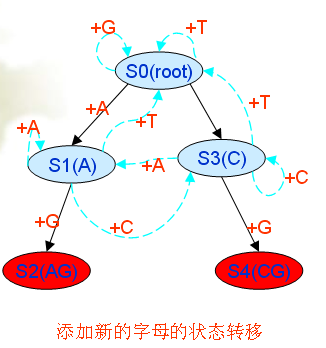
要注意当前前缀 i 匹配的前缀 j 可能不是可行的前缀。
1 //STATUS:C++_AC_313MS_624KB 2 #include<stdio.h> 3 #include<stdlib.h> 4 #include<string.h> 5 #include<math.h> 6 #include<iostream> 7 #include<string> 8 #include<algorithm> 9 #include<vector> 10 #include<queue> 11 #include<stack> 12 #include<map> 13 #include<set> 14 using namespace std; 15 //define 16 #define pii pair<int,int> 17 #define mem(a,b) memset(a,b,sizeof(a)) 18 #define lson l,mid,rt<<1 19 #define rson mid+1,r,rt<<1|1 20 #define PI acos(-1.0) 21 //typedef 22 typedef __int64 LL; 23 typedef unsigned __int64 ULL; 24 //const 25 const int N=101; 26 const int INF=0x3f3f3f3f; 27 const int MOD=100000,STA=8000010; 28 const LL LNF=1LL<<60; 29 const double EPS=1e-8; 30 const double OO=1e15; 31 //Daily Use ... 32 template<class T> T gcd(T a,T b){return b?gcd(b,a%b):a;} 33 template<class T> T lcm(T a,T b){return a/gcd(a,b)*b;} 34 template<class T> inline T Min(T a,T b){return a<b?a:b;} 35 template<class T> inline T Max(T a,T b){return a>b?a:b;} 36 template<class T> inline T Min(T a,T b,T c){return min(min(a, b),c);} 37 template<class T> inline T Max(T a,T b,T c){return max(max(a, b),c);} 38 template<class T> inline T Min(T a,T b,T c,T d){return min(min(a, b),min(c,d));} 39 template<class T> inline T Max(T a,T b,T c,T d){return max(max(a, b),max(c,d));} 40 //End 41 42 int idx[N]; 43 int ch[N][4]; 44 int val[N],f[N],sta[N]; 45 int sz,n,m; 46 int size; 47 48 struct Matrix{ 49 LL ma[N][N]; 50 Matrix friend operator * (const Matrix a,const Matrix b){ 51 Matrix ret; 52 mem(ret.ma,0); 53 int i,j,k; 54 for(k=0;k<size;k++) 55 for(i=0;i<size;i++) 56 for(j=0;j<size;j++) 57 ret.ma[i][j]=(ret.ma[i][j]+a.ma[i][k]*b.ma[k][j])%MOD; 58 return ret; 59 } 60 }ans,mta; 61 62 void mutilpow(LL k) 63 { 64 int i,j; 65 mem(ans.ma,0); 66 for(i=0;i<size;i++) 67 ans.ma[i][i]=1; 68 for(;k;k>>=1){ 69 if(k&1)ans=ans*mta; 70 mta=mta*mta; 71 } 72 } 73 74 void init(){sz=1;mem(ch[0],0);} 75 76 void insert(char *s,int v){ 77 int i,len=strlen(s),id,u=0; 78 for(i=0;i<len;i++){ 79 id=idx[s[i]]; 80 if(!ch[u][id]){ 81 mem(ch[sz],0); 82 val[sz]=0; 83 ch[u][id]=sz++; 84 } 85 u=ch[u][id]; 86 } 87 val[u]=v; 88 } 89 90 void getFail() 91 { 92 int u,v,r,c; 93 queue<int> q; 94 mem(mta.ma,0); 95 f[0]=0; 96 sta[0]=size++; 97 for(c=0;c<4;c++){ 98 u=ch[0][c]; 99 if(u){ 100 f[u]=0;q.push(u); 101 if(!val[u]){ 102 sta[u]=size++; 103 mta.ma[0][sta[u]]++; 104 } 105 } 106 else mta.ma[0][0]++; 107 } 108 while(!q.empty()){ 109 r=q.front();q.pop(); 110 if(sta[r]==-1)continue; 111 for(c=0;c<4;c++){ 112 u=ch[r][c]; 113 if(!u){ 114 v=ch[r][c]=ch[f[r]][c]; 115 if(sta[v]==-1)continue; 116 mta.ma[sta[r]][sta[v]]++; 117 } 118 else { 119 q.push(u); 120 v=f[r]; 121 while(v && !ch[v][c])v=f[v]; 122 f[u]=ch[v][c]; 123 if(val[u] || sta[f[u]]==-1)continue; 124 sta[u]=size++; 125 mta.ma[sta[r]][sta[u]]++; 126 } 127 } 128 } 129 } 130 131 int main() 132 { 133 // freopen("in.txt","r",stdin); 134 int i,j; 135 LL sum; 136 char s[20]; 137 idx['A']=0;idx['C']=1;idx['T']=2,idx['G']=3; 138 while(~scanf("%d%d",&m,&n)) 139 { 140 init(); 141 for(i=0;i<m;i++){ 142 scanf("%s",s); 143 insert(s,1); 144 } 145 146 mem(sta,-1); 147 size=0; 148 getFail(); 149 mutilpow(n); 150 for(i=sum=0;i<size;i++){ 151 sum+=ans.ma[0][i]; 152 } 153 154 printf("%I64d\n",sum%MOD); 155 } 156 return 0; 157 }