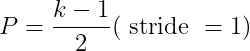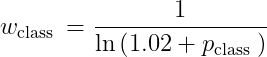彻底讲透U-net医学影像分割-小样本
U-net原文 《2015_Ronneberger_Cite=49316_U-net: Convolutional networks for biomedical image segmentation》
铺垫和引入
医学影像分割
医学影像分割的目的是将医学图像中具有某些特殊含义的部分分割出来,并提取相关特征。
医学上的分割准确率比其他领域如交通领域的分割准确率要求的高很多,
概况
处理对象:各种不同成像机理的医学图像,主流的有X射线成像(X-CT)、核磁共振成像(MRI)、核医学成像(NMI)、超声波成像(UI)、电子显微镜成像(EM)、
应用思路:首先对二维切片图像进行分析和处理,实现对人体器官、软组织和病变体的分割提取,然后进行三维重建甚至定量分析,从而大大提高医疗诊断的准确性和可靠性。
医学影像分割的 难点
- 医学分割的数据量往往比较小,这导致了训练起来很困难。比如一场医学影像的挑战赛仅仅提供不到100张训练数据,ImageNet有一百万例,咋训练啊?这么少的数据,对于深度学习模型是很容易过拟合的。比如几万次的脑部CT图才仅仅有十几个脑部肿瘤的照片,我们拥有大量健康大脑的CT图,但是患有肿瘤的CT图样本量太少了,样本不均衡,很难训练。
- 图片的尺寸太大了。单张图片尺寸大、分辨率高,对模型处理速度有较高要求。
- 对于分割结果的准去度要求极高。医学影像边界模糊、梯度复杂,很难分割的很准确。但是毕竟涉及病情诊断,要求准确率高也是应该的。
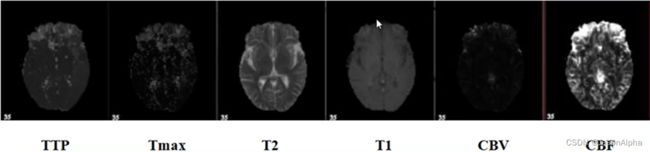
- 数据有可能是多模态的。比如ISLES脑梗竞赛,官方不仅仅提供了一种形式的数据,提供了用许多其他医学成像的方法拍出来的成像图,比如CBF MTT CBV TMAX CTP。比如下面这个,六张图都是排的一个大脑,六张图都多多少少都有你要用的信息,如何把六张图一起用,而且用的尽可能的好,这就是个多模态的问题。
U-net的研究成果及意义
- 分数高:赢得了ISBI cell tracking challenge 2015
- 速度快:对一个512 × 512的图像,使用一块GPU仅仅用来不到1秒的时间
- 成为大多做医疗影像语义分割任务的baseline,启发大量研究者去思考U型语义分割网络
- U-net结合了低分辨率信息和高分辨率信息,完美适用于医学影像分割。低分辨率信息提供物体类别识别依据,高分辨率信息提供精确分割定位依据。
收缩路径contracting path to capture context,扩张路径symmetric expanding path that enables precise localization
Introduction
Ciresan et al. 在滑动窗口sliding window设置中训练网络,以某一像素的领域(local region)(一个像素的正方形领域也就是一个patch)作为输入,用于预测每个像素的类别标签。网络的优点有:
(1)网络具有局部感知能力。
(2)用于训练上的样本数量远大于训练图像的数量。(我觉得,他这样把图片切成小方框,就是为了增大训练样本的数量)
(3)该网络获得了EM分割挑战赛ISBI 2012冠军。
该网络的不足:
(1)该网络运行效率很慢,对于每个邻域,网络都要运行一次,且对于领域重叠overlapping patches的部分,网络进行重复运算,实际上这些计算都是冗余redundancy
(我们滑动窗口是以某像素作为邻域作为输入,随着像素点向右滑动,以前在左边是边缘的东西慢慢变成了中心,继续滑动又从中心变成了右边,那么在这个过程里,最开始在像素点左边的这个区域被分割了不止一次,这就好比一个学生重复做他做过的题、已经掌握的题,请问意义在哪呢?)
(2)网络需要在精确的定位和获取上下文信息之间进行权衡(意思就是二者是矛盾的,无法兼顾)trade-off between localization accuracy and the use of context。
越大的patch需要越多的最大池化层(为啥需要?为了降低参数量吗?其实我不知道),这会降低定位的准确度localization accuracy。(大的patch可以捕捉到更多的上下文信息,涉及的面广,自然可以兼顾更多)
而小的patch使得网络获取较少的上下文信息。
U-net 通过弹性变形elastic deformation的方式增加数据量。(前面提及的镜像反转mirroring是弹性变形的一种)。这种方法在生物医学图片中是是十分重要的,因为这种方法模拟了真实的场景,变形是生物细胞组织当中最常见的变化,真实的变形可以有效的模拟这种形变。因此这种数据增强是不会破坏图片的原有结构的,还可以给模型的鲁棒性带来一定的帮助。
你在做实验的时候,如果你对的模型特别有自信,你就可以选择不同场景的数据集,全都做一遍训练,常见的有(1)道路Cavid(2)室内 SegRGBD,因为比较小,不要用NYU,因为这个data特别的imbalance,会导致你的分数很低(3)自然 PACVOC(4)医学。‘’
U-net对于FCN的改进之处
改进带来的优势
(1)仅仅需要少量图片就可以工作(FCN那个训练的时候用了挺多图片才训练成功并work的)
(2)分割准确率比FCN更高
如何改进的:
- 设计了一个完全对称的U型结构,可以更好的融合图片特征
- 在上采样部分也包含了大量特征通道,使网络能够将全局信息传播到更高的分辨率层。
- 采用镜像输入图像的方式进行补全缺失内容(就是前面讲的Overlap-tile strategy)
- 设计了加权损失函数
改进1和2——U型结构和跨层数据融合
前置知识
Padding
用于调整从输入到输出的图片尺寸大小的。
应对的问题
它用于去应对卷积操作中存在的什么问题?
- 卷积操作是的图像越变越小
- 图像的边界信息丢失,图像角落和边界的信息发挥作用小(道理很简单,处于图片中央的像素点,被上下左右的经过的卷积核都卷了,但是比如处于正上方的、也是处于边缘的像素点,他只被左、右、下方的卷积核卷过少了上方卷积这个方向。自然传递到下一层,图片中央的像素点的信息被保留的更多,处于边缘位置的像素点的信息保留的少)
怎么做
Padding是怎么做的?
在图像块上下左右加格子,可以补一行、可以补多行,格子里填0.
作用
padding的作用:
- 可以使得输出图片的size和输入一致,针织可以配合卷积做deconvolution来放大图片。
- 对于处于图片边缘的那部分信息,尽可能多的去保留这部分信息。(但是如果你padding了太多行或者列,这么多0参与卷积运算,会使得保留下来的边界信息变得很微弱、稀疏。这种情况你比较在意的话,建议使用mirroring边界镜像反转填充,起码镜像砌几层围墙里面的数字都是真实的值。)
三种卷积方式
Padding情况下的三种卷积方式
(1)full:——卷积核和image有一点点相交就进行卷积,(不必等到卷积核的核心那一小块与图像相交)
(2)same:——当卷积核的中心和image的边缘一点点重合了,就开始卷积。
(3)valid:——只有卷积核的全部格子和图片都重合了,才开始卷积,相当于padding没有用。
第二种same是最常用的,因为第一种在图像的边缘计算,太多的0参与进去卷积操作,但是对于在图片中心的像素点,3×3的卷积核9个元素人家乘出来起码0 特别少吧,那么边缘的卷积运算结果比起中心位置的结果要小特别多。因此在padding保证了边缘参与卷积运算次数多的情况下,我希望更多的非零元素参与卷积运算,因此第二种same是最好的。
卷积运算后output的维度的公式
in个维度的输入图片,先paddingP列或行,然后拿着kernel size = k的卷积核,每s步卷积一次,得出的输出的feature map的维度是下面这样
如果要使得输入和输出完全相等,也就是卷积结束后图片的尺寸没有发生变化,且在步长stride=1的情况下,padding填充几行或几列呢?下面这个公式
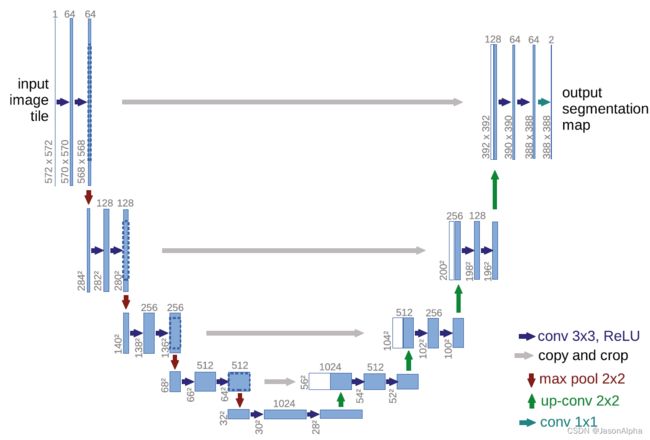
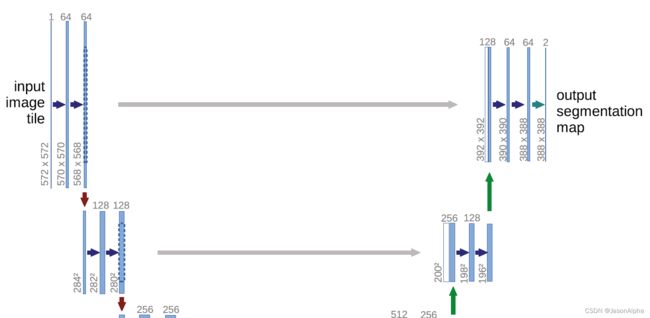
算法架构
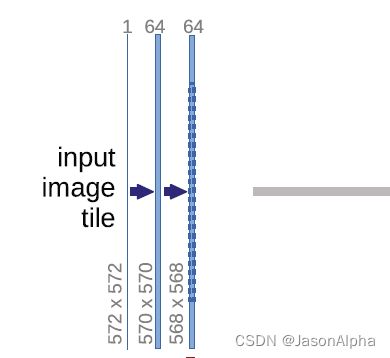
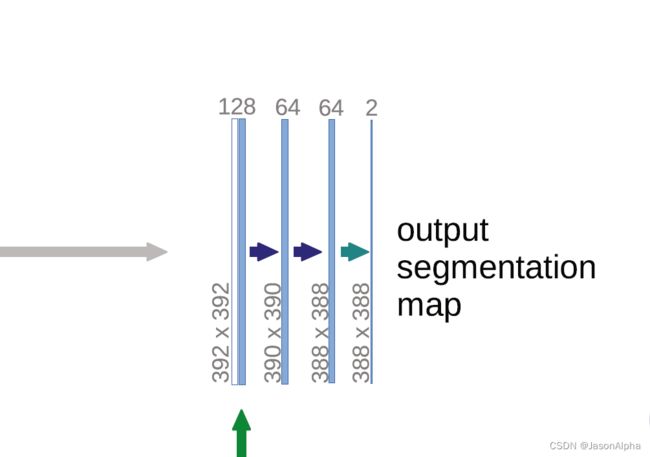
输出是388×388的图片,根据端到端end to end的原则,输入图片也应该是388×388的呀,但是我们发现输入图片的尺寸是572×572,这是怎么回事?原图是388×388的,通过镜像翻转,向四周翻转一定尺寸,得到了572×572尺寸的图片作为输入。
输入图片的维度是572×572,channel数为1,因为是灰度图,不是彩图(彩图channel数为3)
再去看输出的维度388×388,channel数为2,表示语义分割只有两个类别。这也是医学这块的特色,类别只有两个。也因此出现了一些具有医学特色的损失函数,只做二分类的。
input进来的维度是572×572,然后经过一个3×3的卷积和ReLU,得到一个570×570,channel数为64的输出。channel为64的意思是,用64个不同的卷积核做的卷积运算,出来有64个feature map,因此channel为64。请问这个维度是怎么从572变到570的?
带入上面讲过的这个公式,kernel size = 3,in = 572,out = (572 + 2×0 - 3) / 1 + 1=570。因此 padding为0这里没做padding,stride=1这里是一步一个卷积走的
从第二层的570到568的变化是下面这样得来的。out =( 570+2×0-3)/ 1 + 1 =568。
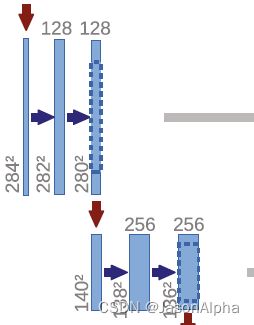
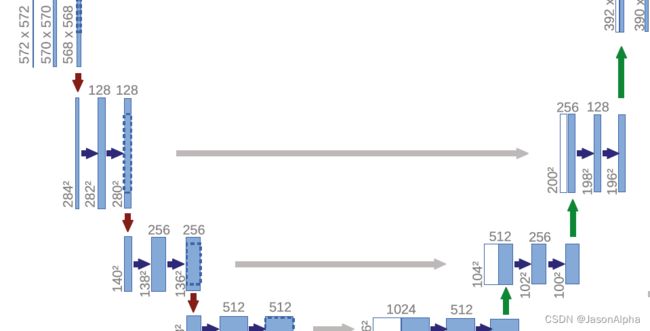
然后这个向下指的红色箭头表示max pooling,2×2的,自然使得feature map的size减半。所以从上一层的568×568直接减半成了284×284
然后后面从284变到282,再变到280都是和上面一样,padding=0,stride=1,怎么算上面演示两次了。feature map的size缩小了一半,但是channel扩大了一倍,从64变成了128
(大家都用的一个准则就是经过一次上采样,channel数减半;经过一次下采样,channel数扩大一倍)
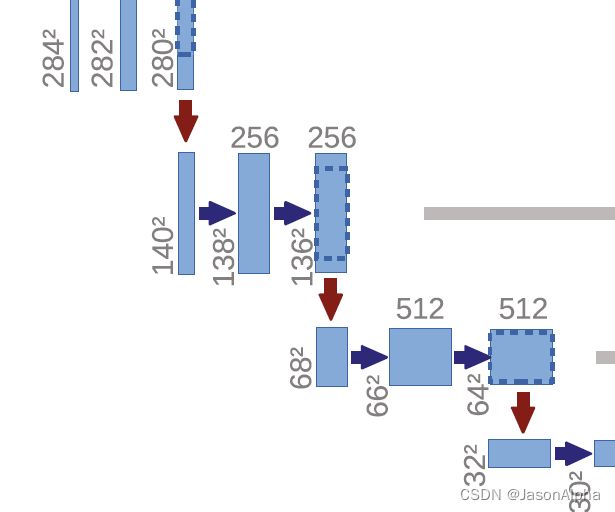
再说下一个下采样。通过max pooling,feature map的size从280×280减半变成140×140.后面还是老样子,padding=0,stride=1的3×3的卷积核走两次,feature map变成了136×136。
channel数不用我说,很自然的随着feature map的size减半,channel数从原来的128放大到了256。
再经过一次下采样,136减半变成68,channel数从256放大二倍到512。然后两个3×3的kernel size=3的卷积stride=1的卷积运算,从68降低到66吗,然后64.channel数保持不变。
最后一次下采样,从64缩减一半到32,channel数扩大两倍从512变到1024。然后继续padding=0,stride=1的两从3×3的卷积,从32到30最后到28。
我个人觉得上面这种“卷积卷积池化”这种卷积搭积木的方式是模仿了VGG的搭模型思路
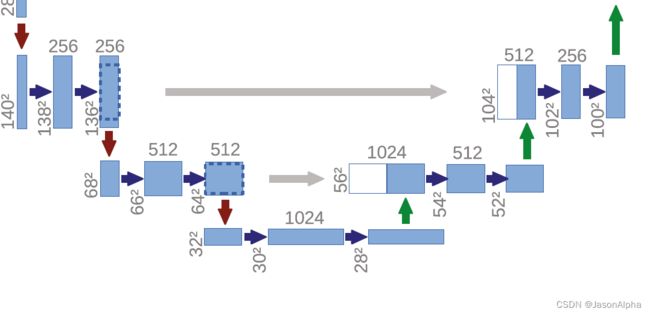
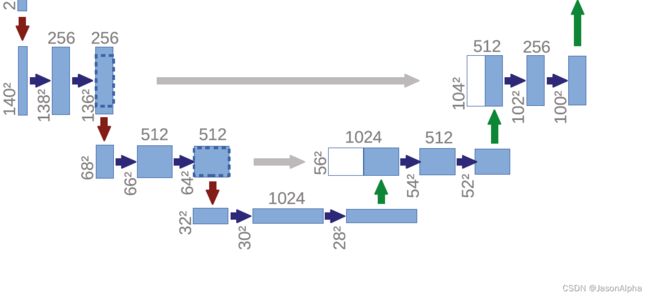
前面带着大家走完了U-net这个U形网络的左侧,从上向下滑滑梯的过程。下面带着大家从U型结构的右侧,从谷底爬上U型的右侧。向右爬的这个过程中,除了传统艺能的上采样,还有一个特色,就是会从U型的左侧那数据过来进行数据融合,所以U型的中间才有四个灰色的箭头从左边指向右边。
然后上采样将28放大到56.(注意,他上面画的这个图,有歧义,上采样之后的size为56,而不是图中离得很近的那个54,54指的是右边那个位置的维度)。
灰色箭头,左边这个拿过来做数据融合的东西size为64×64,远远大于右边的56×56。所以先做crop裁剪,把比56×56多的的那一圈裁掉,也变成56×56。
上采样这一步,size扩大一倍从28变成了56,channel数也随之跌去一半从1024变成了512。拿过来做拼接的那组左边的数据本身的channel数也是512.
然后,这两组数据咋融合呢?官方说法叫concate拼接。实际意思是,单纯把两组数据直接叠在一起,扩大channel数,不加哦。channel数就从512变成了1024.
channel数如果保持1024的话,参数量会大,因此下一步把channel数又恢复成512。卷积的时候实现的。
两次卷积,padding=0,stride=1,feature map的size从56降低到54,再到52。
然后反卷积上采样,size从52扩大一倍到104,channel数减半变成256。
然后从左边拿过来136×136 channel数=256的数据过来做融合。老样子,把136×136的图片裁剪成104×104的图片.那么咋融合呢?左边拿过来的数据是channel为256的,下面上采样出来的数据的channel是256,两组图片直接做叠加,数据内部不做加减乘除,保留各自每张feature map上的数据不变。最后channel数为256+256=512。
然后特征图的size经过两次3×3卷积,padding=0,stride=1,size缩小两次从104到102到100
现在有个问题究竟我咱能知道是哪一层和哪一层融合呢?
很简单。右边上采样出来的channel数你拿到了,去左边找channel数字一样的就是对应融合的。比如第三个箭头,左边的channel数为128,右边融合前拿到的下面送上来的数据的channel也是128(不然怎么能叠加出channel为256的层呢?)
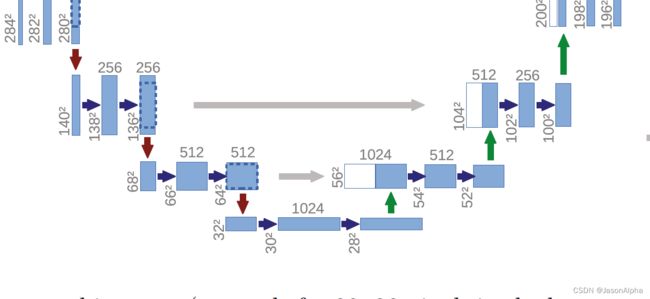
然后反卷积上采样,size从100扩大到200,channel数减半从256变到128.然后和左边280×280channel为12的数据进行融合(前提是crop到200乘200),简单叠在一起,channel数字变成128+128这么大,然后卷积一次channel数降到128然后再维持一次。
两次卷积,卷积核为3×3,stride=1,padding=1,size两次下降,从200到198到196.
两次卷积,channel数每次减半,从256到128,然后继续128
下面是最后一块了
196×196size的channel为64的这一块数据,经过上采样size放大两倍为392×392,channel缩减一半从128变为64。然后把左边的568×568的图片裁剪成392×392的图片,左边的channel数为64。两个channel为64的393×392的图片单纯的叠在一起,出来一个392×392,channel为128的数据,完成了融合。
然后下一层是一个卷积,再把channel数从128降低到64,下一层卷积,channel数保持不变。
然后紧接着两个卷积层,stride=1,padding=0,size不断缩小从392到390到388
最后一个层是一个1×1的卷积层,有padding,size不变,channel数变成2,实现了二分类(最后是几分类,最后的channel数就是几)
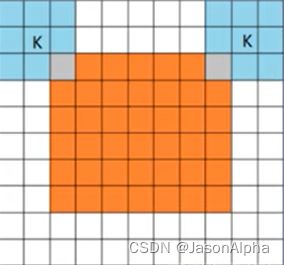
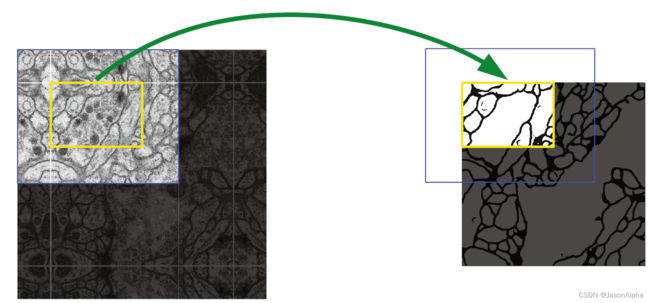
改进3——镜像补全图像Overlap-tile strategy
处于边缘的像素点经常分类错误或者说模糊分割的原因:一个处于图片中心位置的点,上下左右都是可以参考的信息,通过观察他们,也就知道了自己大概是啥。但是对于那些处于边缘位置的点,比如一个处于图片最上方中间的的一个像素,可以参考的点只有自己下面的点,自己上面是就是空白了,相比处于中心的点,起码少了一半的可参考信息。处于边界的分割点在分割的时候,是没有一个参考的,可参考的上下文context是少的。
Overlap-tile strategy:输入比输出大一圈,输入外面那一大圈都不直接用,都是为了给处于右图patch边缘的这些像素点提供contex信息的。实际操作是大概有个宽度,一边缘为翻转轴做镜像翻转mirroring(你仔细看下面左边这张图,左上角,黄框外边缘多出来这一块和黄框内的纹理是对称,右边、下边不是对称的),翻转出四面的这个想厚镜框一样的东西。tile是瓦片瓷砖的意思。
改进4——加权损失函数
为什么要加这个东西?
U-net试图对抗的另一个challenge是in many cell segementation tasks, another challenge is the separation of touching objects of the same class.分割连在一起的细胞很困难。因为一个有一点点过拟合的模型很容易把两个连在-互相接触-的细胞分割成一个细胞。那怎样处理,才能把长得很像、物理距离又很近的、贴在一起的两个细胞t分开或给他们一个明确的边界划分呢?
具体怎么加 ?
为了解决这个问题,作者引入了一个加权的损失函数。连接细胞之间的那些背景获得了一个较大的权重,从而让模型集中注意力在那些贴的很近的细胞。对于分散的那些细胞(彼此之间距离很远的那些细胞),给他那个附近的权重小一点。让模型把注意不要放在那些好分割的地方,把更多的注意力放在贴的很近不好分割的地方,着重突破难点。
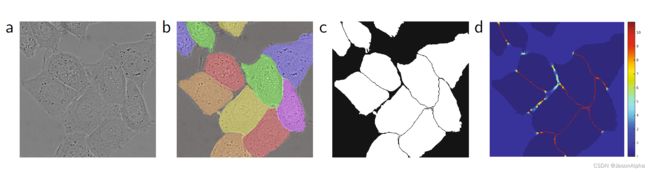
加完以后,图片上长啥样?
这个权重,作者下面这幅图图片上也展示了。第一幅图a是原始图表。第二幅图是标注出来的实例分割图,每个颜色不同的不规则形状块代表不同的细胞。第三幅图是mask掩码图,是制作出第四幅图的基础。首先第三幅图中,非细胞的区域比如背景用黑色表示,细胞区域用白色表示。细胞和大面积的背景交界的地方我们不感兴趣,因为这是细胞与细胞之间隔得比较远的地方。我们更加关心的是细胞与细胞交界的地方,就是图中细胞与细胞接触的地方那条细细的黑色的线,我们会在第四幅图里面把这些细细长长的黑线用红色标注出来。
第四幅图。越红表示权重越大-格外关注,图中细胞接触的地方最紧密,就越红。越蓝表示权重越小,大多数地方都是蓝色的,因为不在边界上。
用数学公式怎么表达?
d1(x)表示图中某一背景像素点到离这个点最最近(排名第一近)的细胞边界的距离。denotes表示the distance to the border of the nearest cell
d2(x)表示离这个像素点第二近的细胞的距离。the distance to the border of the second nearest cell
那么我问你为啥要设计这个d1(x)和d2(x),理由是啥?(1)这个像素点如果和第一近的像素点和第二近的像素点的距离都很小,说明这个像素点和两个细胞都挨的很近,也就是说这个点在两个细胞紧密交接的地带,因此应该分配更高的权重,多多花精力在分割这些地方。(2)如果一个像素点,d1(x)和d2(x)都很大,说明它在远离细胞的背景里,因此给他分配一个低的权重,少花甚至不花精力在这些区域的分割。
wc:常规的样本加权方式,the weight map to balance the class frequency,其实就是样本多的让他权重小一点,样本少的让它权重大一点。w class这个公式就是告诉你具体是怎么平衡的吗,这个公式。
整个公式想说明的是:在细胞边界比较近的像素点给予大一些的权重(这会使得在这个区域的细胞分割的更准确),对离细胞比较远的像素点赋予较小的权重。
参数初始化
在卷积神经网络中,尤其是这种做了很多次跨层特征融合的网络, a good initialization of the weights is extremely important,参数初始化十分重要。否则网络的默写部分可能过度激活,而网络的其他部分可能不发挥作用。如果你用随机初始化,是会存在上面这些风险的。
调整初始权重的值,使网络当中的每个特征图是具有近似的单位方差的,让特征图之间的差距不那么大。
从standard deviation为 根号下2/N的高斯分布中随机抽取数字作为权重参数。N表示的是denotes the number of incoming nodes of one-neuron(一层网络里面的神经元的个数),比如一个3×3的卷积,channel in the previous layer数为64,N = 3×3×64=576
医学分割指标
(1)Pixel error: 比较预测的label和实际的label,找出错误的点,用错误的点除以总数,就是像素误差。
(2)Rand error:它是将Rand index进行改造以后用来衡量分割性能的。Rand index用于衡量两组数据聚类的相似性。给定一张图片S,有n个像素点,同时有两个分割X和Y(实际和预测)
a表示两个分割同属于一个聚类的像素点
b表示两个分割不属于一个聚类的像素点的数量
Rand Index用于衡量相似度的,越高越好。所以Rand error就是RE = 1 - RI
(3)Warping error:在我看来就是求你分类的结果和实际的label之间的Hamming Distance,求二者的相似度。官方的说法是warping error用于衡量分割目标的拓扑形状效果。