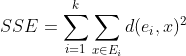K-means聚类算法
目录
简介
对距离的度量及SSE
问题及如何避免
k-means示例
k值选取
肘方法
可视化方法
TSNE
雷达图
总结一些对K-means聚类算法的理解。
K-means是一种聚类分析方法,聚类分析即是在没有给定划分类别的情况下,根据数据自身的相似度对样本数据进行分组的一种方法。
K-means也是一种无监督的聚类算法,所谓无监督,即算法需要我们给出分类的数量k,算法本身是不知道需要聚成多少类的(这点可以从K-means的计算步骤看出),即k是K-means 聚类中的超参数,下面先说明K-means的具体内容,再谈k值的选择。
简介
| 方法名称 | K-means |
|---|---|
| 所属方法 | 聚类分析 |
| 监督or非监督 | 非监督 |
| 输入 | 一组未被标记的样本,即不知道具体分类的样本 |
| 依据 | 数据自身的距离or相似度(常用欧氏距离) |
| 划分原则 | 组内距离最小,组间距离最大 |
| 输出 | 每个分类的聚类中心,及每个样本的所属分类,本次聚类的SSE |
| 聚类质量判定 | 误差平方和(SSE) |
| 优点 | 原理简单;可解释性强;便于处理大量数据 |
| 缺点 | k值难确定;聚类效果依赖于聚类中心的初始化,导致局部最优;仅能处理连续性数据;对噪音和异常点敏感 |
对距离的度量及SSE
对距离的度量常用的方法是欧氏距离,假设![]() 样本有p个属性,即
样本有p个属性,即![]() ,则样本
,则样本![]() 和样本
和样本![]() 之间的欧氏距离为:
之间的欧氏距离为:
误差平方和SSE是作为聚类质量的判定,公式为:
其中![]() 是
是![]() 个聚类中心中的第
个聚类中心中的第![]() 个聚类中心的聚类中心点。
个聚类中心的聚类中心点。
即目标是使得k个分类后,每一类中样本与该类的中心的距离尽可能的小,所有样本与其对应的分类中心的距离之和尽可能的小。
K-means是在最小化误差函数的基础上把数据划分为预先给定的k类,步骤为:
- 确定聚成k类(业务经验、肘方法等)
- 从n个样本中随机选取k个对象作为初始的聚类中心
- 分别计算每个样本到k个聚类中心的距离,将样本分配到距离最近的类中
- 对所有样本分配结束后,重新计算k个类的聚类中心
- 与前一次的聚类中心相比较,如果聚类中心发生变化,则转至3,否则转至6
- 当聚类中心不再发生变化时,停止输出并给出聚类结果
问题及如何避免
| 问题 | 解决方法 |
|---|---|
| k值难确定 | 业务经验、肘方法 |
| 聚类效果依赖于聚类中心的初始化,导致局部最优 | 实践中会多次选择不同的初始聚类中心,多次运行K-means算法 |
| 仅能处理连续性数据 | 离散型数据可以用K-中心点算法,思想类似 |
| 对噪音和异常点敏感 |
提前处理异常数据 |
k-means示例
数据样式:
即数据有三个属性,R、F、M
代码:
import pandas as pd
# 参数初始化
inputfile = r'F:\...\consumption_data.xls' # 销量及其他属性数据
outputfile = r'F:\...\data_type.xls' # 保存结果的文件名
k = 3 # 聚类的类别
iteration = 500 # 聚类最大循环次数
data = pd.read_excel(inputfile, index_col = 'Id') # 读取数据
data_zs = 1.0*(data - data.mean())/data.std() # 数据标准化
from sklearn.cluster import KMeans
model = KMeans(n_clusters = k, n_init = 4, max_iter = iteration,random_state=1234) # 分为k类,并发数4
model.fit(data_zs) # 开始聚类
# 简单打印结果
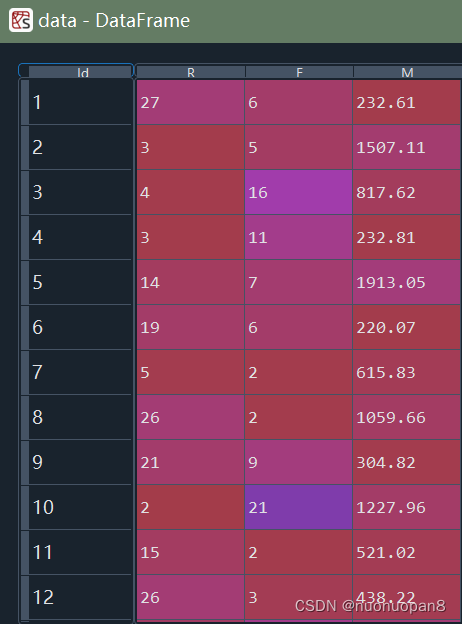
r1 = pd.Series(model.labels_).value_counts() # 统计各个类别的数目
r2 = pd.DataFrame(model.cluster_centers_) # 标准化数据的聚类中心
r2.columns=data.mean().index #修改r2的列,方便逆标准化计算
r_1 = pd.concat([r2*data.std()+data.mean(), r1], axis = 1) # 横向连接(0是纵向),得到聚类中心对应的类别下的数目
r_1.columns = list(data.columns) + ['类别数目'] # 重命名表头
print(r_1)
# 详细输出原始数据及其类别
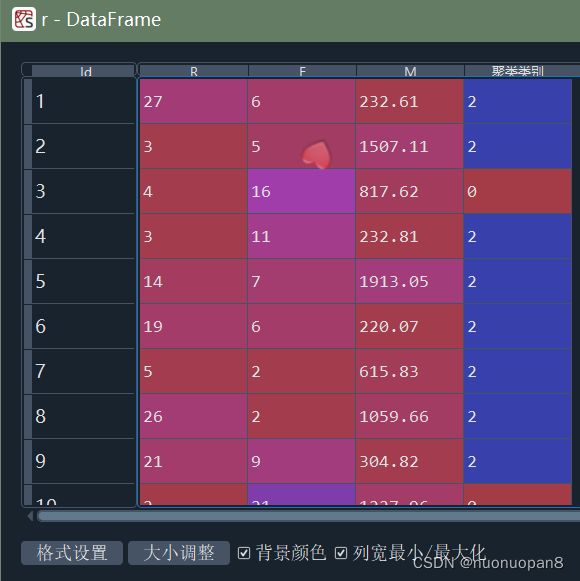
r = pd.concat([data, pd.Series(model.labels_, index = data.index)], axis = 1) # 详细输出每个样本对应的类别
r.columns = list(data.columns) + ['聚类类别'] # 重命名表头
# r.to_excel(outputfile) # 保存结果
#同时保存两个结果到xls中
with pd.ExcelWriter(outputfile) as writer: # doctest: +SKIP
r_1.to_excel(writer, sheet_name='聚类中心及数目')
r.to_excel(writer, sheet_name='原始数据聚类类别')
def density_plot(data): # 自定义作图函数
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
p = data.plot(kind='kde', linewidth = 2, subplots = True, sharex = False)
[p[i].set_ylabel('密度') for i in range(k)]
plt.legend()
plt.tight_layout()
return plt
pic_output = r'F:\...\分群'
# 概率密度图文件名前缀
for i in range(k):
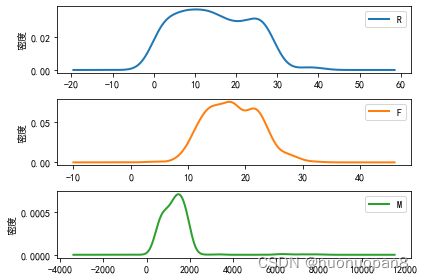
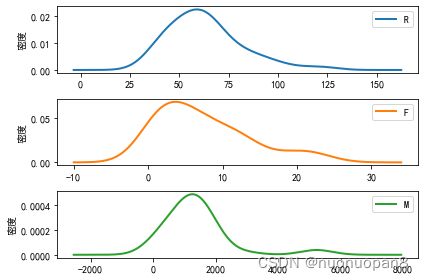
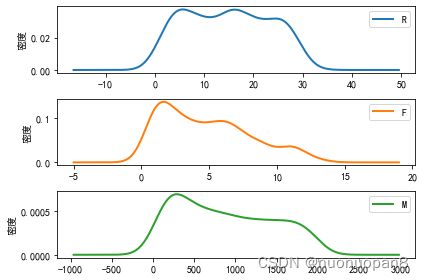
density_plot(data[r['聚类类别']==i]).savefig('%s%s.png' %(pic_output, i+1))结果:
①聚类中心和数目
② 原始数据的聚类结果
③每个分类的每个变量的概率密度函数图
k值选取
k值选取:
1.根据具体业务具体分析(经验)
2.肘方法--根据具体数据的性质
3. 轮廓图定量分析法
k值的选择一方面需要结合具体业务来确定,另一方面可以通过肘方法(下面会有例子)来估计,或者是利用轮廓图定量分析聚类质量(之后再补充)。
肘方法
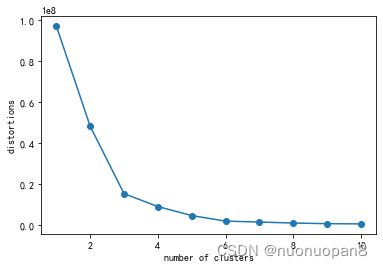
肘方法得到的最优参数k是以最小化误差平方和SSE为依据的。K-means使用误差平方和SSE来作为聚类质量的度量,对于两种不同的聚类结果,误差平方和SSE较小的结果更优,SSE是计算每个样本与其所属类别聚类中心的距离的平方,SSE会随着分类k的增加而减小,但随着分类数量k的增大,SSE的值趋于平缓,即分类数量再增加,SSE的也不会改变很大,也就是聚类的质量不会发生显著的提高了。根据这一情况,肘方法选取的k,是SSE发生骤变且骤变之后随着分类数量的增大而趋于平滑的k值。
肘方法的操作步骤:
1. 设定不同的k值,计算此k值下的聚类结果质量SSE
2. 画出k值-SSE图,确定k值
代码:
d=[]
for i in range(1,11): #k取值1~11,做kmeans聚类,看不同k值对应的簇内误差平方和
km=KMeans(n_clusters=i,init='k-means++',n_init=10,max_iter=300,random_state=0)
km.fit(data_zs)
d.append(km.inertia_) #inertia簇内误差平方和
plt.plot(range(1,11),d,marker='o')
plt.xlabel('number of clusters')
plt.ylabel('distortions')
plt.show()
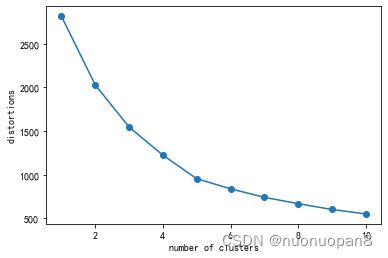
结果:
看这个结果,其实选择k=5比较好,k从1到2,从2到3,从3到4,从4到5,误差平方和SSE的减少都相当明显,从5到6,及之后,SSE的减少都有限,所以最佳的k值应该为5。由于上图看上去像一只手肘,理论最佳分类k值在手肘的肘处取得,这也是该方法叫肘方法的缘故。但如果根据具体问题具体分析得到分类k=3比较好,那也可以直接用3分类,肘方法是从数据层面去考虑的一个理论值,在实际操作中需要具体问题具体分析,肘方法可以用于验证3分类也可行,也比较好。
下面这个肘方法结果图是我做另一个数据分析时的结果,效果更佳好,可以直接看得出SSE的骤变:
可视化方法
将数据聚类结束后,利用可视化的方式展示分类结果效果更佳。
TSNE
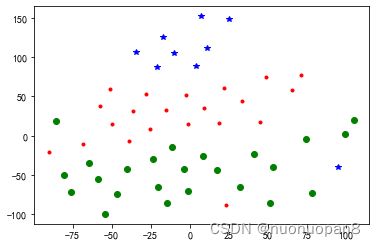
TSNE是一个聚类结果可视化的工具,其定位是高维数据的可视化。该工具会对数据进行降维,然后展示在二维或三维平面上。
代码:
import pandas as pd
from sklearn.manifold import TSNE
tsne = TSNE(random_state=105)
tsne.fit_transform(data_zs) # 用标准化数据进行数据降维
tsne = pd.DataFrame(tsne.embedding_, index = data_zs.index) # 转换数据格式,tsne.embedding_是降维后得到的数据,把他与原始数据的索引连接在一起(即与原始数据对应)
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号
# 不同类别用不同颜色和样式绘图
d = tsne[r['聚类类别'] == 0] #用原始数据的类别,对应取tsne降维后的结果
plt.plot(d[0], d[1], 'r.')
d = tsne[r['聚类类别'] == 1]
plt.plot(d[0], d[1], 'go')
d = tsne[r['聚类类别'] == 2]
plt.plot(d[0], d[1], 'b*')
plt.show()结果:
下面这个结果图是我做另一个数据分析时把20维的变量结果降到到2维平面的结果,说明之前我的分类结果具有较高的准确性,类别非常清晰:
雷达图
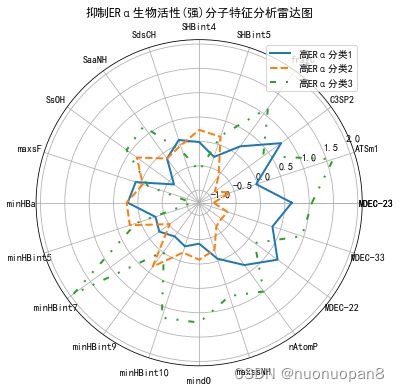
雷达图可以画出各个聚类中心在不同维度上的值,可以形象化的展示不同类的特点,下图是我比赛时,让给出每个变量的取值范围,但处理数据的时候,画变量的概率密度分布图发现某些变量有两个峰值,不能轻易的认为这些变量就只能有一个小范围的取值,我就对数据进行了一次聚类,利用聚类来区别同一类别不同取值的而形成的新的分类。
该数据用的是一个20维的数据,与前面使用的数据不同,这里仅展示雷达图显示分类结果的效果。
代码:
import pandas as pd
cluster_center=r_1.loc[:,col_1] #这里是取前面计算结果得到的二十维数据
import matplotlib.pyplot as plt
#客户分群雷达图
labels=list(cluster_center.columns)
legen=['高ERα分类'+str(i+1) for i in cluster_center.index] #客户群命名
lstype=['-','--',(0,(3,5,1,5,1,5)),':','-.']
kinds=list(cluster_center.iloc[:,0])
#由于雷达图要保证数据闭合,因此再添加L列,并转换为np.array
a=pd.concat([cluster_center,cluster_center.iloc[:,0]],axis=1)
centers=np.array(a)
#分割圆周长,并让其闭合
n=len(labels)
angle=np.linspace(0,2*np.pi,n,endpoint=False)
angle=np.concatenate((angle,[angle[0]]))
#绘图
fig=plt.figure(figsize=(8,6))
ax=fig.add_subplot(111,polar=True)
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
#画线
for i in range(len(kinds)):
ax.plot(angle,centers[i],linestyle=lstype[i],linewidth=2,label=kinds[i])
#添加属性标签
labels=labels+list([labels[0]])
ax.set_thetagrids(angle*180/np.pi,labels)
plt.title('抑制ERα生物活性(强)分子特征分析雷达图')
plt.legend(legen)
plt.show()
结果: