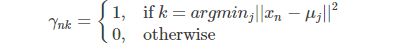kmeans聚类算法python实现、显示折线图_聚类算法kmeans,kmeans++及python实现
1、kmeans
kmeans, k-均值聚类算法,能够实现发现数据集的 k 个簇的算法,每个簇通过其质心来描述。
kmeans步骤:
(1)随机找 k 个点作为质心(种子);
(2)计算其他点到这 k 个种子的距离,选择最近的那个作为该点的类别;
(3)更新各类的质心,迭代到质心的不变为止。
Q:如何选择 k 值?
A: 根据 k 取不同的值时,模型性能曲线图。
横坐标是聚类数目k,纵坐标是各点到距离中心的距离和。在拐点处的K(距离和下降速率变慢)作为选择的值。
2、kmeans++
因为,kmeans的初始种子的随机找的,这样,算法的收敛快慢与初始值关系非常大,于是,kmeans++ 主要针对初始值的选取进行改进。
初始值选取,如下:
1、也是随机选取一个种子;
2、计算其他点到这个种子的距离;
3、选择这些较大的距离的那个点,替代随机选取的那个种子,作为新的种子点(这里的较大的距离,可以理解为maxDistance*随机数,随机数可以选取0.5-1;);
4、迭代1-3步,直到选取k个种子为止。
3. 评估聚类效果
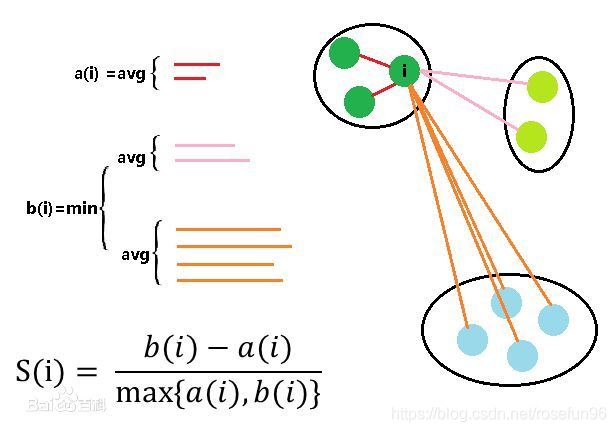
轮廓系数法SC
轮廓系数(Silhouette Coefficient))结合了聚类的凝聚度(Cohesion)和分离度(Separation),用于评估聚类的效果。该值处于-1~1之间,值越大,表示聚类效果越好。
计算步骤:
(1)选择一个点 ![]() ,计算该点 到同一簇内所有其他元素距离的平均值,记为
,计算该点 到同一簇内所有其他元素距离的平均值,记为 ![]() , 量化簇内的凝聚度;
, 量化簇内的凝聚度;
(2)选择一个其他簇,计算该点到簇内的平均距离。选择该点与其他簇平均距离最小的值,记为 ![]() , 用于量化簇间的分离度。
, 用于量化簇间的分离度。
(3)对于样本点![]() ,轮廓系数为:
,轮廓系数为:
a(i) :i向量到同一簇内其他点不相似程度的平均值
b(i) :i向量到其他簇的平均不相似程度的最小值
当轮廓系数<0, 该点可能误分; 值 = 0, 该点在两簇边缘; 值=1, 离其他簇很远。
计算所有点的轮廓系数,求平均值, 即为当前聚类的整体轮廓系数。
因此,轮廓系数可以用来确定Kmean的K值,选取轮廓系数最大的K作为最终的类数目。
(2)误差平方和
4. 其他问题
(1)kmeans一定会收敛吗
kmeans的目标函数:
E步:评估隐变量,每类样本属于的类别
M步:固定数据点的分配,更新参数
由于EM算法具有收敛性,kmeans也会最终收敛。
(2) Kmeans 和KNN的区别
KNN:
分类算法;
监督学习;
数据集是带Label的数据;
没有明显的训练过程,基于Memory-based learning;
K值含义 - 对于一个样本X,要给它分类,首先从数据集中,在X附近找离它最近的K个数据点,将它划分为归属于类别最多的一类
K-means:
聚类算法;
非监督学习;
数据集是无Label,杂乱无章的数据;
有明显的训练过程;
K值含义- K是事先设定的数字,将数据集分为K个簇,需要依靠人的先验知识
不同点:
两种算法之间的根本区别是,K-means本质上是无监督学习,而KNN是监督学习;K-means是聚类算法,KNN是分类(或回归)算法。
K-means算法把一个数据集分割成簇,使得形成的簇是同构的,每个簇里的点相互靠近。该算法试图维持这些簇之间有足够的可分离性。由于无监督的性质,这些簇没有任何标签。KNN算法尝试基于其k(可以是任何数目)个周围邻居来对未标记的观察进行分类。它也被称为懒惰学习法,因为它涉及最小的模型训练。因此,它不用训练数据对未看见的数据集进行泛化。
5. 实践
Kmeans python 代码:
import numpy as np
class KMeans(object):
def __init__(self, n_cluster, epochs=10):
self.n_cluster = n_cluster
self.epochs = epochs
pass
def init_centers(self, X):
idx = np.random.randint(len(X), size=(self.n_cluster,))
centers = X[idx,:]
return centers
def calculate_distance(self,arr1,arr2):
# L2 distance.
distance = np.mean(np.sqrt((arr1-arr2)**2))
return distance
def update_centers(self, X):
predict_class = self.predict(X)
# update centers
centers = self.centers
for ct in range(len(centers)):
idx, = np.where(predict_class == ct)
samples = X[idx, :]
assert len(samples)>0
centers[ct] = np.mean(samples,axis=0)
self.centers = centers
return self.centers
def fit(self, X, y=None):
self.centers = self.init_centers(X)
for epoch in range(self.epochs):
self.centers = self.update_centers(X)
return self.centers
def predict(self,X):
predict_class = np.zeros(shape=(len(X),))
centers = self.centers
for n_sample,arr in enumerate(X):
min_distance = float("inf")
p_class = 0
for ct in range(len(centers)):
distance = self.calculate_distance(arr,centers[ct])
if distance < min_distance:
min_distance = distance
p_class = ct
predict_class[n_sample] = p_class
return predict_class
def score(self, X):
pass复制代码
示例:
定义3个高斯分布。
np.random.seed(1)
data1 = np.random.normal(1, 0.5, size=(100, 2))
np.random.seed(1)
data2 = np.random.normal(4, 0.8, size=(100, 2))
np.random.seed(1)
data3 = np.random.normal(7, 0.8, size=(100, 2))
X = np.concatenate((data1, data2, data3),axis=0)#np.random.randint(10,size=(100,2))
print(X.shape)
kmeans = KMeans(3)
centers = kmeans.fit(X)
print(centers)复制代码
可视化聚类的结果:
# Generate scatter plot for training data
import matplotlib.pyplot as plt
plt.scatter(X[:,0], X[:,1], marker="o", picker=True)
plt.title('Clusters of data')
plt.xlabel('x1')
plt.ylabel('x2')
plt.scatter(centers[:,0], centers[:,1], marker="v", picker=True)
plt.show()复制代码
参考:
本文使用 mdnice 排版