基于SVM的乳腺癌数据集分类
目录
- 1.作者介绍
- 2.SVM算法介绍
-
- 2.1 SVM算法
- 2.2 SVM算法理解与分析
- 3.乳腺癌数据集介绍
- 4.基于SVM的乳腺癌数据集分类实验
-
- 4.1 导入所需要的包
- 4.2 导入乳腺癌数据集
- 4.3 输出数据集、特征等数据
- 4.4 可视化乳腺癌数据集
- 4.5 建模训练
- 4.6 输出训练分数以及测试分数
- 4.7 完整代码
- 5.结论
- 6.参考
1.作者介绍
车晨洁,女,西安工程大学电子信息学院,21级研究生
研究方向:机器视觉及人工智能
电子邮件:[email protected]
刘帅波,男,西安工程大学电子信息学院,2021级研究生,张宏伟人工智能课题组
研究方向:机器视觉与人工智能
电子邮件:[email protected]
2.SVM算法介绍
2.1 SVM算法
支持向量机(support vector machines,SVM)是一种二分类模型,SVM 的目的就是找一条线,以 “最好地” 区分这两类点,以至如果以后有了新的点,这条线也能做出很好的分类,这是在二维中的说明。在高维空间中,我们想要区分两类样本数据,需要找到一个超平面来区分两类样本数据。SVM 适合中小型数据样本、非线性、高维的分类问题。
“三八线”可以看作二维空间中SVM的形象解释,它传递出了以下几点重要的信息:
(1)是一条直线(线性函数);
(2)能将桌面分为两个部分,分别属于你和我(具有分类功能,是一种二值分类);
(3)位于课桌正中间,不偏向任何一方(注重公平原则,才能保证双方利益最大化)。
以上三点是SVM算法的中心思想。

2.2 SVM算法理解与分析
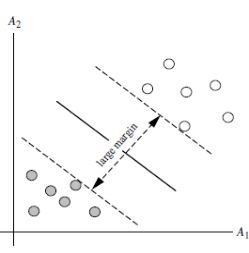
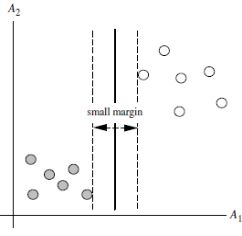
SVM 将会寻找可以区分两个类别并且能使间隔(margin)最大的划分超平面。比较好的划分超平面,样本局部扰动时对它的影响最小、产生的分类结果最鲁棒、对未见示例的泛化能力最强。由下图可知,H1是线性不可分的,H2、H3是线性可分的。这时,我们使用间隔最大的原理选择H3作为下图区分两类样本点的超平面。

从下图可以看出,虚线上的点到划分超平面的距离都是一样的,实际上只有这几个点共同确定了超平面的位置,因此被称作 “支持向量(support vectors)”,“支持向量机” 也是由此来的。
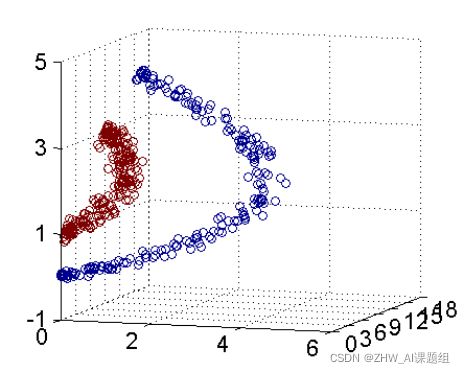
事实上,大部分时候数据并不是线性可分的,这个时候满足这样条件的超平面就根本不存在。对于非线性的情况,SVM 的处理方法是选择一个核函数 κ(⋅,⋅) ,通过将数据映射到高维空间,最终在高维特征空间中构造出最优分离超平面,从而把平面上本身不好分的非线性数据分开。如图所示,一堆数据在二维空间无法划分,从而映射到三维空间里划分:
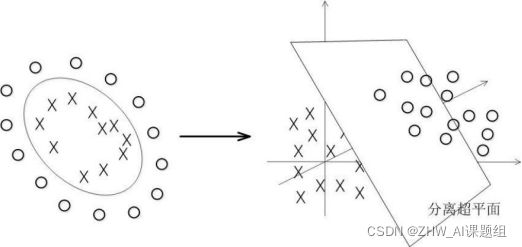
核函数的目的就是把数据进行分类,本课题用到了线性核、多项式核、高斯核(rbf)以及sigmoid核函数进行测试以及说明。
我们用一个动图来展示一下上述表述内容:
3.乳腺癌数据集介绍
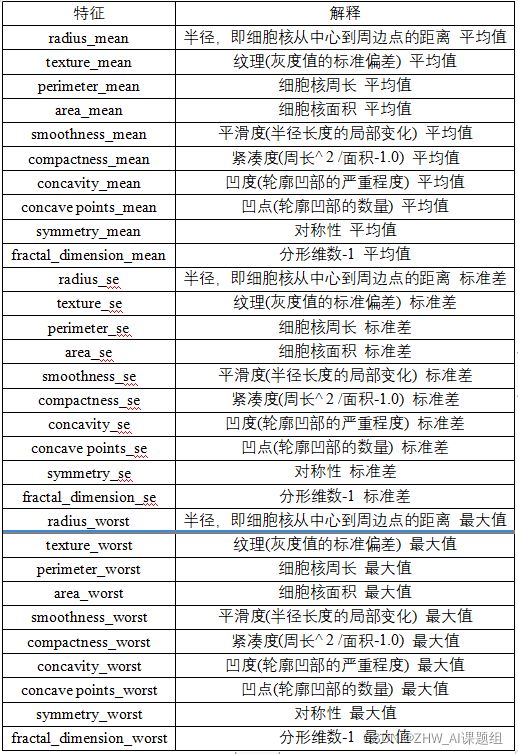
本课题使用的是Breast Cancer Wisconsin (Diagnostic) Data Set(威斯康星州乳腺癌(诊断)数据集),乳腺癌数据集一共有569个样本,30个特征(10个平均值,10个标准差,10个最值),标签为二分类。下图是乳腺癌数据集展示以及30个特征的具体描述。下面是二分类标签的具体类型和个数以及乳腺癌数据集的部分截图:


30个特征以及对应的解释具体如下:
4.基于SVM的乳腺癌数据集分类实验
4.1 导入所需要的包
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
import numpy as np
4.2 导入乳腺癌数据集
cancers = load_breast_cancer() #下载乳腺癌数据集
X = cancers.data #获取特征值
Y = cancers.target #获取标签
4.3 输出数据集、特征等数据
print("数据集,特征",X.shape) #查看特征形状
print(Y.shape) #查看标签形状
#print(X)#输出特征值
#print(Y)#输出特征值
#print(cancers.DESCR) #查看数据集描述
print('特征名称')#输出特征名称
print(cancers.feature_names) # 特征名
print('分类名称')#输出分类名称
print(cancers.target_names) # 标签类别名
# 注意返回值: 训练集train,x_train,y_train,测试集test,x_test,y_test
# x_train为训练集的特征值,y_train为训练集的目标值,x_test为测试集的特征值,y_test为测试集的目标值
# 注意,接收参数的顺序固定
# 训练集占80%,测试集占20%
x_train, x_test, y_train, y_test = train_test_split(X, Y, test_size=0.2)
print('训练集的特征值和目标值:', x_train, y_train)
#输出训练集的特征值和目标值
print('测试集的特征值和目标值:', x_test, y_test)
#输出测试集的特征值和目标值
#print(cancers.keys())
#可以根据自己写代码的习惯输出上述参数
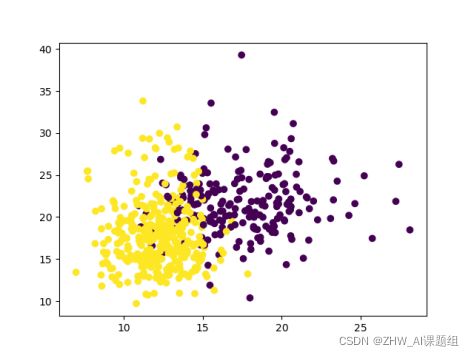
4.4 可视化乳腺癌数据集
np.unique(Y) # 查看label都由哪些分类
plt.scatter(X[:, 0], X[:, 1], c=Y)
plt.show() #显示图像
4.5 建模训练
#下面是四种核函数的建模训练
# 线性核
model_linear = SVC(C=1.0, kernel='linear')
# 多项式核
#degree表示使用的多项式的阶数
model_poly = SVC(C=1.0, kernel='poly', degree=3)
# 高斯核(RBF核)
#gamma是核函数的一个参数,gamma的值会影响测试精度
model_rbf = SVC(C=1.0, kernel='rbf', gamma=0.1)
# sigmoid核
gammalist=[] #把gammalist定义为一个数组
score_test=[] #把score_test定义为一个数组
gamma_dis=np.logspace(-100,-5,50)
#gamma_dis从10-100到10-5平均取50个点
for j in gamma_dis:
model_sigmoid = SVC(kernel='sigmoid', gamma=j,cache_size=5000).fit(x_train, y_train)
gammalist.append(j)
score_test.append(model_sigmoid.score(x_test, y_test))
#找出最优gammalist值
print("分数--------------------",score_test)
print("测试最大分数, gammalist",max(score_test),gamma_dis[score_test.index(max(score_test))])
plt.plot(gammalist,score_test) #横轴为gammalist纵轴为score_test
plt.show()#显示图片
输出结果如下:
![]()
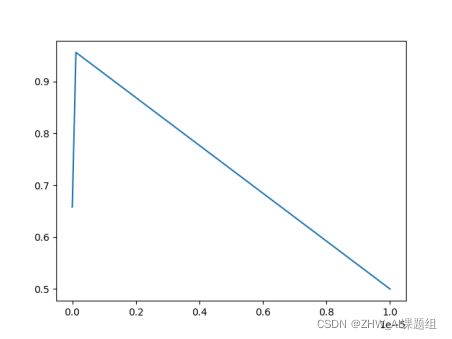
从输出的数据以及图片可知当gamma = 1.1513953993264481e-07,测试精度最高,为0.9298245614035088,测试精度最高时,我们把对应的gamma值叫做最优gamma值。
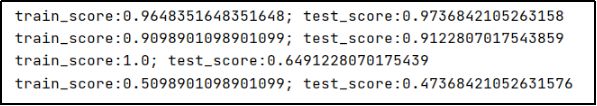
4.6 输出训练分数以及测试分数
model_linear.fit(x_train, y_train)
train_score = model_linear.score(x_train, y_train)
test_score = model_linear.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_poly.fit(x_train, y_train)
train_score = model_poly.score(x_train, y_train)
test_score = model_poly.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_rbf.fit(x_train, y_train)
train_score = model_rbf.score(x_train, y_train)
test_score = model_rbf.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_sigmoid.fit(x_train, y_train)
train_score = model_sigmoid.score(x_train, y_train)
test_score = model_sigmoid.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score,test_score))
4.7 完整代码
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
import numpy as np
cancers = load_breast_cancer() #下载乳腺癌数据集
X = cancers.data #获取特征值
Y = cancers.target #获取标签
print("数据集,特征",X.shape) #查看特征形状
print(Y.shape) #查看标签形状
#print(X)#输出特征值
#print(Y)#输出特征值
#print(cancers.DESCR) #查看数据集描述
print('特征名称')#输出特征名称
print(cancers.feature_names) # 特征名
print('分类名称')#输出分类名称
print(cancers.target_names) # 标签类别名
# 注意返回值: 训练集train,x_train,y_train,测试集test,x_test,y_test
# x_train为训练集的特征值,y_train为训练集的目标值,x_test为测试集的特征值,y_test为测试集的目标值
# 注意,接收参数的顺序固定
# 训练集占80%,测试集占20%
x_train, x_test, y_train, y_test = train_test_split(X, Y, test_size=0.2)
print('训练集的特征值和目标值:', x_train, y_train)
#输出训练集的特征值和目标值
print('测试集的特征值和目标值:',x_test, y_test)
#输出测试集的特征值和目标值
#print(cancers.keys())
#可以根据自己写代码的习惯输出上述参数
np.unique(Y) # 查看label都由哪些分类
plt.scatter(X[:, 0], X[:, 1], c=Y)
plt.show() #显示图像
#下面是四种核函数的建模训练
#下面是四种核函数的建模训练
# 线性核
model_linear = SVC(C=1.0, kernel='linear')
# 多项式核
#degree表示使用的多项式的阶数
model_poly = SVC(C=1.0, kernel='poly', degree=3)
# 高斯核(RBF核)
#gamma是核函数的一个参数,gamma的值会影响测试精度
model_rbf = SVC(C=1.0, kernel='rbf', gamma=0.1)
# sigmoid核
gammalist=[] #把gammalist定义为一个数组
score_test=[] #把score_test定义为一个数组
gamma_dis=np.logspace(-100,-5,50)
#gamma_dis从10-100到10-5平均取50个点
for j in gamma_dis:
model_sigmoid = SVC(kernel='sigmoid', gamma=j,cache_size=5000).fit(x_train, y_train)
gammalist.append(j)
score_test.append(model_sigmoid.score(x_test, y_test))
#找出最优gammalist值
print("分数--------------------",score_test)
print("测试最大分数, gammalist",max(score_test),gamma_dis[score_test.index(max(score_test))])
plt.plot(gammalist,score_test) #横轴为gammalist纵轴为score_test
plt.show()#显示图片
# 线性核
model_linear.fit(x_train, y_train)
train_score = model_linear.score(x_train, y_train)
test_score = model_linear.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_poly.fit(x_train, y_train)
train_score = model_poly.score(x_train, y_train)
test_score = model_poly.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_rbf.fit(x_train, y_train)
train_score = model_rbf.score(x_train, y_train)
test_score = model_rbf.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
model_sigmoid.fit(x_train, y_train)
train_score = model_sigmoid.score(x_train, y_train)
test_score = model_sigmoid.score(x_test, y_test)
print('train_score:{0}; test_score:{1}'.format(train_score, test_score))
#sigmoid函数输出训练精度和测试精度
5.结论
- 通过比较,线性核(linear)和多项式核(poly)测试精度较高,高斯核(rbf)和sigmoid核测试精度较低,因此本课题使用线性核以及多项式核测试得到的效果比较理想(大家后续也可以自己修改代码提升rbf核函数以及sigmoid核函数的精度);
- 高斯核的测试精度为1;
- 在sigmoid核函数中,gamma的值对测试精度有影响。且当
gamma=1.1513953993264481e-07,测试精度最高,为0.9298245614035088
6.参考
乳腺癌数据集链接:
https://pan.baidu.com/s/1DN4AlRzDkmBSZlnk8dY15g 提取码:i6u6
博客参考链接:
https://blog.csdn.net/qq_42363032/article/details/107210881