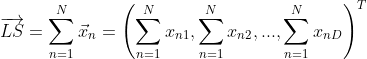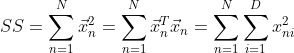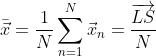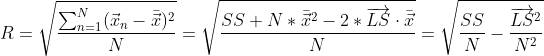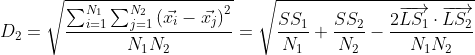机器学习笔记(九)聚类算法Birch和层次聚类Hierarchical clustering
本篇文章我们继续介绍另一种聚类算法——Birch模型,相对于K-means和DBSCAN,Birch的应用并没有那么广泛,不过它也有一些独特的优势,Birch算法比较适合于数据量大,类别数K也比较多的情况,它运行速度很快,只需要单遍扫描数据集就能进行聚类,这在数据量日益庞大的今天是一个比较大的优势。
一、原理
Birch(Balanced Iterative Reducing and Clustering using Hierarchies)是层次聚类的典型代表,天生就是为处理超大规模数据集而设计的,它利用一个树结构来快速聚类,这个树结构类似于平衡B+树,一般将它称之为聚类特征树(Clustering Feature Tree,简称CF Tree)。这颗树的每一个节点是由若干个聚类特征(Clustering Feature,简称CF)组成。
1.1 聚类特征
设某簇中有N个D维的数据点,,则该簇的聚类特征CF定义为三元组:![]() ,其中N为该簇中数据点的数量,
,其中N为该簇中数据点的数量,![]() 代表了这个CF中拥有的样本点各特征维度的和向量(下文的LS都是表示向量),SS代表了这个CF中拥有的样本点各特征维度的平方和。
代表了这个CF中拥有的样本点各特征维度的和向量(下文的LS都是表示向量),SS代表了这个CF中拥有的样本点各特征维度的平方和。
假设在某个簇中,有下面5个样本{(3,4), (2,6), (4,5), (4,7), (3,8)}。则它对应的:![]() ,
,![]() 。则由上述定义可知对于两个簇的聚类特征有下面的线性可加性:
。则由上述定义可知对于两个簇的聚类特征有下面的线性可加性:
![]()
1.2 聚类特征树
和前面我们介绍 KNN 利用 KD 树和球树来加速最近邻寻找效率类似,Birch 原理同样也构建了一个树结构——聚类特征树。这个数结构类似于平衡B+树,一般将它称之为聚类特征树(Clustering Feature Tree,简称CF Tree)。这颗树的每一个节点是由若干个聚类特征(Clustering Feature,简称CF)组成。从下图(注意图中的n只是一个变量,并不是每个节点的CF数要一样,每个节点的n都可以是不相等的)我们可以看看聚类特征树是什么样子的:每个节点包括叶子节点都有若干个CF,叶子节点的CF是最小单位簇,而内部节点的CF有指向子节点的指针,所有的叶子节点用一个双向链表链接起来,可以快速的检索所有的叶子节点。从图中可以看出,根节点的CF的三元组的值,可以从它指向的子节点的值相加得到。这样我们在更新CF Tree的时候,可以很高效。
还需要说明的是,CF树上不保存任何数据点,仅有树的结构信息以及相关的聚类特征信息,因此CF树可以达到压缩数据的效果。对于CF Tree,我们一般有几个重要参数,第一个参数是每个内部节点的最大CF数B,第二个参数是每个叶子节点的最大CF数L,第三个参数是针对叶子节点中某个CF中的样本点来说的,它是叶节点每个CF的最大样本半径阈值T,也就是说,在这个CF中的所有样本点一定要在半径小于T的一个超球体内。
理解了CF Tree,我们再定义几个关于CF Tree的概念变量,在构造CF Tree的过程中,也多是依赖这些变量构建的。设某簇中有N个D维的数据点,,
则该簇的形心或者质心为:
簇半径为(相当于全局标准差):
注意簇半径并不是所有点到簇中心距离的均值。
簇直径为:
分子是簇中任意两点的距离平方。
任意两个簇之间的距离(默认不相交):
这里的距离称为簇连通平均距离,也可以使用形心欧几里得距离、形心曼哈顿距离或者全连通平均距离作为衡量标准,本文我们都按照簇连通平均距离为准。
可以发现簇中心、簇半径、簇直径都可以由CF来计算,所以,基于CF Tree的Birch聚类算法计算会非常快,每次更新的时候,不需要计算和所有样本点的距离。
1.3 构建CF Tree
前面我们介绍了CF Tree的结构,Birch聚类原理主要就是利用CF Tree,从而只需要扫描一次,就可以完成聚类过程。本节我们以案例的形式详细介绍Birch构建CF Tree的过程。假设已有数据集为:{[0, 1], [0.3, 1], [-0.3, 1], [0, -1], [0.3, -1], [-0.3, -1], [-0.3,-1.1],[1,2]},构建 CF Tree 每个内部节点的最大CF数B和每个叶子节点的最大CF数L都取2,叶节点每个CF的最大样本半径阈值T为0.2。CF Tree的构建过程如下:
(1)初始化CF树为空树,从训练集读入第一个样本点 [0, 1],将它放入一个新的CF三元组 A,显然这个聚类特征三元组为 (1,(0,1),1),将这个新的CF放入根节点。
(2)读入第二个样本点 [0.3, 1],这个样本点和A组成的新聚类特征三元组为 (2,(0.3,2),2.09),由1.2节的簇半径计算公式可得 ![]() ,即在半径为T=0.2的超球体范围内,也就是说,他们属于一个CF,我们将第二个点也加入A,并和第一个样本点组成簇1,更新A的三元组值为 (2,(0.3,2),2.09)。
,即在半径为T=0.2的超球体范围内,也就是说,他们属于一个CF,我们将第二个点也加入A,并和第一个样本点组成簇1,更新A的三元组值为 (2,(0.3,2),2.09)。
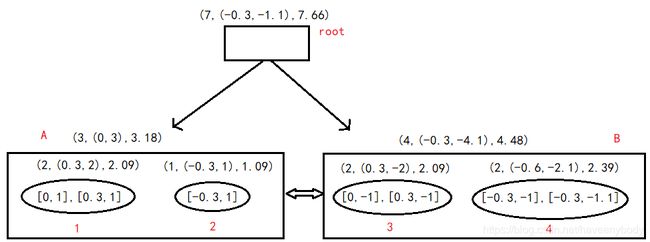
(3)读入第三个样本点 [-0.3, 1],这个样本点和A组成的新聚类特征三元组为 (3,(0,3),3.18),则新的 R=0.24,即不在半径为T=0.2的超球体范围内,不能分到同一个CF,不过现在A中只有一个簇,每个叶子节点的最大CF数L为2,因此为第三个点在A中分配一个新的簇2,簇2的聚类特征为 (1,(-0.3,1),1.09),A的聚类特征更新为 (3,(0,3),3.18),此时的CF Tree如图2。
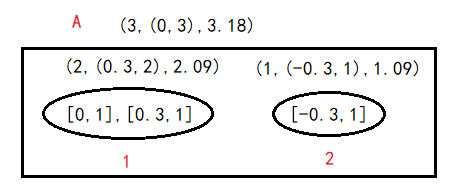
(4)读入第四个样本点 [0, -1],此时的叶节点只有一个,根据1.2节簇间距离公式,分别计算新样本点到簇1和簇2的距离为:![]() ,
,![]() ,即离A更近,新样本点和A组成的新聚类特征三元组为 (3,(0.3,1),3.09),则新的 R=0.95,即不在半径为T=0.2的超球体范围内,不能分到同一个CF,因此第四个样本点分配到簇3,特征三元组为 (1,(0,-1),1),不过现在A中已经有两个簇,不能再分配新的簇。计算簇1、2之间的距离为:
,即离A更近,新样本点和A组成的新聚类特征三元组为 (3,(0.3,1),3.09),则新的 R=0.95,即不在半径为T=0.2的超球体范围内,不能分到同一个CF,因此第四个样本点分配到簇3,特征三元组为 (1,(0,-1),1),不过现在A中已经有两个簇,不能再分配新的簇。计算簇1、2之间的距离为:![]() ,簇间距离最大的是2和3,把2和3分配到两个叶节点中,簇1距离2更近,因此把1分配到和2同一个叶节点中,则新的CF Tree如下图3。
,簇间距离最大的是2和3,把2和3分配到两个叶节点中,簇1距离2更近,因此把1分配到和2同一个叶节点中,则新的CF Tree如下图3。
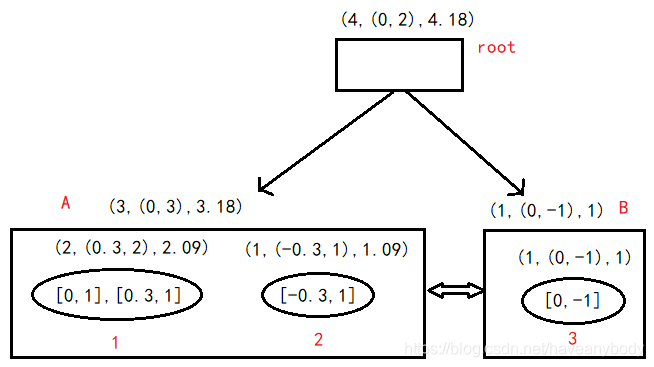
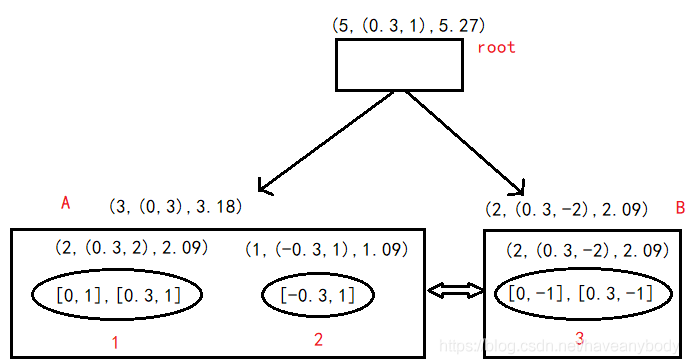
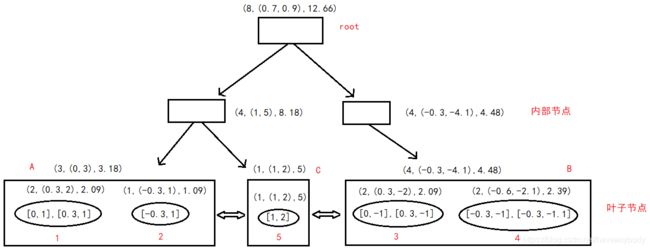
(5)读入第五个样本点 [0.3, -1],分别计算新样本点到叶节点A和B的距离,可以发现距离B更近,新样本点和簇3组成的新特征三元组为 (2,(0.3,-2),2.09),计算新的 R=0.15,即在半径为T=0.2的超球体范围内,也就是说,他们属于一个CF,更新簇3、叶节点B和根节点的特征三元组如图4。
(6)读入第六个样本点 [-0.3, -1],分别计算新样本点到叶节点A和B的距离,可以发现距离B更近,继续计算发现,新样本点和簇3组成的新簇半径大于0.2,因此在B中为新样本点分配新的簇4,并更新B和root的特征三元组。继续读入第七个样本点,计算发现最终也是划分到簇4中,则此时的CF Tree如图5。
(7)读入最后一个样本点[1,2],分别计算新样本点到叶节点A和B的距离为:1.435、3.22,即距离A更近,且距离簇1更近,新样本点和簇1合并后的新特征三元组为 (3,(1.3,4),7.09),新簇半径 R=0.63,大于0.2,不能划分到簇1中,为新样本点分配簇5,因为A中已经有两个簇,不能再分配新的簇,簇1、2、5之间距离最大的是2和5,把2和5重新分配到两个叶节点中,因为1距离2更近,因此1分配到和2同一个叶节点中,则最终的CF Tree如图6。
至此,我们就完成了整个CF Tree的构建,树高度为3,最终的聚类结果划分为5个簇,如果有新的样本点,继续按照上述流程即可。假如新的样本点距离左子树更近,其次距离A更近,如果又不能划分到簇1和簇2中,则需要分配新的簇6,但是因为A中已经有了两个簇,因此A需要分裂,按照簇1、2、6之间距离最大的两个簇(假设1、6最大,2距离1更近)分配到两个叶节点,则A的父节点就有了三个叶子,分别是 (1,2)、(5)、(6)。因为内部节点的最大CF数B定义也是2,所以需要在 (1,2)、(5)、(6) 三个特征三元组中找出距离最大的分配到两个叶节点中,并把剩下的划分到距离自己最近的即可,具体内容不再赘述。另外需要注意的是,这里为了方便理解,把每个样本到画在了图中,在实际中 CF Tree 是不保存样本的,只保存对应样本的索引或者说指向样本的指针,因此CF Tree不需要耗费太多的内存资源。
下面我们总结一下CF Tree的一般构建流程:
- 从根节点向下寻找和新样本距离最近的叶子节点和叶子节点里最近的CF节点
- 如果新样本加入后,这个CF节点对应的超球体半径仍然满足小于阈值T,则更新路径上所有的CF三元组,插入结束。否则转入3
- 如果当前叶子节点的CF节点个数小于阈值L,则创建一个新的CF节点,放入新样本,将新的CF节点放入这个叶子节点,更新路径上所有的CF三元组,插入结束。否则转入4
- 将当前叶子节点划分为两个新叶子节点,选择旧叶子节点中所有CF元组里超球体距离最远的两个CF元组,分别作为两个新叶子节点的第一个CF节点。将其他元组和新样本元组按照距离远近原则放入对应的叶子节点。依次向上检查父节点是否也要分裂,如果需要按照和叶子节点相同的方法进行分裂。
1.4 Birch原理
前面我们详细介绍了CF Tree的构建过程,其实将所有的训练集样本建立了CF Tree,一个基本的Birch算法就完成了,对应的输出就是若干个CF节点,每个节点里的样本点就是一个聚类的簇。也就是说BIRCH算法的主要过程,就是建立CF Tree的过程。现在我们就来看看 BIRCH算法的流程[1]:
- 将所有的样本依次读入,在内存中建立一颗CF Tree
- (可选)将第一步建立的CF Tree进行筛选,去除一些异常CF节点,这些节点一般里面的样本点很少。对于一些超球体距离非常近的元组进行合并
- (可选)利用其它的一些聚类算法比如K-Means对所有的CF元组进行聚类,得到一颗比较好的CF Tree。这一步的主要目的是消除由于样本读入顺序导致的不合理的树结构,以及一些由于节点CF个数限制导致的树结构分裂。可以认为是一个半监督K-Means过程。此外,Birch还可以作为其他聚类算法的输入,例如 AgglomerativeClustering。
- (可选)利用第三步生成的CF Tree的所有CF节点的质心,作为初始质心点,对所有的样本点按距离远近进行聚类。这样进一步减少了由于CF Tree的一些限制导致的聚类不合理的情况。
从上面可以看出,Birch算法的关键就是步骤1,也就是CF Tree的生成,其他步骤都是为了优化最后的聚类结果。Birch算法可以不用输入类别数K值,这点和K-Means,Mini Batch K-Means不同,其实更像KNN。如果不输入K值,则最后的CF元组的组数即为最终的K,否则会按照输入的K值对CF元组按距离大小进行合并。
一般来说,Birch算法适用于样本量较大的情况,这点和Mini Batch K-Means类似,但是Birch适用于类别数比较大的情况,而Mini Batch K-Means一般用于类别数适中或者较少的时候。Birch除了聚类还可以额外做一些异常点检测和数据初步按类别规约的预处理。但是如果数据特征的维度非常大,则Birch不太适合,根据经验,如果n_features大于20,使用MiniBatchKMeans通常更好。
下面我们再总结一下Birch的优缺点:
(1)优点
- 节约内存,所有的样本都在磁盘上,CF Tree仅仅存了CF节点和对应的指针(指向父节点和孩子节点的指针)。
- 聚类速度快,只需要一遍扫描训练集就可以建立CF Tree,CF Tree的增删改都很快,由于B树是高度平衡的,所以在树上进行插入或查找操作很快,而且合并两个簇只需要两个CF算术相加即可,计算两个簇的距离只需要用到聚类特征。
- 可识别噪声点,即那些包含数据点少的MinCluster,还可以对数据集进行初步分类的预处理。
(2)缺点
- 对非球状的簇聚类效果不好,取决于簇直径和簇间距离的计算方法。
- 对高维数据聚类效果不好,此时的计算量主要在于SS和簇间距离的计算,此时可以选择Mini Batch K-Means。
- 结果依赖于数据点的插入顺序。本属于同一个簇的点可能由于插入顺序相差很远而分到不同的簇中。
- 由于每个节点只能包含一定数目的子节点,最后得出来的簇可能和实际簇相差很大。
- Birch适合于处理大数据量和大类别的数据集,但在整个过程中算法一旦中断,一切必须从头再来。
- 局部性也导致了Birch的聚类效果欠佳。当一个新点要插入B树时,它只跟很少一部分簇进行了相似性(通过计算簇间距离)比较,高的efficient导致低的effective。
二、实践
2.1 API
Birch的sklearn API定义如下:
sklearn.cluster.Birch(*,
threshold=0.5, ##最大样本半径阈值
branching_factor=50, ##内部节点和叶子节点的最大CF数,sklearn对两个值取了相同值
n_clusters=3, ##聚类数量,默认是3
compute_labels=True, ##训练的时候是否输出簇标签
copy=True)这里,再解释一下n_clusters参数,如果取None,则输出CF Tree的簇结果,也即最细粒度的聚类结果;如果是整型值,则对CF Tree的聚类结果,通过AgglomerativeClustering按照簇之间距离最近原则,合并最近的簇,直到达到指定的簇数;该参数还可以设置为其他聚类算法,并以CF Tree的聚类结果,对原始的细粒度簇结果做聚类,参考1.4节Birch流程的第3步。另外,Birch还提供了transform方法,返回样本点到所有簇中心的距离。
2.2 案例
(1)测试前面的demo
from sklearn.cluster import Birch
X = [[0, 1], [0.3, 1], [-0.3, 1], [0, -1], [0.3, -1], [-0.3, -1], [-0.3,-1.1], [1,2]]
brc = Birch(n_clusters=None,threshold=0.25,branching_factor=2)
brc.fit(X)
brc.predict(X)
array([0, 0, 0, 2, 2, 3, 3, 1])(2)比较Birch和MiniBatchKMeans
from itertools import cycle
from time import time
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as colors
from sklearn.cluster import Birch, MiniBatchKMeans
from sklearn.datasets import make_blobs
from sklearn import metrics
xx = np.linspace(-22, 22, 10)
yy = np.linspace(-22, 22, 10)
xx, yy = np.meshgrid(xx, yy)
n_centres = np.hstack((np.ravel(xx)[:, np.newaxis],
np.ravel(yy)[:, np.newaxis]))
# Generate blobs to do a comparison between MiniBatchKMeans and Birch.
X, y = make_blobs(n_samples=100000, centers=n_centres, random_state=0)
# Use all colors that matplotlib provides by default.
colors_ = cycle(colors.cnames.keys())
fig = plt.figure(figsize=(12, 4))
fig.subplots_adjust(left=0.04, right=0.98, bottom=0.1, top=0.9)
# Compute clustering with Birch with and without the final clustering step
# and plot.
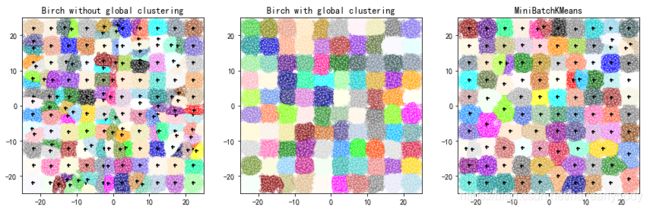
birch_models = [Birch(threshold=1.7, n_clusters=None),
Birch(threshold=1.7, n_clusters=100)]
final_step = ['without global clustering', 'with global clustering']
for ind, (birch_model, info) in enumerate(zip(birch_models, final_step)):
birch_model.fit(X)
# Plot result
labels = birch_model.labels_
centroids = birch_model.subcluster_centers_
n_clusters = np.unique(labels).size
print("n_clusters : %d" % n_clusters)
ax = fig.add_subplot(1, 3, ind + 1)
for this_centroid, k, col in zip(centroids, range(n_clusters), colors_):
mask = labels == k
ax.scatter(X[mask, 0], X[mask, 1],
c='w', edgecolor=col, marker='.', alpha=0.5)
if birch_model.n_clusters is None:
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_ylim([-25, 25])
ax.set_xlim([-25, 25])
ax.set_autoscaley_on(False)
ax.set_title('Birch %s' % info)
labels = birch_model.predict(X)
print(metrics.silhouette_score(X, labels, metric='euclidean'))
print(metrics.calinski_harabasz_score(X, labels))
mbk = MiniBatchKMeans(init='k-means++', n_clusters=100, batch_size=100,
n_init=10, max_no_improvement=10, verbose=0,
random_state=0)
mbk.fit(X)
mbk_means_labels_unique = np.unique(mbk.labels_)
ax = fig.add_subplot(1, 3, 3)
for this_centroid, k, col in zip(mbk.cluster_centers_,
range(n_clusters), colors_):
mask = mbk.labels_ == k
ax.scatter(X[mask, 0], X[mask, 1], marker='.',
c='w', edgecolor=col, alpha=0.5)
ax.scatter(this_centroid[0], this_centroid[1], marker='+',
c='k', s=25)
ax.set_xlim([-25, 25])
ax.set_ylim([-25, 25])
ax.set_title("MiniBatchKMeans")
ax.set_autoscaley_on(False)
plt.show()
labels = mbk.predict(X)
print(metrics.silhouette_score(X, labels, metric='euclidean'))
print(metrics.calinski_harabasz_score(X, labels))Birch的两个重要参数 threshold 和 branching_factor 并不容易调节,在应用中,也可通过网格搜索的方法联合调参。
三、层次聚类
在前面介绍Birch原理中,有一步是对CF Tree的结果进行再次聚类优化,提到了可以使用AgglomerativeClustering根据簇距离大小进行二次聚类,AgglomerativeClustering就是层次聚类的典型代表。
3.1 原理
层次聚类(Hierarchical clustering),顾名思义,就是一层一层的进行聚类,可以由上向下把大的簇依次分割为小簇,叫作分裂法;也可以由下向上对小的类别按照距离最近原则进行合并,直到达到指定的簇数或者簇间距离大于指定的阈值,叫作凝聚法,一般用的较多的是由下向上的凝聚法,AgglomerativeClustering就是凝聚法的代表。本文会对分裂法略作简介和举个例子说明,重点介绍凝聚法,并且附上代码。
(1)分裂法
原始数据集是一个簇,然后分裂为两个簇,分裂的原则是不在同一个簇的样本距离尽可能大,在同一个簇的样本之间尽量紧密。例如,第一步可以选择原始簇中距离最远的两个点作为原始质心,然后把剩下的点分到距离自己最近的质心簇中,然后调整质心,重新划分簇直到稳定,第二步选择已有簇中较为松散的簇继续执行第一步的过程,循环直到达到指定的簇数即可。在选择要分隔的簇时,可以选择簇半径较大的簇、簇中间隔最远的样本点之间距离最大的簇或者簇中样本到簇质心距离(SSE)最大的簇等。其实,分裂法一定程度上可以看作是不断进行两聚类的 k-means++ 。
(2)凝聚法
凝聚法从最小单位簇开始,不断合并距离最小的簇,直到达到指定的簇数,数学过程表达如下:
- 原始数据集每个样本划分为一个簇,即
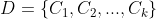
- 依次计算D中任意两个簇的距离,假如最近的两个簇是
 和
和 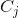 ,则合并
,则合并  和
和 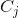 为新的簇
为新的簇 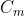 ,从 D 中删除
,从 D 中删除  和
和 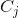 ,并将
,并将 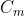 添加到 D 中
添加到 D 中 - 重复2,直到达到指定的簇数或者距离最近的簇间距离大于指定的阈值
凝聚法的关键问题是如何判断两个距离最小的簇,主要方法有:
- Maximum/complete linkage:两个簇中距离最大的样本点之间的距离最小的两个簇
- Single linkage:两个簇中距离最近的样本点之间的距离最小的两个簇
- Average linkage:两个簇中,任意两个样本对之间的平均距离最小的两个簇
- Ward:合并后所有簇内差异的平方和最小的两个簇,即簇内样本点到簇质心的距离平方和,和 k-means 的目标函数一样(参考聚类算法K-means原理和实践)
3.2 实践
sklearn官网只提供了凝聚法的API AgglomerativeClustering,对于分裂法自己实现也很容易·,只要自定义一个选择要分裂簇的指标,对其进行 k-means 二聚类就可以了。
(1)API
sklearn.cluster.AgglomerativeClustering(
n_clusters=2, ##簇的数量,如果指定了distance_threshold参数,则该参数需为None
*,
affinity='euclidean', ##计算linkage参数的度量,默认是欧氏距离,可选“euclidean”, “l1”, “l2”, “manhattan”, “cosine”, or “precomputed”,如果是“precomputed”,在fit时,需要传入一个距离矩阵
memory=None, ##计算树的缓存输出,默认不输出,如果指定一个路径,则输出
connectivity=None, ##连接矩阵。某些情况下,我们希望只有相邻(挨着)的样本才能聚类到一起,此时就可以传入一个约束连接矩阵。这个矩阵可以由先验信息构建,它也可以从数据中学习,例如使用sklearn.neighbors.kneighbors_graph限制合并到最近的邻居,或者使用sklearn.feature_extraction.image。grid_to_graph允许只合并图像上的相邻像素
compute_full_tree='auto',
linkage='ward', ##最小距离簇判定方法,默认 'ward',可选‘ward’, ‘complete’, ‘average’, ‘single’
distance_threshold=None, ##超过该链接距离阈值的簇将不会被合并。如果不是None, n_clusters必须为None, compute_full_tree必须为True
compute_distances=False)(2)案例
使用scipy可视化AgglomerativeClustering。
import numpy as np
from matplotlib import pyplot as plt
from scipy.cluster.hierarchy import dendrogram
from sklearn.datasets import load_iris
from sklearn.cluster import AgglomerativeClustering
def plot_dendrogram(model, **kwargs):
# Create linkage matrix and then plot the dendrogram
# create the counts of samples under each node
counts = np.zeros(model.children_.shape[0])
n_samples = len(model.labels_)
for i, merge in enumerate(model.children_):
current_count = 0
for child_idx in merge:
if child_idx < n_samples:
current_count += 1 # leaf node
else:
current_count += counts[child_idx - n_samples]
counts[i] = current_count
linkage_matrix = np.column_stack([model.children_, model.distances_,
counts]).astype(float)
# Plot the corresponding dendrogram
dendrogram(linkage_matrix, **kwargs)
iris = load_iris()
X = iris.data
# setting distance_threshold=0 ensures we compute the full tree.
model = AgglomerativeClustering(distance_threshold=0, n_clusters=None)
model = model.fit(X)
plt.title('Hierarchical Clustering Dendrogram')
# plot the top three levels of the dendrogram
plot_dendrogram(model, truncate_mode='level', p=3)
plt.xlabel("Number of points in node (or index of point if no parenthesis).")
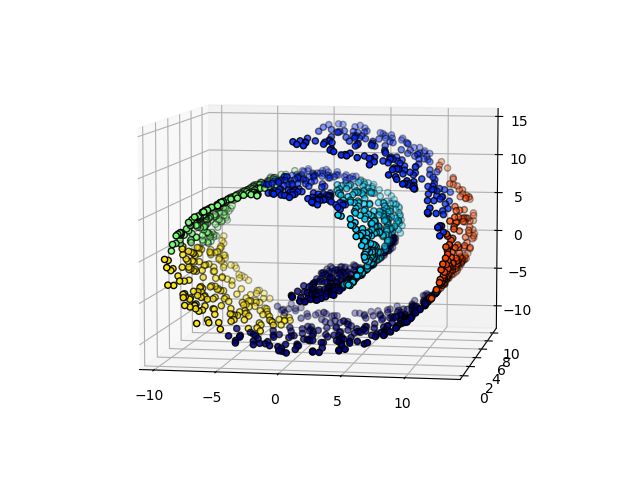
plt.show()添加/不添加连接矩阵在“瑞士卷”数据集上的表现,可以发现,指定连接矩阵的结果更合理。
import time as time
import numpy as np
import matplotlib.pyplot as plt
import mpl_toolkits.mplot3d.axes3d as p3
from sklearn.cluster import AgglomerativeClustering
from sklearn.datasets import make_swiss_roll
# #############################################################################
# Generate data (swiss roll dataset)
n_samples = 1500
noise = 0.05
X, _ = make_swiss_roll(n_samples, noise=noise)
# Make it thinner
X[:, 1] *= .5
# #############################################################################
# Compute clustering
print("Compute unstructured hierarchical clustering...")
st = time.time()
ward = AgglomerativeClustering(n_clusters=6, linkage='ward').fit(X)
elapsed_time = time.time() - st
label = ward.labels_
print("Elapsed time: %.2fs" % elapsed_time)
print("Number of points: %i" % label.size)
# #############################################################################
# Plot result
fig = plt.figure()
ax = p3.Axes3D(fig)
ax.view_init(7, -80)
for l in np.unique(label):
ax.scatter(X[label == l, 0], X[label == l, 1], X[label == l, 2],
color=plt.cm.jet(float(l) / np.max(label + 1)),
s=20, edgecolor='k')
plt.title('Without connectivity constraints (time %.2fs)' % elapsed_time)
# #############################################################################
# Define the structure A of the data. Here a 10 nearest neighbors
from sklearn.neighbors import kneighbors_graph
connectivity = kneighbors_graph(X, n_neighbors=10, include_self=False)
# #############################################################################
# Compute clustering
print("Compute structured hierarchical clustering...")
st = time.time()
ward = AgglomerativeClustering(n_clusters=6, connectivity=connectivity,
linkage='ward').fit(X)
elapsed_time = time.time() - st
label = ward.labels_
print("Elapsed time: %.2fs" % elapsed_time)
print("Number of points: %i" % label.size)
# #############################################################################
# Plot result
fig = plt.figure()
ax = p3.Axes3D(fig)
ax.view_init(7, -80)
for l in np.unique(label):
ax.scatter(X[label == l, 0], X[label == l, 1], X[label == l, 2],
color=plt.cm.jet(float(l) / np.max(label + 1)),
s=20, edgecolor='k')
plt.title('With connectivity constraints (time %.2fs)' % elapsed_time)
plt.show()
![]()
聚类+分类器进行归纳学习。
参考文献
[1] https://www.cnblogs.com/pinard/p/6179132.html
[2] https://www.cnblogs.com/zhangchaoyang/articles/2200800.html
[3] https://www.cnblogs.com/tiaozistudy/p/6129425.html