基于Python计算Landsat8OLI遥感生态指数RSEI
1 简介
遥感生态指数RSEI, 是一种使用卫星遥感影像数据通过反演计算得到的数据,可以用来对城市的生态状况进行快速监测和评价,该指数主要利用主成分分析方法,对植被指数、湿度、地表温度以及建筑指数四项指数指标进行集成。详细信息还请参考阅读:徐涵秋.城市遥感生态指数的创建及其应用[J].生态学报,2013,33(24):7853-7862.
前段时间帮朋友用Python写了个基于Landsat8 OLI计算RSEI的脚本,最近想起来了,所以简单做个记录,有需要的可以参考下,其中计算的主要步骤如下
1、读取数据各个波段
2、计算NDVI
3、计算WET
4、计算LST
5、计算NDBSI
6、PCA主成分分析
7、结果数据导出
1.1 数据介绍
本次所采用的Landsat8 OLI数据来源于GEE已经进行过预处理的数据,挑选了其中的
B1 B2 B3 B4 B5 B6 B7 B10 B11波段并导出为tif格式。
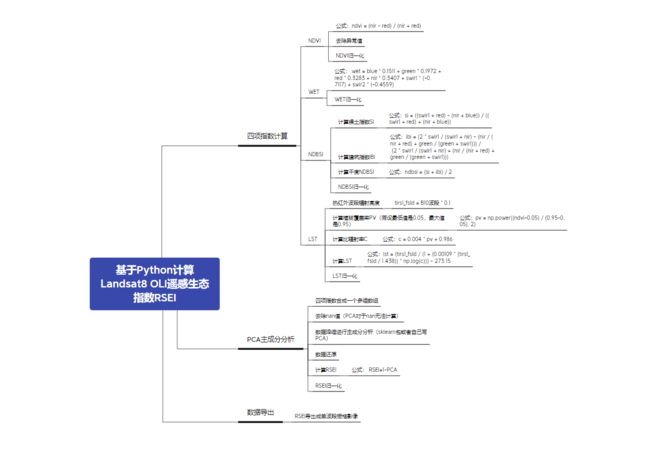
1.2 思维导图
从思维导图中可以看出,我们首先需要计算出四项指数,然后再用PCA主成分分析对四项指数进行计算得到RSEI,最后将结果导出即可,思维导图获取方式在文末。
2 分析过程和部分代码
栅格数据的处理主要使用的是rasterio包,矢量数据的处理是用的geopandas,环境的安装可以参考我以前写的文章教程。
2.1 改变数据类型
在数据处理的过程中,我发现我使用的数据类型是int16,但是在后续的计算中因为涉及到计算和小数的出现,我将数据类型改变为了float64,代码
def changeDtype(path, outpath):
"""
改变影像的数据类型
:param path: 影像的路径
:param outpath: 改变后影像的输出路径
:return: outpath
"""
src = rio.open(path)
profile = src.profile.copy()
profile['dtype'] = 'float64'
data = src.read().astype('float64')
with rio.open(outpath, mode='w', **profile) as dst:
dst.write(data)
src.close()
return outpath
2.2 栅格影像裁剪
因为下载数据时并没有对影像边界进行裁剪,而有时候我们的研究区仅仅是影像的一部分,这时候就需要一个根据矢量数据裁剪或者提取栅格影像的功能,这里使用了掩膜提取
def mask(imagePath, shpPath, outImagePath):
"""
按照矢量裁剪栅格
:param imagePath: 目标影像路径
:param shpPath: shp形状路径
:param outImagePath: 输出影像路径
:return: outImagePath
"""
shpData = GeoDataFrame.from_file(shpPath)
geo = shpData.geometry[0]
shapes = [geo.__geo_interface__]
with rio.open(imagePath) as src:
out_image, out_transform = rio.mask.mask(src, shapes, crop=True, nodata=np.nan)
out_meta = src.meta
out_meta.update({"driver": "GTiff",
"height": out_image.shape[1],
"width": out_image.shape[2],
"transform": out_transform,
# 影像压缩 用的LZW算法,可以改
"compress": 'LZW'
})
with rio.open(outImagePath, "w", **out_meta) as dest:
dest.write(out_image)
return outImagePath
2.3 计算NDVI
NDVI的计算主要是用到了红外和近红外波段,在计算NDVI的函数中,需要传入这两个波段数据,同时在计算后,需要剔除异常值,也就是不在(-1,1)之间的值,并将异常值赋值为nan,至于NDVI的归一化我并没有放在此处,因为有时候我们可能需要不归一化的值,所以把归一化另写成了一个函数。
def ndvi(red, nir):
"""
计算植被指数 ndvi
:param red: landsat8的B4波段,红外波段red
:param red: landsat8的B5波段,近红外波段nir
:return: ndvi 计算结果
"""
ndvi = (nir - red) / (nir + red)
# 去除异常值
ndvi = np.where((ndvi > -1) & (ndvi < 1), ndvi, np.nan)
return ndvi
2.4 计算WET
湿度的计算用到了6个波段,此处计算的公式是Landsat8的,如果你想计算Landsat5或7的可以把公式进行替换
def wet(blue, green, red, nir, swir1, swir2):
"""
计算湿度 wet
:param blue: landsat8的B2波段,蓝色波段blue
:param green: landsat8的B3波段,绿色波段green
:param red: landsat8的B4波段,红外波段red
:param nir: landsat8的B5波段,近红外波段nir
:param swir1: landsat8的B6波段,短波红外1 swir1
:param swir2: landsat8的B7波段,短波红外2 swir2
:return: wet 计算结果
"""
blue = blue * 0.0001
green = green * 0.0001
red = red * 0.0001
nir = nir * 0.0001
swir1 = swir1 * 0.0001
swir2 = swir2 * 0.0001
wet = blue * 0.1511 + green * 0.1972 + red * 0.3283 + nir * 0.3407 + swir1 * (-0.7117) + swir2 * (-0.4559)
return wet
2.5 计算NDBSI
NDBSI的计算需要先算出裸土指数SI和建筑指数IBI,进而再求出NDBSI,同样,按函数说明传入指定波段值就行
def ndbsi(blue, green, red, nir, swir1):
"""
计算干度 ndbsi
:param blue: landsat8的B2波段,蓝色波段blue
:param green: landsat8的B3波段,绿色波段green
:param red: landsat8的B4波段,红外波段red
:param nir: landsat8的B5波段,近红外波段nir
:param swir1: landsat8的B6波段,短波红外1 swir1
:return: ndbsi 计算结果
"""
blue = blue * 0.0001
green = green * 0.0001
red = red * 0.0001
nir = nir * 0.0001
swir1 = swir1 * 0.0001
# 计算裸土指数 si
si = ((swir1 + red) - (nir + blue)) / ((swir1 + red) + (nir + blue))
# 计算建筑指数 ibi
ibi = (2 * swir1 / (swir1 + nir) - (nir / (nir + red) + green / (green + swir1))) / \
(2 * swir1 / (swir1 + nir) + (nir / (nir + red) + green / (green + swir1)))
# 计算干度
ndbsi = (si + ibi) / 2
return ndbsi
2.6 计算LST
landsat8的第10波段是热红外波段,可以用其辐射亮度结合植被覆盖度和比辐射率来计算热度LST,
在计算植被覆盖度时此处假设最低是0.05,最高是0.95,如果有别的数据要求此处需要自行更改。
def lst(tirs1, ndvi):
"""
计算热度指数 lst
:param tirs1: landsat8的B10波段,热红外波段1 tirs1
:param ndvi: ndvi
:return:
"""
# 选择第Band10辐射亮度波段
tirs1_fsld = tirs1 * 0.1
# 计算植被覆盖度pv,假设最低值为0.05,最大值为0.95
pv = np.power((ndvi-0.05) / (0.95-0.05), 2)
# 计算比辐射率c
c = 0.004 * pv + 0.986
# 计算lst
lst = (tirs1_fsld / (1 + (0.00109 * (tirs1_fsld / 1.438)) * np.log(c))) - 273.15
return lst
2.7 数据归一化
数据归一化用的线性归一化,比较简单,如果有别的需求可以更改
def normalize(array):
"""
线性归一化
:param array: 数组
:return: 处理后的数组
"""
# 线性归一化
max = np.nanmax(array)
min = np.nanmin(array)
normalize_value = (array-min) / (max-min)
return normalize_value
2.8 PCA主成分分析计算RSEI
PCA主成分分析这部分主要是使用的sklearn包中现成的函数(我自己其实也写了,但,感觉不是很自信哈哈),其中碰到的难点应该就是做成PCA需要的面板数据时一直没有清除掉所有的nan值,不过很幸运的是碰到了位大佬—胡哥,我下楼吃饭前发给的大哥,饭没吃完,大哥就把结果发我了,然后第二天大哥写的那几句代码我学习了一天,属实牛逼。
言归正传,以下是计算代码
def rsei(ndvi, wet, ndbsi, lst):
"""
PCA主成分分析并计算RSEI
:param ndvi: ndvi
:param wet: wet
:param ndbsi: ndbsi
:param lst: lst
:return: rsei, pc_ratio(保留成分的方差百分比)
"""
# 将四个指标合成一个多维数组,此时data的shape是(4, m, n),是一个三维数据
data = np.array([wet, ndvi, lst, ndbsi])
# 对数组进行掩膜,不让nan参与PCA
panel_data = np.hstack([data[i, :, :].flatten().reshape(-1, 1) for i in range(data.shape[0])])
mask = np.isnan(panel_data).sum(axis=1) > 0
train_x = panel_data[~mask, :]
# 主成分分析
pca = PCA(n_components=1)
pca_data = pca.fit_transform(train_x)
# 保留成分的方差百分比(如果用不到可以不用返回,可以去掉)
pc_ratio = pca.explained_variance_ratio_
final_result = np.ones_like(mask) * 1.0
final_result[~mask] = pca_data.flatten()
final_result[mask] = np.NAN
result_data = final_result.reshape(data.shape[1:])
# 计算rsei
rsei = 1 - result_data
# 对rsei进行归一化
rsei = normalize(rsei)
return rsei, pc_ratio
2.9 保存计算结果数据为单波段影像
使用rasterio将数据保存为单波段tif,其中投影等信息参照读取到的landsat8的元数据信息。
def save(outPath, data, meta):
"""
保存单波段影像
:param outPath: 输出影像
:param data: 波段数据
:param meta: 原始影像的元数据
:return:
"""
meta.update({'count': 1,
'dtype': 'float64'
})
with rio.open(outPath, "w", **meta) as dest:
dest.write_band(1, data)
3 总结
计算结果,经朋友验证,和GEE计算的结果基本是保持一致的
以上如有错误,请大佬指正!感激!
完整代码和思维导图可以在公众号“壹贰叁言”回复‘RSEI’进行获取