DeepLung代码复现(一)-------------数据预处理
本文是对论文:DeepLung: Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification中所开源的代码的复现。
代码地址:https://github.com/wentaozhu/DeepLung
拿到代码首先运行./DeepLung-master/prepare.py进行数据预处理,将config_training.py中的地址改为自己本地的地址即可运行成功。
预处理的工作是使用LUNA16数据集中所有的肺部数据 (mhd格式),肺实质分割数据(mhd格式),肺结节标签数据(csv格式)转换为numpy格式。转换后的文件存储在subset0~subset9中。
每个文件夹中的每个case有6个对应的标签数据。_clean存储CT坐标(关于左边请参考上一篇文章)下裁剪肺实质的最大外接立方体后在扩充若干像素值的肺部数据,_extendbox存储肺实质的最大外接立方体扩充后的box,_label存储CT坐标下的肺结节坐标及半径,_mask存储肺实质的二值图,_origin,_spacing存储CT坐标下的原点与像素间隔。
预处理的重点函数为:
def savenpy_luna(id, annos, filelist, luna_segment, luna_data,savepath):
# 处理某个subset中的某个数据
islabel = True
isClean = True
resolution = np.array([1,1,1])
# resolution = np.array([2,2,2])
name = filelist[id]
# 读取CT图像并转换为numpy格式,在这个过程中直接把CT坐标下的图像转为图像坐标下
# 这步要理解坐标的转换,否则对后面的理解会很有困难
sliceim,origin,spacing,isflip = load_itk_image(os.path.join(luna_data,name+'.mhd'))
Mask,origin,spacing,isflip = load_itk_image(os.path.join(luna_segment,name+'.mhd'))
if isflip:
Mask = Mask[:,::-1,::-1]
# CT坐标下的图像大小 单位是mm
newshape = np.round(np.array(Mask.shape)*spacing/resolution).astype('int')
# 0代表背景 3代表左肺 4代表右肺 5代表血管
m1 = Mask==3
m2 = Mask==4
# 左肺右肺掩膜
Mask = m1+m2
# Mask中为True的三维坐标 索引,也就是肺实质的范围索引
xx,yy,zz= np.where(Mask)
# 含有肺实质的最小外接立方体
box = np.array([[np.min(xx),np.max(xx)],[np.min(yy),np.max(yy)],[np.min(zz),np.max(zz)]])
# box转换为CT坐标下的大小 只变了大小 原点偏移没改变
box = box*np.expand_dims(spacing,1)/np.expand_dims(resolution,1)
box = np.floor(box).astype('int')
margin = 5
# 扩大box 左移5 右移10 上移5 下移10 高移5 低移10
extendbox = np.vstack([np.max([[0,0,0],box[:,0]-margin],0),np.min([newshape,box[:,1]+2*margin],axis=0).T]).T
this_annos = np.copy(annos[annos[:,0]==(name)])
if isClean:
convex_mask = m1
# 进行空洞填充,并膨胀
dm1 = process_mask(m1)
dm2 = process_mask(m2)
dilatedMask = dm1+dm2
Mask = m1+m2
# 二值图像按位异或 相同为0 不同为1
extramask = dilatedMask ^ Mask
bone_thresh = 210
pad_value = 170
if isflip:
sliceim = sliceim[:,::-1,::-1]
print('flip!')
# 标准化 灰度值映射到[0,255]自然图像范围
sliceim = lumTrans(sliceim)
# 获取掩膜部分的CT图像 除掩膜外的图像设置为170
sliceim = sliceim*dilatedMask+pad_value*(1-dilatedMask).astype('uint8')
bones = (sliceim*extramask)>bone_thresh
# 将原图中骨头的灰度值改为210
sliceim[bones] = pad_value
# 重采样(121,512,512)-> (302, 390, 390) 原始图像变为真实坐标下的图像
# 这里函数名是重采样,实则是将图像坐标系下的数据转化为CT坐标下的数据
sliceim1,_ = resample(sliceim,spacing,resolution,order=1)
# 将重采样后的图像裁取含有肺实质的最大范围 这里截取图像都是在CT坐标系下
sliceim2 = sliceim1[extendbox[0,0]:extendbox[0,1],
extendbox[1,0]:extendbox[1,1],
extendbox[2,0]:extendbox[2,1]]
# 增加一个维度
sliceim = sliceim2[np.newaxis,...]
np.save(os.path.join(savepath, name+'_clean.npy'), sliceim)
np.save(os.path.join(savepath, name+'_spacing.npy'), spacing)
np.save(os.path.join(savepath, name+'_extendbox.npy'), extendbox)
np.save(os.path.join(savepath, name+'_origin.npy'), origin)
np.save(os.path.join(savepath, name+'_mask.npy'), Mask)
if islabel:
this_annos = np.copy(annos[annos[:,0]==(name)])
label = []
if len(this_annos)>0:
for c in this_annos:
# 结节位置从CT坐标系转为图像坐 标系
pos = worldToVoxelCoord(c[1:4][::-1],origin=origin,spacing=spacing)
if isflip:
pos[1:] = Mask.shape[1:3]-pos[1:]
# 加入图像坐标系下的半径
label.append(np.concatenate([pos,[c[4]/spacing[1]]]))
#shape(1186,4) 1186 case数 4 肺结节的坐标及半径的真实坐标
label = np.array(label)
if len(label)==0:
label2 = np.array([[0,0,0,0]])
else:
# shape = (4, 1186)
label2 = np.copy(label).T
# 结节在真实坐标系下的位置 * 体素间隔 / 1 = 世界世界坐标系下的(体素坐标 - 原点)
label2[:3] = label2[:3]*np.expand_dims(spacing,1)/np.expand_dims(resolution,1)
# 真实坐标系下的结节半径 * 体素间隔 / 1 = 世界坐标系下的半径
label2[3] = label2[3]*spacing[1]/resolution[1]
# 以CT坐标下裁剪后的图像原点为原点
label2[:3] = label2[:3]-np.expand_dims(extendbox[:,0],1)
label2 = label2[:4].T
np.save(os.path.join(savepath,name+'_label.npy'), label2)
print(name)接下来分步看下每步的结果:
islabel = True
isClean = True
resolution = np.array([1,1,1])
# resolution = np.array([2,2,2])
name = filelist[id]
# 读取CT图像并转换为numpy格式,在这个过程中直接把CT坐标下的图像转为图像坐标下
sliceim,origin,spacing,isflip = load_itk_image(os.path.join(luna_data,name+'.mhd'))
Mask,origin,spacing,isflip = load_itk_image(os.path.join(luna_segment,name+'.mhd'))
if isflip:
Mask = Mask[:,::-1,::-1]def load_itk_image(filename):
# 通过mhd头文件判断是否是flip
with open(filename) as f:
# 获取mhd的头信息 判断是否需要flip
contents = f.readlines()
line = [k for k in contents if k.startswith('TransformMatrix')][0]
transformM = np.array(line.split(' = ')[1].split(' ')).astype('float')
transformM = np.round(transformM)
if np.any( transformM!=np.array([1,0,0, 0, 1, 0, 0, 0, 1])):
isflip = True
else:
isflip = False
# 读取mhd图像转为numpy
itkimage = sitk.ReadImage(filename)
numpyImage = sitk.GetArrayFromImage(itkimage)
# 注意此处的reversed 后面会使用
numpyOrigin = np.array(list(reversed(itkimage.GetOrigin())))
numpySpacing = np.array(list(reversed(itkimage.GetSpacing())))
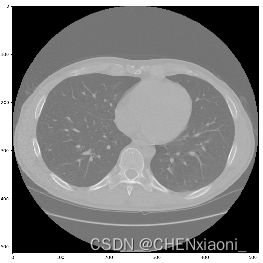
return numpyImage, numpyOrigin, numpySpacing,isflipsliceim与Mask是读取的的图像与分割标签转为numpy格式的显示,在转numpy过程中同时也将CT坐标系转为图像坐标系。以该case为例,numpy.shape = (121,512,512),图像值的范围为[2103,-3024],分割标签的值的范围为[0,5] 0代表背景 3代表左肺 4代表右肺 5代表血管。
newshape是通过图像坐标下的shape计算的得到的CT坐标下的图像大小,单位是mm。前文程序中的reversed的原因是读取到的spacing的顺序是(x,y,z),而Mask.shape的顺序是(z,y,x)故需要reversed才能正确对应。
# CT图像中的图像大小
newshape = np.round(np.array(Mask.shape)*spacing/resolution).astype('int')
# 0代表背景 3代表左肺 4代表右肺 5代表血管
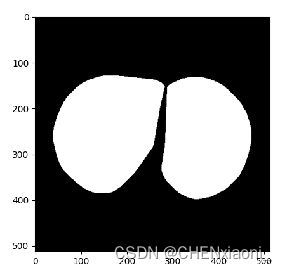
m1 = Mask==3
m2 = Mask==4
# 左肺右肺掩膜
Mask = m1+m2
# Mask中为True的三维坐标 索引
xx,yy,zz= np.where(Mask)
# 含有肺实质的最小外接立方体
box = np.array([[np.min(xx),np.max(xx)],[np.min(yy),np.max(yy)],[np.min(zz),np.max(zz)]])
# box转换为CT图像中大小 只变了大小 原点偏移没改变
box = box*np.expand_dims(spacing,1)/np.expand_dims(resolution,1)
box = np.floor(box).astype('int')
margin = 5
# 扩大box 左移5 右移10 上移5 下移10 高移5 低移10
extendbox = np.vstack([np.max([[0,0,0],box[:,0]-margin],0),np.min([newshape,box[:,1]+2*margin],axis=0).T]
this_annos = np.copy(annos[annos[:,0]==(name)])
if isClean:
convex_mask = m1
# 进行空洞填充,并膨胀
dm1 = process_mask(m1)
dm2 = process_mask(m2)
dilatedMask = dm1+dm2
Mask = m1+m2
# 二值图像按位异或 相同为0 不同为1
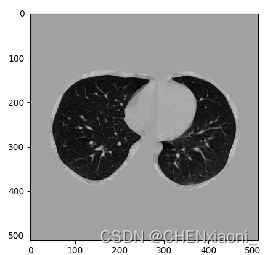
extramask = dilatedMask ^ MaskdilatedMask是分割标签的肺实质通过空洞填充与膨胀后的图像,extramask膨胀后的图像与原maks取异或,大致提取肺周围的骨骼部分。
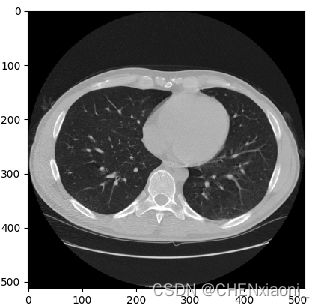
# 标准化 灰度值映射到[0,255]自然图像范围
sliceim = lumTrans(sliceim)
# 获取掩膜部分的CT图像 除掩膜外的图像设置为170
sliceim = sliceim*dilatedMask+pad_value*(1-dilatedMask).astype('uint8')
bones = (sliceim*extramask)>bone_thresh
# 将原图中骨头的灰度值改为210
sliceim[bones] = pad_valuesliceim:通过lumTrans函数将原图标准化,灰度范围为自然图像范围[0,255]。
sliceim = sliceim * dilatedMask 原图与腐蚀后的肺实质相乘。
sliceim = sliceim * dilatedMask + pad_value * (1 - dilatedMask).astype('uint8') 将除去腐蚀过的肺实质部分的其他部分灰度值设为170。
提取骨骼部分,并将骨骼部分的灰度值设为210。
最后将处理好的肺实质进行resample,然后提取含有肺实质的最小外接矩形(经过一定marging膨胀)。这里的resample并不是重采样,函数内是将图像坐标下的图像转化为CT坐标下的图形。
# 重采样 (121,512,512)-> (302, 390, 390) 原始图像变为真实坐标下的图像
sliceim1,_ = resample(sliceim,spacing,resolution,order=1)
# 将重采样后的图像裁取含有肺实质的最大范围
sliceim2 = sliceim1[extendbox[0,0]:extendbox[0,1],
extendbox[1,0]:extendbox[1,1],
extendbox[2,0]:extendbox[2,1]]