sklearn初探(七):DBSCAN算法聚类及可视化
sklearn初探(七):DBSCAN算法聚类及可视化
前言
本次任务采用DBSCAN算法对青蛙叫声的MFCC文件进行聚类分析,使用f-m指数与调整后兰德指数进行评分与调参,使用t-sne对聚类结果进行降维,使用matplotlib将结果可视化。数据集链接及完整源代码见文末。
概述
DBSCAN算法
The DBSCAN 算法将簇视为被低密度区域分隔的高密度区域。由于这个相当普遍的观点, DBSCAN发现的簇可以是任何形状的,与假设簇是凸的 K-means 相反。 DBSCAN 的核心概念是 core samples, 是指位于高密度区域的样本。 因此一个簇是一组核心样本,每个核心样本彼此靠近(通过某个距离度量测量) 和一组接近核心样本的非核心样本(但本身不是核心样本)。算法中的两个参数, min_samples 和 eps,正式的定义了我们所说的 稠密(dense)。较高的 min_samples 或者较低的 eps 都表示形成簇所需的较高密度。
更正式的,我们定义核心样本是指数据集中的一个样本的 eps 距离范围内,存在 min_samples 个其他样本,这些样本被定为为核心样本的邻居( neighbors) 。这告诉我们,核心样本在向量空间的稠密区域。一个簇是一个核心样本的集合,可以通过递归来构建,选取一个核心样本,查找它所有的邻居样本中的核心样本,然后查找新获取的核心样本的邻居样本中的核心样本,递归这个过程。 簇中还具有一组非核心样本,它们是簇中核心样本的邻居的样本,但本身并不是核心样本。 显然,这些样本位于簇的边缘。
当参数min_samples 主要表示算法对噪声的容忍度(当处理大型噪声数据集时, 需要考虑增加该参数的值), 针对具体地数据集和距离函数,参数eps 如何进行合适地取值是非常关键,这通常不能使用默认值。参数eps控制了点地领域范围。如果取值太小,大部分地数据并不会被聚类(被标注为 -1 代表噪声); 如果取值太大,可能会 导致 相近 的多个簇被合并成一个,甚至整个数据集都被分配到一个簇。
注意,与k-means聚类不同的是,使用DBSCAN并不能确定结果中聚类的数量,这意味着我们要自己调参。
调整后兰德指数
在已知真实簇标签分配(the ground truth class assignment) labels_true 和我们的聚类算法基于相同样本所得到的 labels_pred,调整后的兰德指数(adjusted Rand index) 是一个函数,用于测量两个簇标签分配的值的 相似度 ,忽略排列(permutations)和 with chance normalization:
>>> from sklearn import metrics
>>> labels_true = [0, 0, 0, 1, 1, 1]
>>> labels_pred = [0, 0, 1, 1, 2, 2]
>>> metrics.adjusted_rand_score(labels_true, labels_pred)
0.24...在预测的标签列表中重新排列 0 和 1, 把 2 重命名为 3, 得到相同的得分:
>>> labels_pred = [1, 1, 0, 0, 3, 3]
>>> metrics.adjusted_rand_score(labels_true, labels_pred)
0.24...另外, adjusted_rand_score 是 对称的(symmetric) : 交换参数不会改变得分。它可以作为 共识度量(consensus measure):
>>> metrics.adjusted_rand_score(labels_pred, labels_true)
0.24...完美的标签的得分为 1.0。
fowlkes-mallows指数
当样本的已标定的真实类分配已知时,可以使用 Fowlkes-Mallows 指数 (sklearn.metrics.fowlkes_mallows_score) 。Fowlkes-Mallows 得分 FMI 被定义为 成对的准确率和召回率的几何平均值:
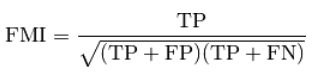
其中的 TP 是 真正例(True Positive) 的数量(即,真实标签组和预测标签组中属于相同簇的点对数),FP 是 假正例(False Positive) (即,在真实标签组中属于同一簇的点对数,而不在预测标签组中),FN 是 假负例(False Negative) 的数量(即,预测标签组中属于同一簇的点对数,而不在真实标签组中)。
得分范围为 0 到 1。较高的值表示两个簇之间的良好相似性。
>>> from sklearn import metrics
>>> labels_true = [0, 0, 0, 1, 1, 1]
>>> labels_pred = [0, 0, 1, 1, 2, 2]>>> metrics.fowlkes_mallows_score(labels_true, labels_pred)
0.47140...在预测的标签列表中重新排列 0 和 1, 把 2 重命名为 3, 得到相同的得分:
>>> labels_pred = [1, 1, 0, 0, 3, 3]
>>> metrics.fowlkes_mallows_score(labels_true, labels_pred)
0.47140...完美的标签得分是 1.0。
数据分割
import pandas as pd
frog_data = pd.read_csv("../datas/Frogs_MFCCs.csv")
tLabel = []
for family in frog_data['Family']:
if family == "Leptodactylidae":
tLabel.append(0)
elif family == "Dendrobatidae":
tLabel.append(1)
elif family == "Hylidae":
tLabel.append(2)
else:
tLabel.append(3)
first_set = frog_data[['MFCCs_ 1', 'MFCCs_ 5', 'MFCCs_ 9', 'MFCCs_13', 'MFCCs_17', 'MFCCs_21']]
second_set = frog_data[['MFCCs_ 3', 'MFCCs_ 7', 'MFCCs_11', 'MFCCs_15', 'MFCCs_19']]DBSCAN处理
fit一下。
db = cluster.DBSCAN(eps=0.1011, min_samples=115, n_jobs=-1)
db.fit(first_set)
r = pd.concat([first_set, pd.Series(db.labels_, index=first_set.index)], axis=1)
r.columns = list(first_set.columns) + [u'聚类类别']
# print(r)
r.to_excel("../output/dbscanSet_1.xlsx")调参
dbscan需要我们调参。本次任务需要调参以达到这样的效果:聚类结果为四类,且评分尽量高。
Set1
| eps | min_samples | 聚类数量 | fowlkes-mallows得分 |
|---|---|---|---|
| 0.1 | 150 | 4 | 0.6762988391651638 |
| 0.1 | 175 | 4 | 0.6712206459932587 |
| 0.1 | 200 | 3 | - |
| 0.1 | 125 | 4 | 0.6813089522934992 |
| 0.1 | 120 | 4 | 0.6815870169183239 |
| 0.1 | 130 | 4 | 0.6805885740745339 |
| 0.1 | 115 | 4 | 0.6830254509032943 |
| 0.1 | 110 | 5 | - |
| 0.09 | 115 | 4 | 0.6642916488855524 |
| 0.11 | 115 | 6 | - |
| 0.095 | 115 | 4 | 0.6748581101560627 |
| 0.105 | 115 | 5 | - |
| 0.101 | 115 | 4 | 0.6848332275397843 |
| 0.102 | 115 | 5 | - |
| 0.1015 | 115 | 5 | - |
| 0.1011 | 115 | 4 | 0.6849251120786362 |
Set2
| eps | min_samples | 聚类数量 | fowlkes-mallows得分 |
|---|---|---|---|
| 0.1 | 115 | 4 | 0.6674327901587477 |
| 0.1 | 120 | 4 | 0.6672727687106378 |
| 0.1 | 110 | 4 | 0.6690055278219934 |
| 0.1 | 105 | 4 | 0.6700344543118204 |
| 0.1 | 100 | 4 | 0.6720072870174673 |
| 0.1 | 95 | 4 | 0.6729892684302224 |
| 0.1 | 90 | 4 | 0.6734227611274493 |
| 0.1 | 85 | 5 | - |
| 0.105 | 90 | 5 | - |
| 0.101 | 90 | 4 | 0.6743460020206788 |
| 0.102 | 90 | 4 | 0.6759196726408404 |
| 0.103 | 90 | 5 | - |
| 0.1025 | 90 | 5 | - |
| 0.1021 | 90 | 4 | 0.6759196726408404 |
| 0.1022 | 90 | 5 | - |
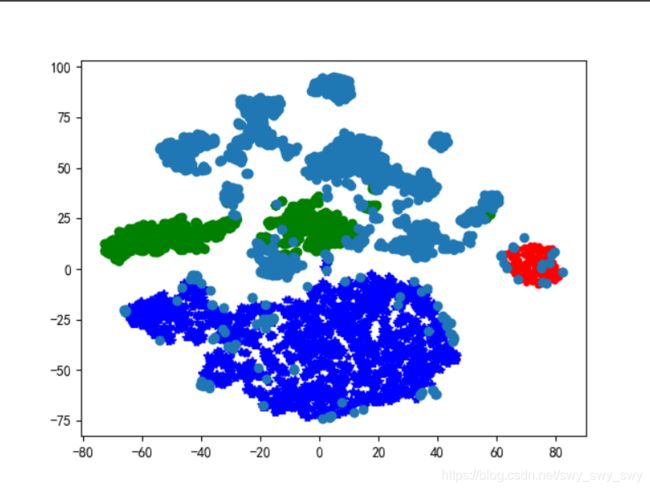
t-sne降维
t_sne_db_1 = TSNE()
t_sne_db_1.fit(first_set)
t_sne_db_1 = pd.DataFrame(t_sne_db_1.embedding_, index=first_set.index)可视化
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
dd = t_sne_db_1[r[u'聚类类别'] == 0]
plt.plot(dd[0], dd[1], 'r.')
dd = t_sne_db_1[r[u'聚类类别'] == 1]
plt.plot(dd[0], dd[1], 'go')
dd = t_sne_db_1[r[u'聚类类别'] == 2]
plt.plot(dd[0], dd[1], 'b*')
dd = t_sne_db_1[r[u'聚类类别'] == -1]
plt.plot(dd[0], dd[1], 'o')
plt.savefig("../output/dbscanSet_1.png")
plt.clf()源代码
这里就只放一个训练集的吧,我没做封装。
import pandas as pd
from sklearn import cluster
from sklearn import metrics
import matplotlib.pyplot as plt
from sklearn.manifold import TSNE
frog_data = pd.read_csv("../datas/Frogs_MFCCs.csv")
tLabel = []
for family in frog_data['Family']:
if family == "Leptodactylidae":
tLabel.append(0)
elif family == "Dendrobatidae":
tLabel.append(1)
elif family == "Hylidae":
tLabel.append(2)
else:
tLabel.append(3)
first_set = frog_data[['MFCCs_ 1', 'MFCCs_ 5', 'MFCCs_ 9', 'MFCCs_13', 'MFCCs_17', 'MFCCs_21']]
second_set = frog_data[['MFCCs_ 3', 'MFCCs_ 7', 'MFCCs_11', 'MFCCs_15', 'MFCCs_19']]
# DBSCAN begins
db = cluster.DBSCAN(eps=0.1011, min_samples=115, n_jobs=-1)
db.fit(first_set)
r = pd.concat([first_set, pd.Series(db.labels_, index=first_set.index)], axis=1)
r.columns = list(first_set.columns) + [u'聚类类别']
# print(r)
r.to_excel("../output/dbscanSet_1.xlsx")
p_labels = list(db.labels_)
with open(scoreFile, "a") as sf:
sf.write("By DBSCAN, the f-m_score of Set_1 is: " + str(
metrics.fowlkes_mallows_score(tLabel, p_labels)) + "\n")
sf.write("By DBSCAN, the rand_score of Set_1 is: " + str(
metrics.adjusted_rand_score(tLabel, p_labels)) + "\n")
t_sne_db_1 = TSNE()
t_sne_db_1.fit(first_set)
t_sne_db_1 = pd.DataFrame(t_sne_db_1.embedding_, index=first_set.index)
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
dd = t_sne_db_1[r[u'聚类类别'] == 0]
plt.plot(dd[0], dd[1], 'r.')
dd = t_sne_db_1[r[u'聚类类别'] == 1]
plt.plot(dd[0], dd[1], 'go')
dd = t_sne_db_1[r[u'聚类类别'] == 2]
plt.plot(dd[0], dd[1], 'b*')
dd = t_sne_db_1[r[u'聚类类别'] == -1]
plt.plot(dd[0], dd[1], 'o')
plt.savefig("../output/dbscanSet_1.png")
plt.clf()数据集
https://download.csdn.net/download/swy_swy_swy/12409033