本周开课 | 第 5 期全基因组/外显子组家系分析理论和实战
福利公告:前4期《临床基因组学数据分析实战》线上/线下课程已圆满结束。现于2022年4月22-24,安排第五期课程在北京。(线上课是通过腾讯会议实时直播线下课,实时互动,并录制有视频回放,无限期观看)。报名参加线上直播课的老师可在365天内选择参加同课程的一次线下课。期待和大家的线上线下相识。
临床基因组学线上/线下开课时间:2022/4/22-2022/02/24
宏基因组线上/线下开课时间:2022/5/27-2022/5/29
扩增子线上/线下开课时间:2022/7/15-2022/7/17
转录组线上/线下开课时间:2022/7/29-2022/7/31
报名链接:http://www.ehbio.com/Training/
近几年测序成本急速降低,基因组测序逐渐成为临床上的常规检测。临床基因组测序一般包括全外显子测序 (WES)或全基因组测序 (WGS),极大地加深了我们对于疾病规律的认识,是检测单基因遗传病或罕见病的利器,同时也越来越多地用于常见病的遗传风险评估。
目前很多临床科室积累了一些病人的WGS/WES数据,但由于缺乏大数据分析平台或专业的生信人才而被长期搁置。一些外包或第三方的分析由于分析手段有限,缺乏对疾病背景信息的深入了解和沟通,经常报告不出来可疑的或明确的致病位点,也难以做出高水平文章所需的图和表格,导致既无法报告给患者,也难以用于科学研究和成果发表。相关政策和法规也不鼓励样本外送及外包分析,此外人类遗传资源的采集、保藏、利用和对外合作也应当遵守《人类遗传资源管理条例》。因此临床科室自己独立分析是满足相关临床需求,进行高水平研究,以及保护遗传样本资源的根本办法。
图1. 利用临床基因组测序发现致病遗传变异的工作流程
在这样的大背景下,易生信培训团队推出《临床基因组学数据分析实战》专题培训,为临床医生和相关领域研究人员提供一条走进临床基因组生信大门的捷径,为同行提供学习和交流的平台,切实助力大家理解分析原理,完成实战分析。
本课程帮助您真正实现临床基因组学数据分析,并根据自己课题的背景优化分析方案。在本课程学习过程中,除了学习基于Linux和R语言的标准分析,还包含运用丰富的公共数据库、疾病数据库对突变位点进行注释和可视化,也包含了大量下游分析工具和方法,如:一代测序拼接和测序峰图绘制,多序列比对,突变所处的蛋白结构域预测,突变蛋白翻译后修饰的变化,突变对蛋白三维结构的影响预测(表面电荷、化学键、空间位阻、亲疏水性等),达到临床应用与学术研究双受益。
在课程时间的设置上,独创四段式教学(3天集中授课+自行练习2周+再集中讲解答疑+上课视频回看反复练习),“教—练—答—用”四个环节统一协调,循序渐进,使学员获得独立分析临床基因组数据的能力。
在分析平台上,提前帮助您在自己的笔记本电脑或工作站安装Windows+Linux的双系统;与此同时提供1个月的免费Linux云服务器的使用权限 (配备了全固态硬盘);各个平台上均配备大量,完善的公共数据库资源。确保程序写作、数据传输和实战分析的顺利进行。
课程简介
请详细阅读课程简介,如果以下内容您全精通,不必参加此培训。
为满足广大读者进一步学习的需求,易生信课程开发团队 (现运营《生信宝典》和《聊生信》两个公众号)经长期规划,筹备和专家研讨,现组织和开展临床基因组学专题培训课程,以便进一步普及和交流临床基因组学分析技术,手把手带您快速入门,节约宝贵的时间,助力科研成果早日产出。
本课程一共3天,每天6节课,共18节课,全部课程均理论与实战结合 (只要课上讲的内容,都是要带你亲自实现的分析)。
课程覆盖:
临床基因组数据分析平台搭建 (操作系统与软件);
基因组与外显子组学研究概述;
测序与数据预处理;
经典文章思路与图表解读;
全基因组比对与短变异 (SNV & InDel)检测;
数据库详解 (OMIM, ClinVar, 1000G, ExAC/gnomAD, MalaCards/GeneCards, SWISS-MODEL/AlphaFold);
变异注释和过滤, 变异有害性预测 (SIFT, PolyPhen, CADD, RVIS);
通路和表型富集分析 (GO, KEGG, Reactome, DisGeNET, Human Phenotype Ontology, OMIM Disease等);
蛋白-蛋白互作网络分析;
Sanger测序拼接及峰图绘制, 多序列比对与保守性分析;
突变对蛋白修饰 (磷酸化和糖基化)的影响预测, 突变所在蛋白结构域或保守区预测;
突变对蛋白三维结构影响 (表面电荷, 化学键, 空间位阻, 亲疏水性和残基位置等);
CNV分析,家系分析及ACMG变异评级。
注意:
本课程不包括
SV等结构变异分析。本课程不能直接用于肿瘤数据的分析。
课程大纲
每节课1小时一个主题,理论结合实战,学懂原理,实战实操,全是多年经验和代码的无私分享。下面是课程安排,如:11代表第一天第一节课,23代表第二天第三节课;01、02和03表示课前准备工作 (提前观看相关软件安装等视频和提前进行软件安装直播)。
| 编号 | 主题 | 简介 |
|---|---|---|
| 01 | 操作系统及软件 | Bioconda, Git, R, Rstudio, R包等, Ubuntu子系统 |
| 02 | Linux和R基础 | Linux系列教程, Linux常用命令, 生信程序基础课节选 |
| 03 | Linux软件安装 | Conda安装与配置, 相关软件安装 |
| 11 | 基因组与外显子组学研究概述 | 基本概念, 发展史, 常用技术适用范围 |
| 12 | 测序与数据预处理 | NGS, MultiQC, 移除接头和低质量碱基, Trim Galore |
| 13 | 经典文章思路与图表解读 (一) | 文献解读, 多篇文章研究思路, 图表在文章中的意义和解读 |
| 14 | 全基因组比对与短变异 (SNV & InDel)检测 | BWA, GATK, Samtools和Vcftools等 |
| 15 | 全基因组比对与短变异 (SNV & InDel)检测 | BWA, GATK, Samtools和Vcftools等 |
| 16 | 全基因组比对与短变异 (SNV & InDel)检测 | BWA, GATK, Samtools和Vcftools等 |
| 21 | 相关数据库详解 | OMIM, ClinVar, gnomAD, 1000G和AlphaFold等 |
| 22 | 变异注释, 过滤, 有害性及致病性预测 | SnpEff变异注释, gnomAD和1000G人群频率过滤, SIFT, Polyphen, CADD和RVIS变异有害性预测, ClinVar/ClinGen, OMIM和Orphanet变异致病性。 |
| 23 | 变异统计与绘图 | GO/KEGG/Reactome通路富集, OMIM Disease, DisGeNET, ClinVar和Human Phenotype Ontology疾病或表型富集, 蛋白组织特异性表达富集, PPI网络, 变异热图, Circles图绘制 |
| 24 | 变异统计与绘图 | GO/KEGG/Reactome通路富集, OMIM Disease, DisGeNET, ClinVar和Human Phenotype Ontology疾病或表型富集, 蛋白组织特异性表达富集, PPI网络, 变异热图, Circles图绘制 |
| 25 | 变异统计与绘图 | GO/KEGG/Reactome通路富集, OMIM Disease, DisGeNET, ClinVar和Human Phenotype Ontology疾病或表型富集, 蛋白组织特异性表达富集, PPI网络, 变异热图, Circles图绘制 |
| 26 | 家系分析 | 新发 (De novo)突变, 隐性复合杂合变异分析 |
| 31 | 蛋白质生物学概述 | 生物分子的强, 弱相互作用及种类, 蛋白结构和功能基础, 酶的功能原理, 结构域, 磷酸化等翻译后修饰原理, 蛋白互作的意义 |
| 32 | 序列拼接、比对和蛋白质结构预测 | Sanger序列拼接, 测序峰图, 多序列比对, 突变对蛋白修饰(磷酸化和糖基化)的影响预测, 结构域预测, 突变对蛋白三维结构影响, 突变对蛋白表面电荷, 化学键, 空间位阻, 亲疏水性的影响 |
| 33 | 串讲, 答疑, 第二/三篇文献解读 | 临床基因组分析套路(1), 临床基因组分析套路(2), 现场回答每人一问 |
| 34 | 分析环境配置, 导入自己数据分析的使用 | 演示如何分析自己的数据 (Linux软件安装 + Ubuntu子系统) |
| 41 | 答疑-线上 | 答疑, 考试内容串讲 |
教程内容简介如下:
一、临床基因组数据分析流程
包含从下机数据质控,公共数据准备,基因组比对,到变异检测,过滤和有害性预测等全流程。
图2. 临床基因组学测序数据分析流程
对原始测序数据去除低质量碱基和接头序列,确保准确地反映样本身的序列信息。
图3. 测序数据质量控制
流程中涉及大量专业软件,方法和分析工具,尤其是大量下游分析工具,全面解析候选变异的致病机制,分析结果满足临床和科研两个方面的需求。
图4. 软件,方法和工具
二、完备的数据库资源
课程中配套了最新权威疾病数据库,如OMIM,ClinVar和Orphanet;广泛覆盖的人群变异频率数据库,如gnomAD和千人基因组计划(1000G)数据库;蛋白质三维结构数据库,如SWISS-MODEL和AlphaFold等。
图5. 调用丰富的数据库
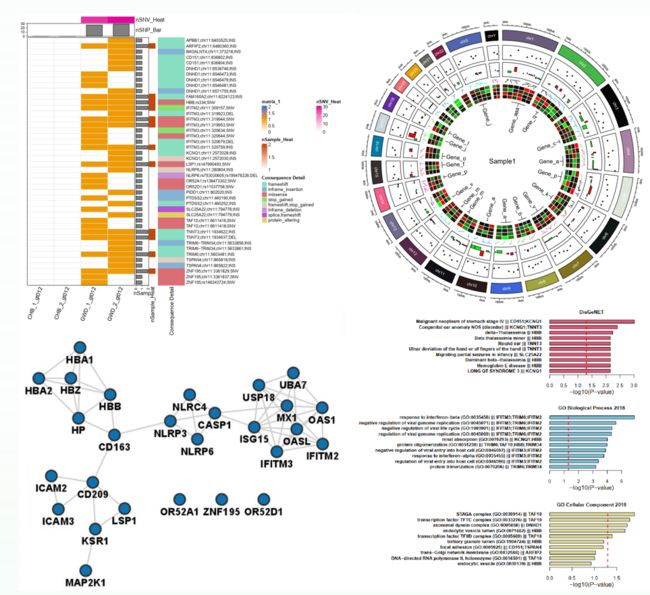
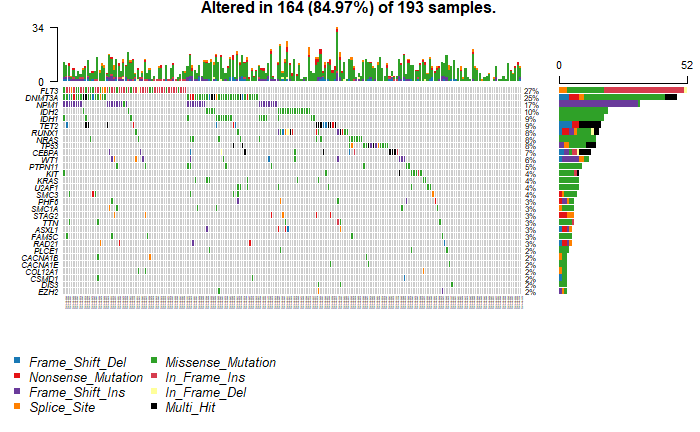
三、生信图表绘制
使用R语言等统计与可视化工具,绘制常用生信相关图形,包括:热图 (变异水平),maftools图 (基因水平),基因的结构、变异位置及结构域标记 (棒棒糖图),基因组Circos图,IGV基因组浏览器,PPI网络图,基因功能的富集结果图。
图6. 常用生信图形的绘制。
四、更多下游分析与可视化
在培训时,我们将结合发表的高水平文章,进一步讲解常用分析图的原理和使用范围,让你不仅读懂图,更知道如何应用于自己的研究,并亲自轻松完成绘图。下面的图形也都会手把手教会你如何绘制:
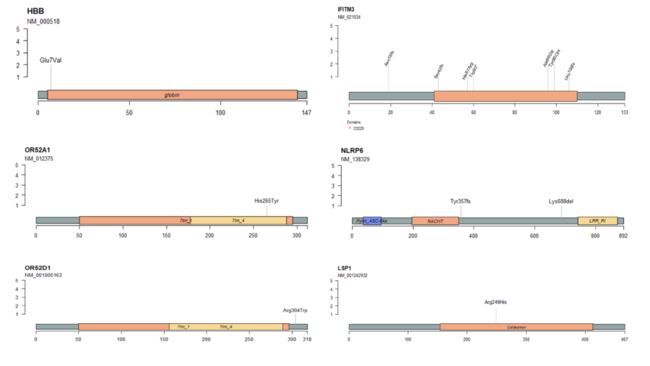
1. Sanger测序拼接和绘图,多序列比对,突变所在的蛋白结构域和翻译后修饰位点预测
图7. 序列拼接、比对、蛋白结构域和修饰预测。
2. 基于蛋白三维结构的分析,研究突变对表面电荷、化学键、空间位阻、亲疏水性和残基位置等的影响,进而预测其对蛋白具体功能的影响,解释疾病发生的分子机制!
图8. 分析变异对蛋白结构的影响。
学习完本课程,你能得到什么?
1. 深彻理解临床测序数据的基本思想
2. 临床基因组学分析和可视化的全套流程
3. 应用于各个领域的分析经验,代码和发表级别的结果可视化
4. 丰富的软件和数据库的使用经验
几十款本领域软件的安装,使用教程
常用公共数据库,疾病数据库的理解和使用
5. 在临床应用的同时,也能享受科研的乐趣,做高大上且深刻的疾病机制解析
6. 高要求的结果可视化
生信热图 (变异水平),maftools图 (基因水平),基因的结构、变异位置及结构域标记 (棒棒糖图),基因组Circos图,IGV基因组浏览器,PPI网络图,基因功能的富集结果图;
下游分析与可视化:
Sanger测序拼接和峰图,多序列比对结果图,蛋白结构域及翻译后修饰模式图,突变蛋白的三维结构图
主讲教师
易生信课程开发团队。由团队导师,课程顾问和多名一线生信工程师组成。
生信工程师
宋红卫:
外显子组,基因组和转录组。
叶明皓:
细菌基因组,蛋白质生物学。
孙馥香:
16s,宏基因组和多组学联合分析。
团队导师
陈同:博士,中国中医科学院,2015毕业于中科院遗传发育所,生物信息博士,在Cell Stem Cell(IF=23.2,第一作者兼封面文章),Nature Communications,Nucleic Acids Research X 2,Protein & Cell等高水平杂志以第一作者或主要作者发表文章,运营有十万+关注的《生信宝典》微信公众号,给你不一样的学习生信体验。
往期课程瞬间
授课模式
本课程以讲解流程和实际操作为主,采用独创四段式教学:
第一阶段 3天集中授课;
第二阶段 自行练习2周;
第三阶段 在线直播答疑;
第四阶段 培训视频继续学习;
实现教-练-答-用四个环节的统一协调。
培训时间
每天早9点到晚6点,半封闭式教学 (最后1小时为集中讨论时间,最后一天会稍微提前一些,多留出时间讨论,也方便老师乘车返回)
报到时间:课程当天
授课地点
在线会议平台,如腾讯会议 (线上为线下的实时直播,可随时互动)。
线下地址:
北京:北京市西城区鼓楼西大街41号院动信通科技园3号楼221 (北京会议较多,线下具体位置开课前1周通知);附近有喆啡,摄影主题,七天等酒店,请自行预定。
广州:三环以里,线下具体位置开课前1周通知。附近酒店,请自行预定。
线下课程3天会提供午餐,如果有忌口请提前告知。
课程价格
开课两周前报名 4500 元/人 (住宿自行解决,提供培训期间午餐)
名额有限,预报速速
提供易汉博基因科技实习机会或工作机会
易生信同时推出多门相关课程,连报优惠——同时选2门课,各减100;三门课9各减200。或者多人组团尊享团购优惠!
课程福利
座位按报名并缴费(或预缴费)成功顺序从前到后龙摆尾式排序
赠送程序基础课一份 (http://bioinfo.ke.qq.com)
多人 (N,10>N>1) 组团报名并同时缴费,每人可减免N-1百元 (最高500)
附推荐与分享对应的招生信息到朋友圈,截图发到[email protected] 可获得200元生信宝典腾讯课堂课程优惠券(可拆分供多个腾讯课堂课程使用,不用于直播或线下课)
易生信同时推出多门相关课程,连报优惠——同时选2门课,各减100;
三门课9各减200。或者多人组团尊享团购优惠!
注意事项 *
需自备笔记本电脑,推荐使用Win10系统,4G以上内存(推荐8G)。
课程实践根据需要会提供云计算平台
培训班所有数据,文档为内部资料,仅供参阅,未经允许不得翻印外传登刊
上课期间禁止录音,录像
成功付款的学员,若临时有紧急事情不能到来的,可申请延期,更换后续培训班;也可申请退款
若开课2周 (含) 前申请退款可退还85%费用;开课3个工作日 (含) 前申请退款退还70%的费用 (若已开发票需承担相应手续费)
不可先延期再退款