R语言实例:基于Boston数据集的数据分析报告——用 logistic 回归、LDA(线性判别法)、K 临近法(k=1 和 k=5)构建分类模型。目的是预测一个区域的犯罪率是否高于所有犯罪率的中位数
文章目录
- 问题
- Boston 数据集
-
- 查看数据集
- 数据描述
- 构建分类模型
-
- 数据可视化
- logistic 分类模型
-
- 构建分类模型的因变量
- 构建三个不同自变量的模型
- 交叉验证
- 结果分析
- LDA 回归模型
-
- 结果分析
- K 临近模型
- 最优子集构建回归模型
-
- 最优子集
- 划分学习和测试数据集
- 预测犯罪率
- 代码
问题
请分析 Boston 数据集,并撰写一个数据分析报告。
在报告中主要分析并回答以下两个问题。
-
用 logistic 回归、LDA(线性判别法)、K 临近法(k=1 和 k=5)构建分类模型。目的是预测一个区域的犯罪率是否高于所有犯罪率的中位数。
在构建每种类型的模型时,请分别选择三组(三个不同子集的)自变量。从三组自变量构造的模型中分别选出一个你认为最好的,你的选择应当基于交叉验证法。请讨论你得到的结果。
-
用最优子集的方法构建回归模型,预测一个区域的犯罪率。
Boston 数据集
查看数据集
> library(MASS)
> head(Boston) # 查看数据前6行
crim zn indus chas nox rm age dis rad tax ptratio black lstat medv
1 0.00632 18 2.31 0 0.538 6.575 65.2 4.0900 1 296 15.3 396.90 4.98 24.0
2 0.02731 0 7.07 0 0.469 6.421 78.9 4.9671 2 242 17.8 396.90 9.14 21.6
3 0.02729 0 7.07 0 0.469 7.185 61.1 4.9671 2 242 17.8 392.83 4.03 34.7
4 0.03237 0 2.18 0 0.458 6.998 45.8 6.0622 3 222 18.7 394.63 2.94 33.4
5 0.06905 0 2.18 0 0.458 7.147 54.2 6.0622 3 222 18.7 396.90 5.33 36.2
6 0.02985 0 2.18 0 0.458 6.430 58.7 6.0622 3 222 18.7 394.12 5.21 28.7
数据描述
在命令行中输入?Boston命令,Rstudio 界面出现该数据集的解释界面,如图所示:

Boston数据集描述波士顿郊区的房价,该数据集共506行、14列。
| 变量 | 含义 |
|---|---|
| crim | 城镇人均犯罪率 |
| zn | 25000平方英尺以上地块的住宅用地比例 |
| indus | 每个城镇的非零售业务面积比例 |
| chas | Charles River 哑变量1(如果道沿河而行,该项数值为 1,否则为0) |
| nox | 氮氧化物浓度(千万分之一) |
| rm | 每个住宅的平均房间数 |
| age | 1940年以前建造的自有住房比例 |
| dis | 五个波士顿就业中心距离的加权平均数 |
| rad | 辐射状公路通达性指数 |
| tax | 按每10,000美元计算的全值物业税税率 |
| ptratio | 城镇师生比例 |
| black | 1000 ( B k − 0.63 ) 2 1000(Bk-0.63)^ 2 1000(Bk−0.63)2,其中 B k Bk Bk是城镇黑人的比例 |
| lstat | 底层阶级人口占比(%) |
| medv | 业主自住住宅的中位价值(以1000美元为单位) |
构建分类模型
数据可视化
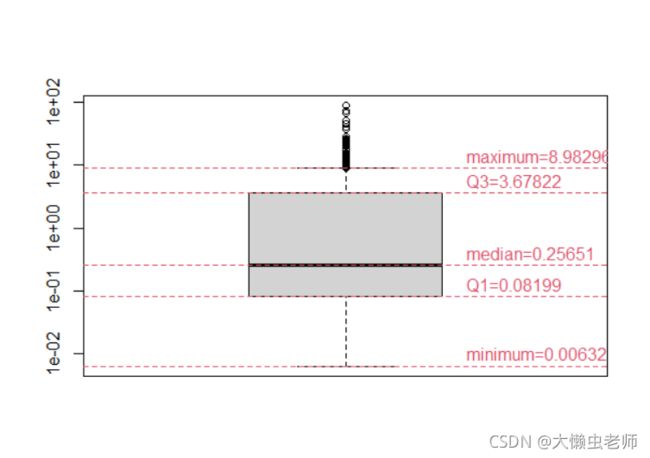
通过查看数据描述,我们知道了每个变量的含义。通过数据可视化,我们可以快速知道数据分布情况,便于下一步构造模型。查看 crim 变量,绘制箱线图。因为数值多分布在0-1范围内,所以在该箱线图中,对y轴的显示取对数,便于更方便地观察数据。
boxplot <- boxplot(Boston$crim,outline = T,log= "y")
boxplot$stats
abline(h=boxplot$stats[1,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[1,], "minimum=0.00632", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[2,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[2,], "Q1=0.08199", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[3,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[3,], "median=0.25651", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[4,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[4,], "Q3=3.67822", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[5,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[5,], "maximum=8.98296", col = 2,adj=c(0,-0.4))
logistic 分类模型
构建分类模型的因变量
构建 logistic 分类模型的因变量,该因变量是二分类的。我们将高于犯罪率(crim)中位数的项记为“1”,否则为“0”。
dt <- Boston# 将 Boston 赋值给 dt
# 构建新变量 crim_bi
# crim_bi:高于 crim 中位数的项记为“1”, 否则为“0”
dt$crim_bi <- ifelse(dt$crim > median(dt$crim), 1, 0)
构建三个不同自变量的模型
#### 构建3个模型 ####
log.fit <- glm(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data = dt , family = "binomial")
summary(log.fit)
log.fit2 <- glm(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt , family = "binomial")
summary(log.fit2)
log.fit3 <- glm(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt , family = "binomial")
summary(log.fit3)
交叉验证
进行交叉验证,将准确率作为衡量标准。
fold_log <- function(log.fit,dt){
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
fold_pre<- predict(log.fit,fold_test,type = "response")
log.class <- ifelse(fold_pre > 0.5, 1, 0)
a <- table(log.class, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
return(mean(accuracy))
}
fold_log(log.fit,dt)
fold_log(log.fit2,dt)
fold_log(log.fit3,dt)
结果分析
> fold_log(log.fit,dt)
[1] 0.9150087
> fold_log(log.fit2,dt)
[1] 0.9229287
> fold_log(log.fit3,dt)
[1] 0.9090433
由输出结果可知,log.fit2 即第二个模型的准确率更高,为 0.9229287 0.9229287 0.9229287。
LDA 回归模型
同理,
lda <- lda(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data = dt)
lda2 <- lda(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt)
lda3 <- lda(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt)
fold_lda <- function(lda,dt){
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
fold_pre<- predict(lda,fold_test)
a <- table(predict(lda,fold_test)$class, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
return(mean(accuracy))
}
fold_lda(lda,dt)
fold_lda(lda2,dt)
fold_lda(lda3,dt)
结果分析
> fold_lda(lda,dt)
[1] 0.8556253
> fold_lda(lda2,dt)
[1] 0.8575861
> fold_lda(lda3,dt)
[1] 0.8635469
由输出结果可知,lda3 即第三个模型的准确率更高,为 0.8635469 0.8635469 0.8635469。
K 临近模型
#### 模型1 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
#### 模型2 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
#### 模型3 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
最优子集构建回归模型
最优子集
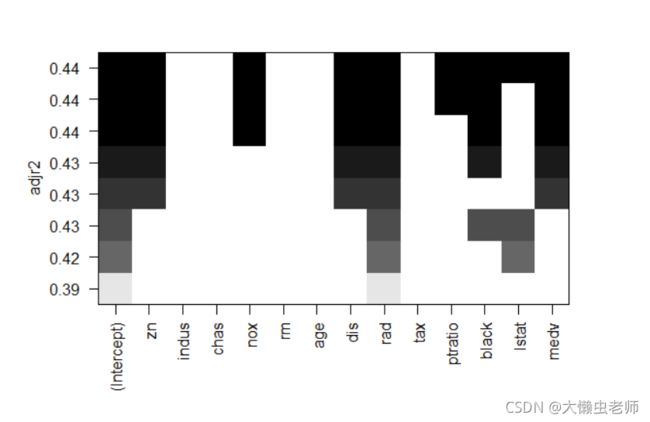
library(leaps)
leaps<- regsubsets(crim ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data=dt)
plot(leaps,scale = "adjr2")
如图所示,截距+rad的调整R平方值为 0.39 0.39 0.39。调整R平方值越高的模型越好,因此最佳预测变量为:
zn+nox+dis+rad+ptratio+black+lstat+medv。
故有:
lmfit<- lm(crim ~ zn+nox+dis+rad+ptratio+black+lstat+medv,
data=dt)
划分学习和测试数据集
随机抽取 70 % 70 \% 70%的数据放入学习数据集,剩余 30 % 30\% 30%放入测试数据集。
dim(dt)
length <- dim(dt)[1]
set.seed(1)
pre <- sample(length,length*0.7)# 随机抽取70 %的观测放入学习数据集
pre <- sort(pre)
train <- dt[pre,]# 学习数据集train
test <- dt[-pre,]# 剩余30 %放入测试数据集test
预测犯罪率
写一个计算均方误差的函数RMSE:
RMSE=function(t,p){
return(sqrt(mean((t-p)^2)))
}
用测试数据集预测犯罪率,并计算均方误差:
lm_pre<- predict(lmfit, test)
RMSE(test$crim,lm_pre)
计算知:
> RMSE(test$crim,lm_pre)
[1] 7.557481
则均方误差为 7.557481 7.557481 7.557481。
代码
rm(list=ls())# 把当前环境中的对象全部删除
library(ggplot2)
library(dplyr)
library(MASS)
head(Boston)# 查看数据前6行
#### 箱线图 ####
boxplot <- boxplot(Boston$crim,outline = T,log= "y")
boxplot$stats
abline(h=boxplot$stats[1,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[1,], "minimum=0.00632", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[2,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[2,], "Q1=0.08199", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[3,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[3,], "median=0.25651", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[4,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[4,], "Q3=3.67822", col = 2,adj=c(0,-0.4))
abline(h=boxplot$stats[5,],lwd=1,col=2,asp = 2,lty = 2)
text(1.25,boxplot$stats[5,], "maximum=8.98296", col = 2,adj=c(0,-0.4))
#### logistic 回归模型 ####
dt <- Boston# 将 Boston 赋值给 dt
# 构建新变量 crim_bi
# crim_bi:高于 crim 中位数的项记为“1”, 否则为“0”
dt$crim_bi <- ifelse(dt$crim > median(dt$crim), 1, 0)
#### 构建3个模型 ####
log.fit <- glm(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data = dt , family = "binomial")
summary(log.fit)
log.fit2 <- glm(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt , family = "binomial")
summary(log.fit2)
log.fit3 <- glm(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt , family = "binomial")
summary(log.fit3)
fold_log <- function(log.fit,dt){
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
fold_pre<- predict(log.fit,fold_test,type = "response")
log.class <- ifelse(fold_pre > 0.5, 1, 0)
a <- table(log.class, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
return(mean(accuracy))
}
fold_log(log.fit,dt)
fold_log(log.fit2,dt)
fold_log(log.fit3,dt)
#### 划分测试和学习数据集 ####
dim(dt)
length <- dim(dt)[1]
set.seed(5)
pre <- sample(length,length*0.8)#随机抽取 80%的观测放入学习数据集
pre <- sort(pre)# 排序
train <- dt[pre,]# 随机抽取 80%的观测放入学习数据集train
test <- dt[-pre,]# 测试数据集test
log.pred <- predict(log.fit2, test, type = "response")
log.class <- ifelse(log.pred > 0.5, 1, 0)
# 混淆矩阵
table(log.class, test$crim_bi)
#### LDA 回归模型 ####
lda <- lda(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data = dt)
lda2 <- lda(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt)
lda3 <- lda(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
data = dt)
fold_lda <- function(lda,dt){
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
fold_pre<- predict(lda,fold_test)
a <- table(predict(lda,fold_test)$class, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
return(mean(accuracy))
}
fold_lda(lda,dt)
fold_lda(lda2,dt)
fold_lda(lda3,dt)
#### K 临近模型 ####
#### 模型1 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
#### 模型2 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+indus+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
#### 模型3 ####
# k=1
library(kknn)
library(caret)
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=1)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
# k=5
set.seed(3)
folds <- createFolds(y=dt[,10],k=10)
accuracy <- as.numeric()
for (i in 1:10){
fold_test <- dt[folds[[i]],] #取folds[[i]]作为测试集
fold_train <- dt[-folds[[i]],] # 剩下的数据作为训练集
knn <- kknn(crim_bi ~ zn+nox+age+dis+rad+tax+ptratio+black+medv,
fold_train,fold_test,k=5)
pre_knn <- fitted(knn)
pre_knn <- ifelse(pre_knn > 0.5, 1, 0)
a <- table(pre_knn, fold_test$crim_bi)
accuracy <- append(accuracy,(a[1]+a[4])/sum(a))
}
mean(accuracy)
library(leaps)
leaps<- regsubsets(crim ~ zn+indus+chas+nox+rm+age+dis+rad+tax+ptratio+black+lstat+medv,
data=dt)
plot(leaps,scale = "adjr2")
lmfit<- lm(crim ~ zn+nox+dis+rad+ptratio+black+lstat+medv,
data=dt)
dim(dt)
length <- dim(dt)[1]
set.seed(1)
pre <- sample(length,length*0.7)# 随机抽取70 %的观测放入学习数据集
pre <- sort(pre)
train <- dt[pre,]# 学习数据集train
test <- dt[-pre,]# 剩余30 %放入测试数据集test
lm_pre<- predict(lmfit, test)
RMSE=function(t,p){
return(sqrt(mean((t-p)^2)))
}
RMSE(test$crim,lm_pre)
哑变量一般指虚拟变量。 虚拟变量 ( Dummy Variables) 又称虚设变量、名义变量或哑变量,用以反映质的属性的一个人工变量,是量化了的自变量,通常取值为0或1。引入哑变量可使线形回归模型变得更复杂,但对问题描述更简明。 ↩︎