遗传算法求解TSP问题
摘要:
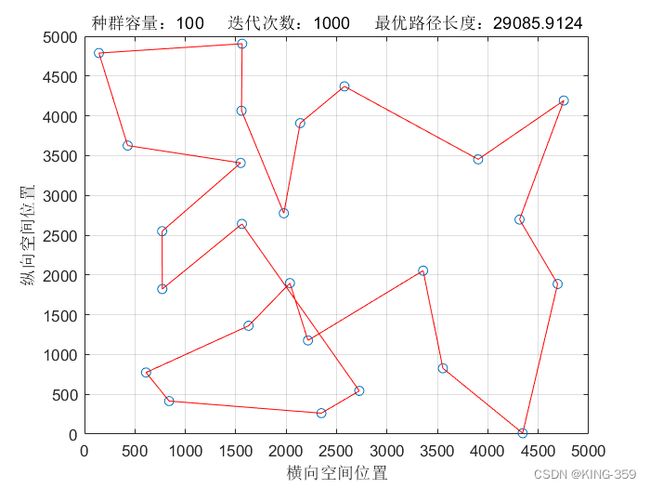
采用遗传算法进行25个城市的TSP问题求解,通过遗传算法求解得出的最短路径值为29085.91,最优路径为24→15→25→20→19→6→8→17→3→13→7→5→11→1→2→14→4→9→12→21→10→16→22→18→23→24(数字为城市序号)。同时根据不同参数下的实验结果,得出结论,随着种群数量的增长及迭代次数的越来越多,遗传算法寻优的结果越来越好。当然,由于遗传算法本身具有一定的随机性,能否快速收敛得看具体参数设定。
1.1 遗传算法原理
遗传算法是一种基于自然选择和群体遗传机理的搜索算法,它模拟了自然选择和自然遗传过程中的繁殖、杂交和突变现象。
在利用遗传算法求解问题时,问题的每一个可能解都被编码成一个“染色体”,即个体,若干个个体构成了群体(所有可能解)。在遗传算法开始时,总是随机的产生一些个体(即初始解),根据预定的目标函数对每一个个体进行评估给出一个适应度值。基于此适应度值,选择一些个体用来产生下一代,选择操作体现了“适者生存"的原理,“好”的个体被用来产生下一代,“坏”的个体则被淘汰,然后选择出来的个体经过交叉和变异算子进行再组合生成新的一代,这一代的个体由于继承了上一代的一些优良性状,因而在性能上要优于上一代,这样逐步朝着最优解的方向进化。
因此,遗传算法可以看成是一个由可行解组成的群体初步进化的过程。
1.2 实验步骤
步骤一:设置 TSP 问题中25个城市的坐标值,限制每个城市只能拜访一次,并且回到出发的城市。本实验中25个城市坐标如下:
[1549,3408; 1561,2641; 3904,3453; 2216,1177; 1563,4906;
3554,827; 2578,4370; 3358,2054; 143,4789; 610,774;
1557,4064; 771,1823; 4753,4192; 2037,1897; 4692,1887;
839,415; 4314,2696; 428,3626; 2725,543; 2349,263;
770,2550; 1627,1361; 2139,3908; 1977,2775; 4345,11];步骤二:设置种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1;
步骤三:初始化种群,计算每个个体的适应度值(即路径长短),执行交叉、变异、选择操作,生成新的种群;
步骤四:判断终止条件(如果满足,则迭代结束。输出最终最短路径值以及绘制最优路径图;否则继续迭代优化,并实时更新最短路径值以及最优路径图)。
源代码如下:
%% 采用遗传算法进行TSP问题求解
%% 清理参数及原始变量
clear al1;
close all;
clc;
%% 设置25个城市坐标
C=[1549,3408;1561,2641;3904,3453;2216,1177;1563,4906;
3554,827;2578,4370;3358,2054;143,4789;610,774;
1557,4064;771,1823;4753,4192;2037,1897;4692,1887;
839,415;4314,2696;428,3626;2725,543;2349,263;
770,2550;1627,1361;2139,3908;1977,2775;4345,11];
%% 设置参数
P=100; % 种群容量
N=size(C,1); % 城市个数
maxIter=1000; % 最大迭代次数
Pm=0.1; % 变异概率
%% 开始寻优
% 初始种群
Chro=[];
for i=1:P
temp=randperm(N);
Chro=[Chro;temp];
end
% 计算种群中每个染色的适应度
fit=fitness(Chro,C);
for iter=1:maxIter
% 交叉
for i=1:P/2
chro1=Chro(i,:); % 交叉配对中的第1个染色体
chro2=Chro(i+P/2,:); % 交叉配对中的第2个染色体
pos=randi(N-2)+1; % 随机选取交叉位置
new_chro1=[chro1(1:pos) chro2(pos+1:end)]; % 新的染色体
new_chro2=[chro2(1:pos) chro1(pos+1:end)]; % 新的染色体
chro1_handled=handle(new_chro1);
chro2_handled=handle(new_chro2);
Chro(i,:)=chro1_handled;
Chro(i+P/2,:)=chro2_handled;
end
% 变异
for i=1:P
if rand()0
chro(i)=0;
end
end
idx=find(chro==0); % 找到删掉的基因所在的位置
for i=1:length(idx) % 逐个替换位未使用的基因
chro(idx(i))=unused(i);
end
end
chro_handled=chro; % 返回值为处理后的染色体
end
%% 计算适应度,即路径的长度
function fit=fitness(Chro,C)
for i=1:size(Chro,1)
possible_sol=Chro(i,:); % 拿到1个染色体
possible_sol=[possible_sol possible_sol(1)]; % 将第一个城市坐标补充到末尾
dist=0; % 用于计算总距离
for j=1:length(possible_sol)-1
city1=possible_sol(j); % 第j个城市的编号
city2=possible_sol(j+1); % 第j+1个城市的编号
city1_C=C(city1,:); % 第j个城市的坐标
city2_C=C(city2,:); % 第j+1个城市的坐标
dist=dist+norm(city1_C-city2_C); % 第j个城市和第j+1个城市的距离
end
fit(i)=dist;
end
end 1.3 实验结果
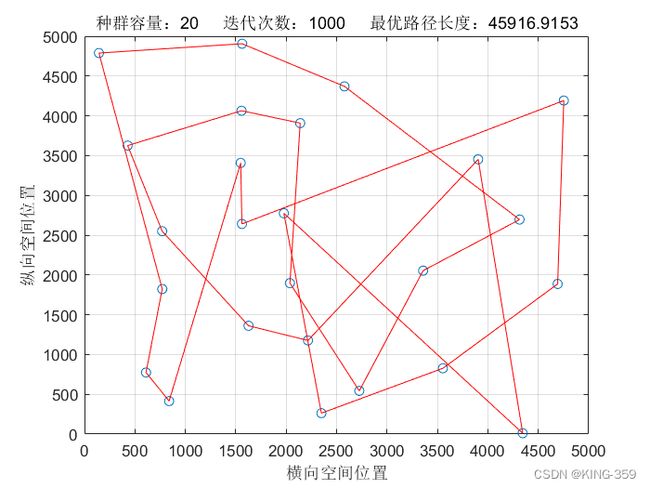
种群数量 P=20,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.1
种群数量 P=40,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.2
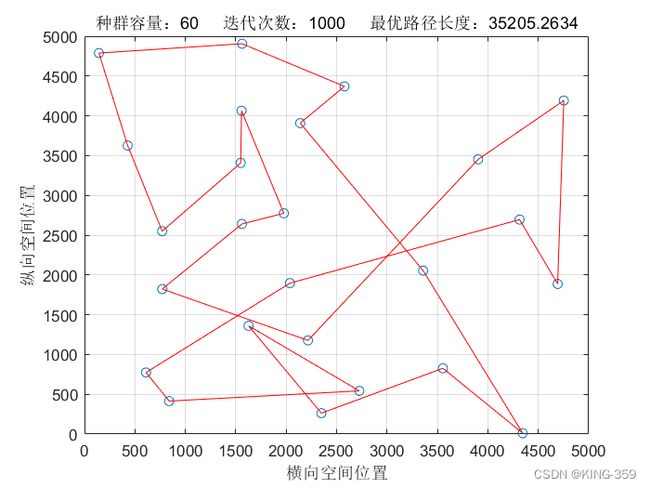
种群数量 P=60,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.3
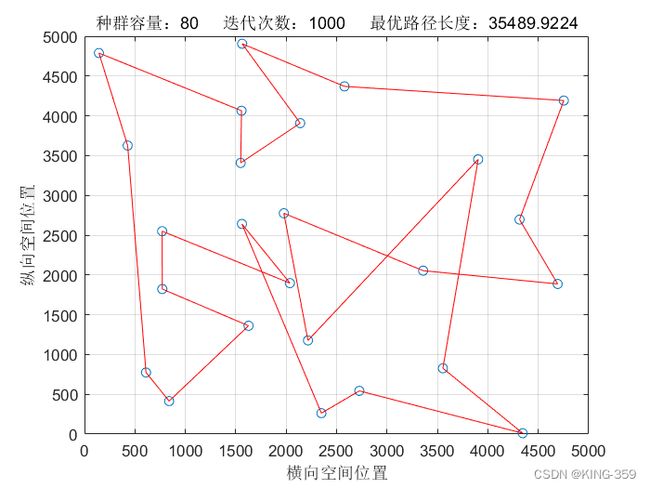
种群数量 P=80,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.4
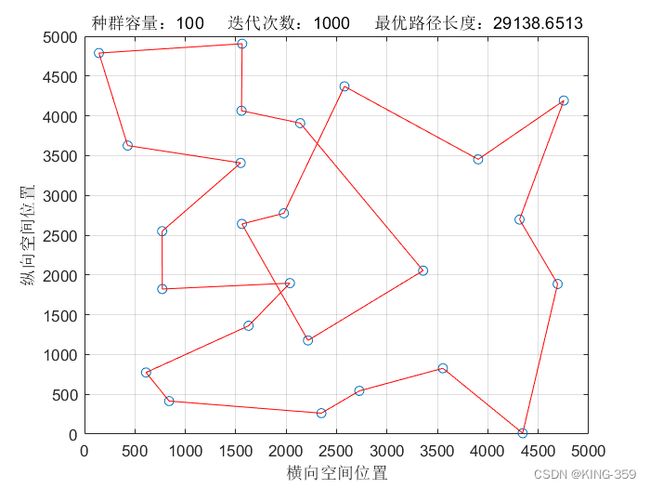
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.5
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=200,变异概率 Pm=0.1时,实验结果如下图所示:
图1.6
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=400,变异概率 Pm=0.1时,实验结果如下图所示:
图1.7
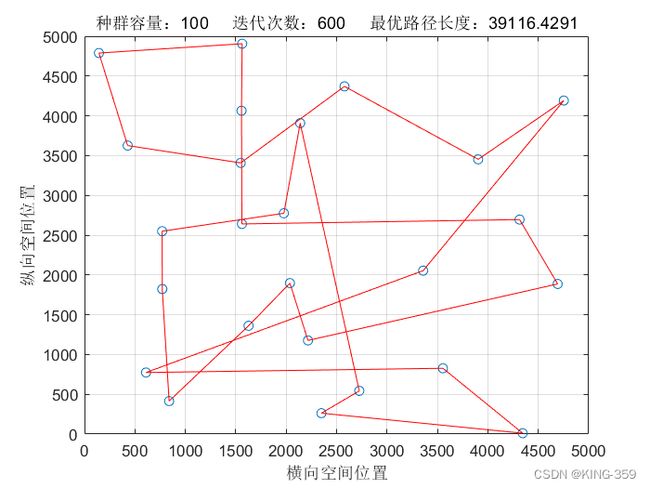
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=600,变异概率 Pm=0.1时,实验结果如下图所示:
图1.8
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=800,变异概率 Pm=0.1时,实验结果如下图所示:
图1.9
种群数量 P=100,染色体基因数 N=25,迭代次数 maxIter=1000,变异概率 Pm=0.1时,实验结果如下图所示:
图1.10
1.4 不同参数下的结果与分析
| 种群数量P |
染色体基因数N |
迭代次数maxIter |
变异概率Pm |
最终结果 |
| 20 |
25 |
1000 |
0.1 |
45916.92 |
| 40 |
25 |
1000 |
0.1 |
39688.31 |
| 60 |
25 |
1000 |
0.1 |
35205.26 |
| 80 |
25 |
1000 |
0.1 |
35489.92 |
| 100 |
25 |
1000 |
0.1 |
29138.65 |
| 100 |
25 |
200 |
0.1 |
42388.17 |
| 100 |
25 |
400 |
0.1 |
41718.27 |
| 100 |
25 |
600 |
0.1 |
39116.43 |
| 100 |
25 |
800 |
0.1 |
35108.83 |
| 100 |
25 |
1000 |
0.1 |
29085.91 |
表1
从表1中不同参数下得到的结果可以看出:针对25个城市的TSP问题,遗传算法求解得出的最短路径值为29085.91,最优路径为24→15→25→20→19→6→8→17→3→13→7→5→11→1→2→14→4→9→12→21→10→16→22→18→23→24(数字为城市序号)。
同时,观察不同参数下得到的结果可以发现,随着种群数量的增长,遗传算法寻优的结果也越来越好;随着迭代次数的越来越多,遗传算法寻优的结果越来越好。当然,由于遗传算法本身具有一定的随机性,能否快速收敛得看具体参数设定。有时候随着种群数量和迭代次数增加,遗传算法寻优的结果可能还不如之前得到结果。