R语言实现决策树和朴素贝叶斯分类预测,并比较准确度(含数据集)
R语言实现决策树和朴素贝叶斯分类预测,并比较准确度(含数据集)
一开始用了《数据科学与大数据分析》(美国EMC教育服务集团)的书上的案例分析是否出去玩,后来发现只有10条训练数据,并且测试数据真值也不知道,故换了鸢尾花数据。
源数据链接:iris.data
提取码: frg4
如果打不开网盘请用这个地址,选iris.data右键保存:http://archive.ics.uci.edu/ml/machine-learning-databases/iris/
(感谢CSDN用户天使健的分享)
注意!!!这个下载后用txt打开也是data文件类型,不要读取错了
加载所需的包
rpart提供了决策树函数,plot绘图,klaR提供了朴素贝叶斯分类函数
library(rpart)
library(rpart.plot)
library(klaR)
读取数据并分集
注意文件路径,文件类型是data不是txt
#导入鸢尾花数据集
setwd("E:/Courses/专业课/大数据分析") #调整默认的文件路径
data <- read.table("iris.data", header = FALSE, sep = ',') #没有sep申明分隔符会导致全存进1个向量
names(data) <- c("Factor1","Factor2","Factor3","Factor4","Class")
print(data)
部分数据展示:

以0.8为划分标准,分出总量*0.8条训练集,其余为测试集。上述150条有120条训练数据,30条测试数据。随机产生。
set.seed(0) #固定随机数,便于检查
train <- sample(nrow(data),0.8*nrow(data)) #随机选择150*0.2=30个数据为测试集,其余为训练集
print(train)
traindata <- data[train,] #将抽样行赋给训练集
testdata <- data[-train,] #将去掉抽样行的剩下的数据赋给测试集
print(traindata)
print(testdata)
n <- length(testdata[,1]) #把测试集数据量赋给n,后面检验准确率用
构建决策树并计算准确率
#构建决策树并预测
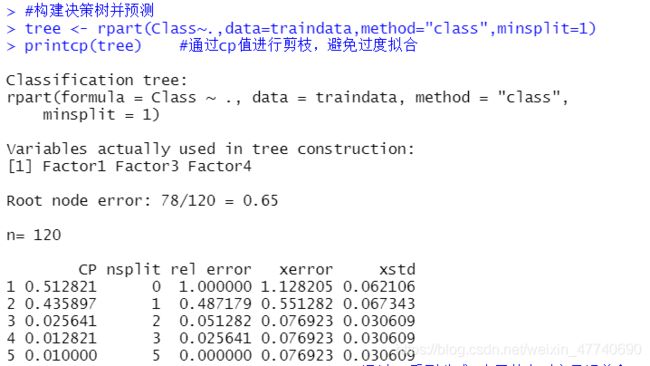
tree <- rpart(Class~.,data=traindata,method="class",minsplit=1)
printcp(tree) #通过cp值进行剪枝,避免过度拟合
tree <- prune(tree, cp=0.025641) #通过cp看到分成3个子节点时交叉误差和4一样
rpart.plot(tree,branch=0,type=4)
关于决策树的算法(ID3,CART)和rpart的各个参数,可以参考这篇blog
画出决策树的图:
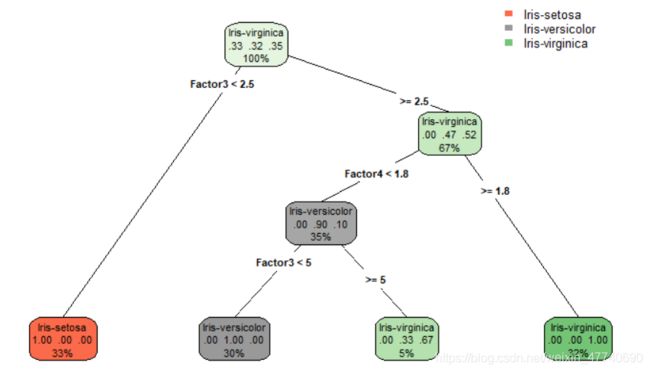
画好决策树后,用之前分出的testdata来预测:
predtree <- predict(tree,newdata=testdata,type="class")
testdata$Tree_Pre_Class <- predtree
a <- testdata$Tree_Pre_Class == testdata$Class
cnt1 <- sum(a[a=TRUE])
treerate <- cnt1/n
print(testdata)
print(a)
print(treerate)
为了便于观察,把预测结果写入testdata最后一列,a用来记录每一条预测的TRUE/FALSE值,cnt1统计了正确的个数,cnt1/n即为准确率。
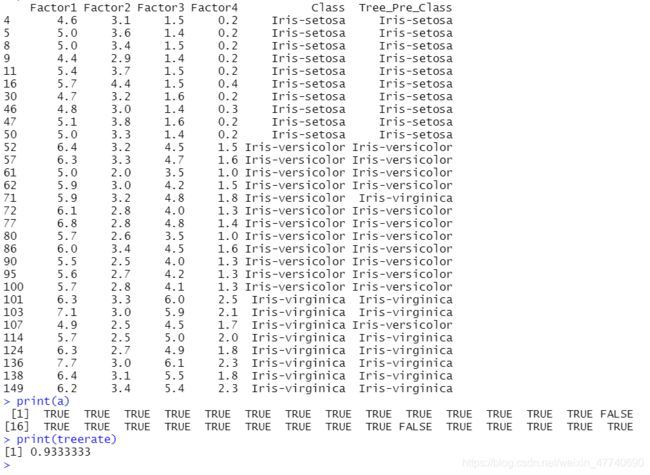
可见有2个预测错误,正确率28/30≈0.9333
朴素贝叶斯预测与准确率
#构建朴素贝叶斯算法并预测
traindata$Class <- as.factor(traindata$Class) #将Class转化为函数可读取的因子,否则会报错
bayes <- NaiveBayes(Class~.,data=traindata)
bayes$apriori #先验概率
bayes$tables #每个变量的条件概率
#plot(bayes)

同样的思路来测试贝叶斯的预测准确度,不再赘述。只需注意相关函数的语法。
predbayes <- predict(bayes,testdata)
testdata$Bayes_Pre_Class = predbayes$class
b <- testdata$Bayes_Pre_Class == testdata$Class
cnt2 <- sum(b[b=TRUE])
bayesrate <- cnt2/n
print(testdata)
print(b)
print(bayesrate)
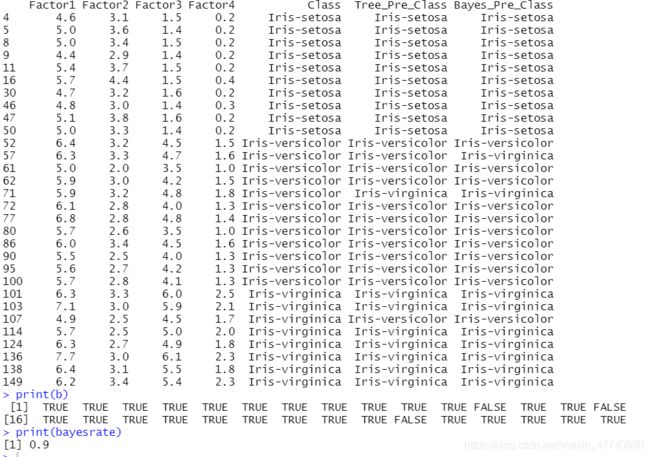
可以看出,在这个例子中贝叶斯有3个预测错误,准确度27/30=0.9,其准确度不如决策树模型。
完整代码如下
library(rpart)
library(rpart.plot)
library(klaR)
#导入鸢尾花数据集
setwd("E:/Courses/专业课/大数据分析") #调整默认的文件路径
data <- read.table("iris.data", header = FALSE, sep = ',')
names(data) <- c("Factor1","Factor2","Factor3","Factor4","Class")
print(data)
set.seed(0) #固定随机数,便于检查
train <- sample(nrow(data),0.8*nrow(data)) #随机选择150*0.2=30个数据为测试集,其余为训练集
print(train)
traindata <- data[train,] #将抽样行赋给训练集
testdata <- data[-train,] #将去掉抽样行的剩下的数据赋给测试集
print(traindata)
print(testdata)
n <- length(testdata[,1]) #把测试集数据量赋给n,后面检验准确率用
#构建决策树并预测
tree <- rpart(Class~.,data=traindata,method="class",minsplit=1)
printcp(tree) #通过cp值进行剪枝,避免过度拟合
tree <- prune(tree, cp=0.025641) #通过cp看到分成3个子节点时交叉误差和4一样
rpart.plot(tree,branch=0,type=4)
predtree <- predict(tree,newdata=testdata,type="class")
testdata$Tree_Pre_Class <- predtree
a <- testdata$Tree_Pre_Class == testdata$Class
cnt1 <- sum(a[a=TRUE])
treerate <- cnt1/n
print(testdata)
print(a)
print(treerate)
#构建朴素贝叶斯算法并预测
traindata$Class <- as.factor(traindata$Class) #将Class转化为函数可读取的因子,否则会报错
bayes <- NaiveBayes(Class~.,data=traindata)
bayes$apriori #先验概率
bayes$tables #每个变量的条件概率
#plot(bayes)
predbayes <- predict(bayes,testdata)
testdata$Bayes_Pre_Class = predbayes$class
b <- testdata$Bayes_Pre_Class == testdata$Class
cnt2 <- sum(b[b=TRUE])
bayesrate <- cnt2/n
print(testdata)
print(b)
print(bayesrate)