T-SNE可视化高维数据,亮瞎审稿人
文章目录
-
- 经典案例-MNIST手写数字降维可视化
- 论文中使用 t-SNE 案例
- t-SNE 实战
-
- MNIST 可视化教程
- MRI 脑肿瘤三维数据可视化
t-Distributed Stochastic Neighbor Embedding (t-分布随机邻域嵌入, 简称 t-SNE) 是一种降维技术,特别适用于 高维数据集的可视化
经典案例-MNIST手写数字降维可视化
MNIST 原始数据大小: 60000 * 784,每个数据 784 维
2D-t-SNE后为: 60000 x 2
3D-t-SNE后为: 60000 x 3
可见,把 784 维数据(图像大小 28x28,拉直后为784,对 MNIST 不了解请百度)降成 2 维或 3 维是很大程度上的压缩。降维后的结果如图所示。
3D-t-SNE
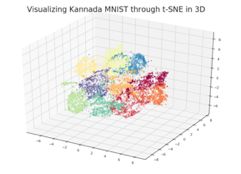
上边是2D-t-SNE,数字0-9一共是10个类,每个类分别是不同的颜色,通过二维图像,我们很容易看到每个类别的分布差异性。
论文中使用 t-SNE 案例
Paper: GANomaly: Semi-Supervised Anomaly Detection via Adversarial Training
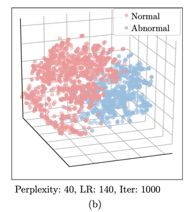
把正常数据和异常数据分开用二维展示,可以明显看出两个类别之间的分布是有界限的。
Paper: MAMA Net: Multi-scale Attention Memory Autoencoder Network for Anomaly Detection
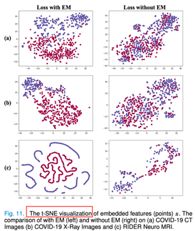
使用 t-SNE 可视化模型的潜在空间(比如 U-NET 模型的瓶颈层),以比较使用EM和不使用 EM loss 对潜在空间的影响。
Paper: Unsupervised Detection of Lesions in Brain MRI using constrain-ed adversarial auto-encoders
使用 t-SNE 可视化健康图像和异常图像之间的分布差异性,通过图像可以看出,二者的分布差异较小。
从上面这些例子可以直观看出 t-SNE 在可视化方面用的非常多。在做分类任务之前,我们也可以用它看看不同类别之间有没有明显的分界线。如果分界线明显,可以说明分类任务比较简单。反之则说明类别之间不容易区分,如果我们的分类结果不是很好,可以用此图说明任务的难度。如果分类结果很好,也可以更加展示算法的优势。
当然,这只是例举一个用处,它的作用不止这些。
t-SNE 实战
它的作用了解了,看看怎么用代码实现它。经过多次实践发现,虽然实现他的方法很多,但是最好用最方便的还是使用 sklearn.manifold.TSNE
接下来讲解2个使用该方法的案例
MNIST 可视化教程
kaggle MNIST 可视化教程 上面讲的很详细,还对比 PCA 和 t-SNE 的区别
MRI 脑肿瘤三维数据可视化
实验背景与目的: brats 是三维脑 MRI 肿瘤数据,大小= 240x240x150, 把每个数据中的肿瘤层面看成是异常层,不含肿瘤的层面看成是正常层。使用 t-SNE 可视化,以观察正常层和异常层在分布上是否有差异。
这里的层指的是 axial-slice, 即横断面。一个三维数据有150层,每层的大小都等于240x240
- 1.导入包
import numpy as np
import matplotlib.pyplot as plt
from sklearn import manifold
from glob import glob
import nibabel as nib
- 2.加载数据
我的数据是三维的,格式为 .nii.gz
# load data
brats = sorted(glob('BraTs/*.gz'))
brats_gt = sorted(glob('BraTsseg/*.gz'))
brats_tra = []
brats_label = []
for i, j in zip(brats, brats_gt):
brats_np = nib.load(i).get_fdata()
brats_gt_np = nib.load(j).get_fdata()
assert brats_gt_np.shape == brats_np.shape
z = brats_np.shape[-1]
for zi in range(z):
brats_tra.append(brats_np[..., zi].flatten()) # 横断层拉直,维度=240x240=57600
if brats_gt_np[..., zi].any():
brats_label.append(1)
else:
brats_label.append(0)
brats_array = np.array(brats_tra, dtype='uint8') # [6200, 57600]
brats_label_array = np.array(brats_label, dtype='uint8') # [6200]
brats_array 大小为[6200, 57600], 表示一共有6200层,每层的数据维度是57600,对它进行降维到[6200, 2]
- 3.可视化-使用sklearn-TSNE
tsne = manifold.TSNE(n_components=2, init='pca', random_state=42).fit_transform(brats_array)
tsne shape [6200, 2], 由于数据维度非常大,还要迭代1000次,因此非常慢。
参数介绍
- n_components(默认值:2):嵌入空间的维度,需要降到几维写几维。
- init: 初始化方法,多采用 PCA 初始化
- perplexity(默认值:30):perplexity 与其他流形学习算法中使用的最近邻的数量有关。考虑选择 5 到 50 之间的值。
- n_iter(默认值:1000):优化的最大迭代次数。应至少为 250。
- random_state: 随机种子
- 还有其他参数可以调整。有关详细信息,请参阅文档
- t-SNE 归一化
# tsne 归一化, 这一步可做可不做
x_min, x_max = tsne.min(0), tsne.max(0)
tsne_norm = (tsne - x_min) / (x_max - x_min)
- 根据label,把正常层和异常层分开
normal_idxs = (brats_label_array == 0)
abnorm_idxs = (brats_label_array == 1)
tsne_normal = tsne_norm[normal_idxs]
tsne_abnormal = tsne_norm[abnorm_idxs]
- 使用matplotlib画出t-SNE
plt.figure(figsize=(8, 8))
plt.scatter(tsne_normal[:, 0], tsne_normal[:, 1], 1, color='red', label='Healthy slices')
# tsne_normal[i, 0]为横坐标,X_norm[i, 1]为纵坐标,1为散点图的面积, color给每个类别设定颜色
plt.scatter(tsne_abnormal[:, 0], tsne_abnormal[:, 1], 1, color='green', label='Anomalous slices')
plt.legend(loc='upper left')
plt.show()
结果展示:
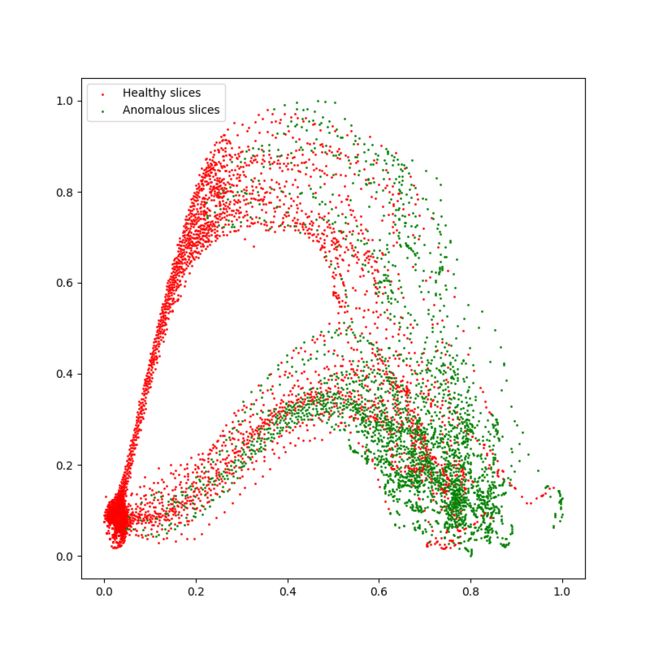
由于进行了归一化,横纵坐标值都在[0,1] 从这个图可以看出,正常层分布在左边,异常在右边,当然也有很多分界不清的区域。
总结: 使用 sklearn.manifold.TSNE 很简单,就一句话搞定了,其余的代码都是为了创建需要降维的数组,以及使用 matplotlib进行展示,真正核心代码就一行。
想了解更多理论知识,请阅读以下链接
TSNE理论知识
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连
![]()