逻辑回归与scikit-learn
欢迎关注本人的微信公众号AI_Engine
LogisticRegression
算法原理
- 一句话概括:逻辑回归假设数据服从伯努利分布,通过极大化似然函数(损失函数)的方法,运用梯度下降或其他优化算法来求解参数,来达到将数据二分类的目的。
- 定义:逻辑回归(Logistic Regression)是一种用于解决二分类(0 or 1)问题的机器学习方法,用于估计某种事物的可能性(不是概率)。比如某用户购买某商品的可能性,某病人患有某种疾病的可能性,以及某广告被用户点击的可能性等。 注意,这里用的是“可能性”,而非数学上的“概率”,logisitc回归的结果并非数学定义中的概率值,不可以直接当做概率值来用。该结果往往用于和其他特征值加权求和,而非直接相乘。
- 区别:逻辑回归(Logistic Regression)与线性回归(Linear Regression)都是一种广义线性模型(generalized linear model)。逻辑回归假设因变量y服从伯努利分布,而线性回归假设因变量y服从高斯分布。因此与线性回归有很多相同之处,如果去除sigmoid映射函数的话,逻辑回归算法就是一个线性回归。可以说,逻辑回归是以线性回归为理论支持的,但是逻辑回归通过sigmoid函数引入了非线性因素,因此可以轻松处理二分类问题。
- 函数:sigmoid函数是逻辑回归中的假设函数,正是因为sigmoid函数与线性回归的结合才使得逻辑回归具有二分类的作用。sigmoid表达式与曲线如下所示:
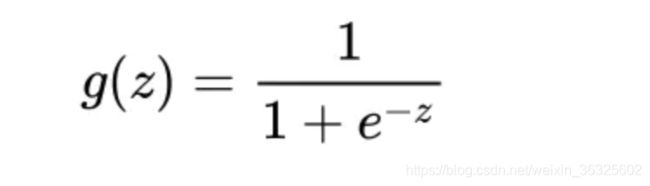
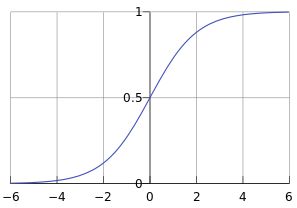
- 决策边界:阈值的方程。当阈值为0.5时,对于sigmoid函数来说z=0就是该模型的决策边界。
- 损失函数:在逻辑回归中,最常用的是代价函数是交叉熵(Cross Entropy)
- 特征离散化:工业界很少直接将连续值作为LR模型的特征输入,而是将连续特征值离散化为一系列的0、1特征交给LR模型。其优势如下:
- 离散特征的增加和减少都很容易,易于模型的快速迭代;
- 稀疏向量内积乘法运算速度快,计算结果方便存储,容易扩展;
- 离散化后的特征对异常数据有很强的鲁棒性:比如一个特征是年龄>30是1,否则0。如果特征没有离散化,一个异常数据“年龄300岁”会给模型造成很大的干扰;
- 逻辑回归属于广义线性模型,表达能力受限;单变量离散化为N个后,每个变量有单独的权重,相当于为模型引入了非线性,能够提升模型表达能力,加大拟合;
- 离散化后可以进行特征交叉,由M+N个变量变为M * N个变量,进一步引入非线性,提升表达能力;
- 特征离散化后,模型会更稳定,比如如果对用户年龄离散化,20-30作为一个区间,不会因为一个用户年龄长了一岁就变成一个完全不同的人。当然处于区间相邻处的样本会刚好相反,所以怎么划分区间是门学问;
- 特征离散化以后,起到了简化了逻辑回归模型的作用,降低了模型过拟合的风险。
实例
def build_train_model(x, y):
length_sample = len(x)
simple_weight = np.ones(shape=(length_sample,))
# simple_weight[:length_sample/2] = 2
model = LogisticRegression(penalty='l2', C=1.0, solver='sag', max_iter=3000, multi_class='ovr')
model.fit(x, y, simple_weight)
return model
def cross_val(x, y):
skf = StratifiedKFold(n_splits=5, shuffle=True, random_state=Config.seed)
model = LogisticRegression(penalty='l2', C=1.0, solver='sag')
result = cross_val_score(model, x, y, cv=skf)
print(result)
def report_evaluation_metrics(y_true, y_pred):
accuracy = accuracy_score(y_true=y_true, y_pred=y_pred)
precision = precision_score(y_true=y_true, y_pred=y_pred, labels=[0, 1], pos_label=1)
recall = recall_score(y_true=y_true, y_pred=y_pred, labels=[0, 1], pos_label=1)
average_precision = average_precision_score(y_true=y_true, y_score=y_pred)
f1 = f1_score(y_true=y_true, y_pred=y_pred, labels=[0, 1], pos_label=1)
conf_matrix = confusion_matrix(y_true, y_pred)
print('Accuracy: {0:0.2f}'.format(accuracy))
print('Precision: {0:0.2f}'.format(precision))
print('Recall: {0:0.2f}'.format(recall))
print('Average precision-recall score: {0:0.2f}'.format(average_precision))
print('F1: {0:0.2f}'.format(f1))
print("confusion matrix:", conf_matrix)
参数
- penalty:正则项,str类型,用于指定惩罚项中使用的规范。可选参数为L1和L2,默认为L2。L1规范假设的是模型的参数满足拉普拉斯分布,L2假设的模型参数满足高斯分布。所谓的范式就是加上对参数的约束,使得模型更不会过拟合(overfit),理论上可以提高模型的泛化能力。在调参时如果我们主要的目的只是为了解决过拟合,一般penalty选择L2正则化就够了。但是如果选择L2正则化发现还是过拟合,即预测效果差的时候,就可以考虑L1正则化。另外,如果模型的特征非常多,我们希望一些不重要的特征系数归零,从而让模型系数稀疏化的话,也可以使用L1正则化。如果penalty是L1正则化的话,就只能选择liblinear了。这是因为L1正则化的损失函数不是连续可导的,而newton-cg,lbfgs,sag这三种优化算法时都需要损失函数的一阶或者二阶连续导数。而liblinear并没有这个依赖。
- c:正则化系数λ的倒数,也就是说数值越小表示正则化程度越强。float类型,默认为1.0,且必须是正浮点型数。
- solver:优化算法选择参数,只有五个可选参数。newton-cg,lbfgs,liblinear,sag,saga,默认为liblinear。solver参数决定了我们对逻辑回归损失函数的优化方法.有5种算法可以选择,分别是:
1. liblinear:使用了开源的liblinear库实现,内部使用了坐标轴下降法来迭代优化损失函数。
2. lbfgs:拟牛顿法的一种,利用损失函数二阶导数矩阵即海森矩阵来迭代优化损失函数。
3. newton-cg:也是牛顿法家族的一种,利用损失函数二阶导数矩阵即海森矩阵来迭代优化损失函数。
4. sag:即随机平均梯度下降,是梯度下降法的变种,和普通梯度下降法的区别是每次迭代仅仅用一部分的样本来计算梯度,适合于样本数据多的时候。
5. saga:线性收敛的随机优化算法的的变种。
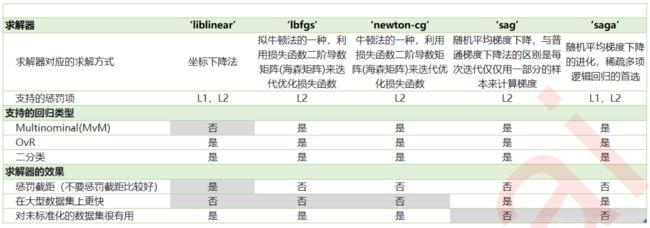
总结:
- liblinear适用于小数据集,而sag和saga适用于大数据集因为速度更快,支持二分类,L1,L2。
- 对于多分类问题,只有newton-cg,sag,saga和lbfgs能够处理多项损失,而liblinear得先把一种类别作为一个类别,剩余的所有类别作为另外一个类别。依次遍历所有类别进行分类。
- newton-cg、sag、lbfgs这三种优化算法时都需要损失函数的一阶或者二阶连续导数,因此不能用于没有连续导数的L1正则化,只能用于L2正则化。而liblinear和saga通吃L1正则化和L2正则化。
- sag每次仅仅使用了部分样本进行梯度迭代,所以当样本量少的时候不要选择它,而如果样本量非常大,比如大于10万,sag是第一选择。但是sag不能用于L1正则化,所以当你有大量的样本,又需要L1正则化的话就要自己做取舍了。要么通过对样本采样来降低样本量,要么回到L2正则化。
从上面的描述,大家可能觉得,既然newton-cg, lbfgs和sag这么多限制,如果不是大样本,我们选择liblinear不就行了嘛!错,因为liblinear也有自己的弱点!我们知道,逻辑回归有二元逻辑回归和多元逻辑回归。对于多元逻辑回归常见的有one-vs-rest(OvR)和many-vs-many(MvM)两种。而MvM一般比OvR分类相对准确一些。郁闷的是liblinear只支持OvR,不支持MvM,这样如果我们需要相对精确的多元逻辑回归时,就不能选择liblinear了。也意味着如果我们需要相对精确的多元逻辑回归不能使用L1正则化了
- class_weight参数用于标示分类模型中各种类型的权重,可以不输入,即不考虑权重,或者说所有类型的权重一样。如果选择输入的话,可以选择balanced让类库自己计算类型权重,或者我们自己输入各个类型的权重,比如对于0,1的二元模型,我们可以定义class_weight={0:0.9, 1:0.1},这样类型0的权重为90%,而类型1的权重为10%。如果class_weight选择balanced,那么类库会根据训练样本量来计算权重。某种类型样本量越多,则权重越低,样本量越少,则权重越高。那么class_weight有什么作用呢?在分类模型中,我们经常会遇到两类问题:第一种是误分类的代价很高。比如对合法用户和非法用户进行分类,将非法用户分类为合法用户的代价很高,我们宁愿将合法用户分类为非法用户,这时可以人工再甄别,但是却不愿将非法用户分类为合法用户。这时,我们可以适当提高非法用户的权重。第二种是样本是高度失衡的,比如我们有合法用户和非法用户的二元样本数据10000条,里面合法用户有9995条,非法用户只有5条,如果我们不考虑权重,则我们可以将所有的测试集都预测为合法用户,这样预测准确率理论上有99.95%,但是却没有任何意义。这时,我们可以选择balanced,让类库自动提高非法用户样本的权重。提高了某种分类的权重,相比不考虑权重,会有更多的样本分类划分到高权重的类别,从而可以解决上面两类问题。当然,对于第二种样本失衡的情况,我们还可以考虑用下一节讲到的样本权重参数:sample_weight,不使用class_weight。
- sample_weight:在调用fit函数时来自己调节每个样本权重。
- max_iter:算法收敛最大迭代次数。int类型,默认为10。仅在正则化优化算法为newton-cg, sag和lbfgs才有用,算法收敛的最大迭代次数。
- multi_class:分类方式选择参数,str类型,可选参数为ovr(二分类)和multinomial(多分类),默认为ovr。ovr即前面提到的one-vs-rest(OvR),而multinomial即前面提到的many-vs-many(MvM)。如果是二元逻辑回归,ovr和multinomial并没有任何区别,区别主要在多元逻辑回归上。
OvR和MvM的不同: OvR的思想很简单,无论你是多少元逻辑回归,我们都可以看做二元逻辑回归。具体做法是,对于第K类的分类决策,我们把所有第K类的样本作为正例,除了第K类样本以外的所有样本都作为负例。然后在上面做二元逻辑回归,得到第K类的分类模型,其他类的分类模型获得以此类推。而MvM则相对复杂,这里举MvM的特例one-vs-one(OvO)作讲解。如果模型有T类,我们每次在所有的T类样本里面选择两类样本出来,不妨记为T1类和T2类,把所有的输出为T1和T2的样本放在一起,把T1作为正例,T2作为负例,进行二元逻辑回归,得到模型参数。我们一共需要T(T-1)/2次分类。可以看出OvR相对简单,但分类效果相对略差(这里指大多数样本分布情况,某些样本分布下OvR可能更好)。而MvM分类相对精确,但是分类速度没有OvR快。如果选择了ovr,则4种损失函数的优化方法liblinear,newton-cg,lbfgs和sag都可以选择。但是如果选择了multinomial,则只能选择newton-cg, lbfgs和sag了。
面试总结
- 1.逻辑回归的求解
由于该极大似然函数无法直接求解,我们一般通过对该函数进行梯度下降来不断逼急最优解。梯度下降分为随机梯度下降,批梯度下降,small batch梯度下降三种方式,批梯度下降会获得全局最优解,缺点是在更新每个参数的时候需要遍历所有的数据,计算量会很大,并且会有很多的冗余计算,导致的结果是当数据量大的时候,每个参数的更新都会很慢。随机梯度下降是以高方差频繁更新,优点是使得sgd会跳到新的和潜在更好的局部最优解,缺点是使得收敛到局部最优解的过程更加的复杂。小批量梯度下降结合了sgd和batch gd的优点,每次更新的时候使用n个样本。减少了参数更新的次数,可以达到更加稳定收敛结果,一般在深度学习当中我们采用这种方法。另外,对于学习率问题中的Adam,动量法等优化方法是衍生的考点。第一个考点是如何对模型选择合适的学习率,因为保持同样的学习率其实不太合适。一开始参数刚刚开始学习的时候,此时的参数和最优解隔的比较远,需要保持一个较大的学习率尽快逼近最优解。但是学习到后面的时候,参数和最优解已经隔的比较近了,若保持最初的大学习率,容易越过最优点,在最优点附近来回振荡。第二个考点是如何对参数选择合适的学习率。在实践中,对每个参数都保持的同样的学习率也是很不合理的。有些参数更新频繁,那么学习率可以适当小一点。有些参数更新缓慢,那么学习率就应该大一点。
- 2.逻辑回归在训练的过程当中,如果有很多的特征高度相关或者说有一个特征重复了100遍,会造成怎样的影响?
对特征本身来说的话,假设只有一个特征,在不考虑采样的情况下,你现在将它重复100遍。训练时这个特征重复使用了100遍,实质上是将原来的特征分成了100份,每一个特征都是原来特征权重值的百分之一。如果在随机采样的情况下,其实训练收敛完以后,还是可以认为这100个特征和原来那一个特征扮演的效果一样,只是可能中间很多特征的值正负相消了。所以,如果在损失函数最终收敛的情况下,其实就算有很多特征高度相关也不会影响分类器的效果。
- 3.为什么我们还是会在训练的过程当中将高度相关的特征去掉?
去掉高度相关的特征会让模型的可解释性更好,且提高训练的速度。如果模型当中有很多特征高度相关的话,就算损失函数本身收敛了,但实际上参数是没有收敛的,这样会拉低训练的速度。其次是特征多了,本身也会增大训练的时间。
L1、L2理解
正则化是用来防止模型过拟合的过程,常用的有L1正则化和L2正则化两种选项,分别通过在损失函数后加上参数向量的L1范式和L2范式的倍数来实现。其中L1范数表现为参数向量中的每个参数的绝对值之和,L2范数表现为参数向量中的每个参数的平方和的开方值。
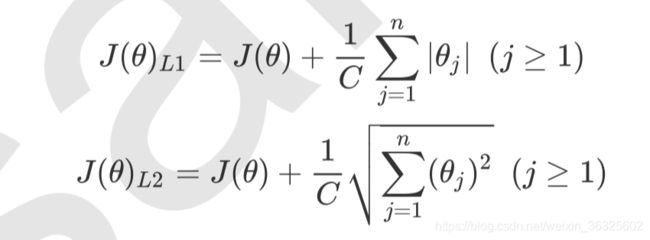
其中 是我们之前提过的损失函数,C是用来控制正则化程度的超参数,n是方程中特征的总数,也是方程中参数的总数,j代表每个参数。在这里,J要大于等于1,是因为我们的参数向量中第一个参数是 我们的截距,它通常是不参与正则化的。
| 参数 | 说明 |
|---|---|
| penalty | 可以输入"l1"或"l2"来指定使用哪一种正则化方式,不填写默认"l2"。 注意,若选择"l1"正则化,参数solver仅能够使用”liblinear",若使用“l2”正则化,参数solver中 所有的求解方式都可以使用。 |
| C | C正则化强度的倒数,必须是一个大于0的浮点数,不填写默认1.0,即默认一倍正则项。 C越小,对损失函数的惩罚越重,正则化的效力越强,参数会逐渐被压缩得越来越小。在很多书籍和博客的原理讲解中, 1/c被写作λ。 |
L1正则化和L2正则化虽然都可以控制过拟合,但它们的效果并不相同。当正则化强度逐渐增大(即C逐渐变小), 参数 的取值会逐渐变小,但L1正则化会将参数压缩为0,L2正则化只会让参数尽量小,不会取到0。
-
在L1正则化在逐渐加强的过程中,携带信息量小的、对模型贡献不大的特征的参数,会比携带大量信息的、对模型有巨大贡献的特征的参数更快地变成0,所以L1正则化本质是一个特征选择的过程,掌管了参数的“稀疏性”。L1正则化越强,参数向量中就越多的参数为0,参数就越稀疏,选出来的特征就越少,以此来防止过拟合。因此,如果特征量很大,数据维度很高,我们会倾向于使用L1正则化。由于L1正则化的这个性质,逻辑回归的特征选择可以由Embedded嵌入法来完成。
-
L2正则化在加强的过程中,会尽量让每个特征对模型都有一些小的贡献,但携带信息少,对模型贡献不大的特征的参数会非常接近于0。通常来说,如果我们的主要目的只是为了防止过拟合,选择L2正则化就足够了。但是如果选择L2正则化后还是过拟合,模型在未知数据集上的效果表现很差,就可以考虑L1正则化。
重要属性
coef_ :逻辑回归的重要属性coef_,查看每个特征所对应的参数。
import matplotlib.pyplot as plt
from sklearn.metrics import accuracy_score
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
data = load_breast_cancer()
x = data.data
y = data.target
data.data.shape
(569, 30)
from sklearn.linear_model import LogisticRegression
l1_lr = LogisticRegression(penalty='l1', solver='liblinear', C=0.5, max_iter=1000)
l1_lr.fit(x, y)
l1_lr.coef_
array([[ 4.01278447, 0.03213562, -0.13907204, -0.01621722, 0. ,
0. , 0. , 0. , 0. , 0. ,
0. , 0.50554115, 0. , -0.0712659 , 0. ,
0. , 0. , 0. , 0. , 0. ,
0. , -0.24609249, -0.12851748, -0.01441286, 0. ,
0. , -2.02967981, 0. , 0. , 0. ]])
n_iter_:损失函数梯度下降时迭代的最优次数
LR特征选择
由于L1正则化会使得部分特征对应的参数为0,因此L1正则化可以用来做特征选择,结合嵌入法的模块SelectFromModel,我们可以很容易就筛选出让模型十分高效的特征。注意,此时我们的目的是尽量保留原数据上的信息,让模型在降维后的数据上的拟合效果保持优秀,因此我们不考虑训练集测试集的问题,把所有的数据都放入模型进行降维。
fullx = []
fsx = []
threshold = np.linspace(0,abs((LR_.fit(data.data,data.target).coef_)).max(),20)
k=0
for i in threshold:
X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(data.data,data.target) fullx.append(cross_val_score(LR_,data.data,data.target,cv=5).mean()) fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=5).mean()) print((threshold[k],X_embedded.shape[1]))
k+=1
plt.figure(figsize=(20,5)) plt.plot(threshold,fullx,label="full") plt.plot(threshold,fsx,label="feature selection") plt.xticks(threshold) plt.legend() plt.show()
为了让模型的拟合效果更好,在这里我们有两种调整方式:
1.调节SelectFromModel这个类中的参数threshold,这是嵌入法的阈值,表示删除所有参数的绝对值低于这个阈值的特征。现在threshold默认为None,所以SelectFromModel只根据L1正则化的结果来选择了特征,即选择了所有L1正则化后参数不为0的特征。我们此时,只要调整threshold的值(画出threshold的学习曲线),就可以观察不同的threshold下模型的效果如何变化。一旦调整threshold,就不是在使用L1正则化选择特征,而是使用模型的属性.coef_中生成的各个特征的系数来选择。coef_虽然返回的是特征的系数,但是系数的大小和决策树中的feature_ importances_以及降维算法中的可解释性方差explained_vairance_概念相似,其实都是衡量特征的重要程度和贡献度的,因此SelectFromModel中的参数threshold可以设置为coef_的阈值,即可以剔除系数小于threshold中输入的数字的所有特征。然而,这种方法其实是比较无效的,大家可以用学习曲线来跑一跑:当threshold越来越大,被删除的特征越来越多,模型的效果也越来越差,模型效果最好的情况下需要保证有17个以上的特征。实际上我画了细化的学习曲线,如果要保证模型的效果比降维前更好,我们需要保留25个特征,这对于现实情况来说,是一种无效的降维:需要30个指标来判断病情,和需要25个指标来判断病情,对医生来说区别不大。
2.第二种调整方法,是调逻辑回归的类LR,通过画C的学习曲线来实现:
(1)求得c值
def feature_select(x, y):
all_features = []
slt_features = []
c = np.arange(4.0, 5.5, 0.1)
for i in c:
lr = LogisticRegression(solver='liblinear', C=i, random_state=666)
all_features.append(cross_val_score(lr, x, y, cv=10).mean())
x_embedded = SelectFromModel(estimator=lr, norm_order=1).fit_transform(x, y)
slt_features.append(cross_val_score(lr, x_embedded, y, cv=10).mean())
c_value = c[slt_features.index(max(slt_features))]
print(max(slt_features), c[slt_features.index(max(slt_features))])
plt.figure(figsize=(20, 5))
plt.plot(c, all_features, label="all features")
plt.plot(c, slt_features, label="selected features")
plt.xticks(c)
plt.legend()
plt.show()
return c_value
def check_reslut(x, y, c_value):
lr = LogisticRegression(solver='liblinear', C=c_value, random_state=666)
score1 = cross_val_score(lr, x, y, cv=10).mean()
x_embedded = SelectFromModel(estimator=lr, norm_order=1).fit_transform(x, y)
score2 = cross_val_score(lr, x_embedded, y, cv=10).mean()
print(score1, score2)
0.9490849969751964 0.9545004753262466
