常见聚类算法及使用--层次聚类(Agglomerative clustering)
文章目录
-
-
- 前言
- 层次聚类的实现
-
- 聚类过程
- 代码实现
- 参考文献
-
前言
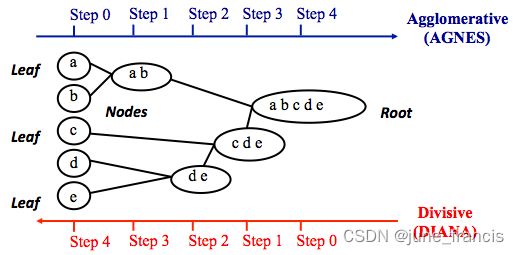
层次聚类顾名思义就是按照某个层次对样本集进行聚类操作,这里的层次实际上指的就是某种距离定义。
层次聚类最终的目的是消减类别的数量,所以在行为上类似于树状图由叶节点逐步向根节点靠近的过程,这种行为过程又被称为“自底向上”。
更通俗的,层次聚类是将初始化的多个类簇看做树节点,每一步迭代,都是将两两相近的类簇合并成一个新的大类簇,如此反复,直至最终只剩一个类簇(根节点)。
与层次聚类相反的是分裂聚类(divisive clustering),又名 DIANA(Divise Analysis),它的行为过程为“自顶向下”。
本文重点为大家介绍层次聚类。
层次聚类的实现
聚类过程
- 数据准备;
- 计算数据集中各样本之间的距离(相似度信息);
- 使用 连接函数(
linkage function)将样本进行分组形成层次聚类数(分组依据是上一步计算出来的距离信息),距离相近的样本会被链接在一起; - 决定在什么地方将层次聚类树截断成多个聚类。
代码实现
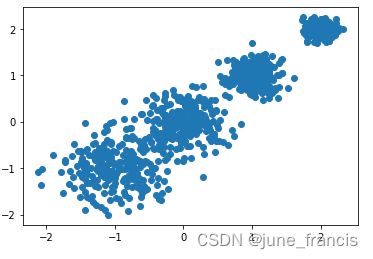
我们先用之前构建的数据来完成一个简单的聚类:
# 综合分类数据集
import numpy as np
from sklearn.datasets import make_blobs
from matplotlib import pyplot as plt
# 创建数据集
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,
# 簇中心在[-1,-1], [0,0],[1,1], [2,2], 簇方差分别为[0.4, 0.2, 0.2, 0.2]
X, y = make_blobs(n_samples=1000, n_features=2,
centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.3, 0.2, 0.1],
random_state=9)
# 数据可视化
plt.scatter(X[:, 0], X[:, 1], marker='o')
plt.show()
然后使用 sklearn.cluster.AgglomerativeClustering 完成聚类:
-
一般参数:
-
n_clusters:一个整数,指定簇的数量。 -
connectivity:一个数组或者可调用对象或者为None,用于指定连接矩阵。它给出了每个样本的可连接样本。 -
affinity:一个字符串或者可调用对象,用于计算距离。可以为:‘euclidean’, ‘l1’, ‘l2’, ‘manhattan’, ‘cosine’, ‘precomputed’ 。
如果linkage=‘ward’,则 'affinity必须是 ‘euclidean’ 。 -
memory:用于缓存输出的结果,默认为不缓存。如果给定一个字符串,则表示缓存目录的路径。 -
n_components:将在scikit-learn v 0.18中移除 -
compute_full_tree:通常当已经训练了n_clusters之后,训练过程就停止。
但是如果compute_full_tree=True,则会继续训练从而生成一颗完整的树。 -
linkage:一个字符串,用于指定链接算法。
‘ward’:采用方差恶化距离variance incress distance 。
‘complete’:全链接complete-linkage算法,采用 d m a x d_{max} dmax。
‘average’:均链接average-linkage算法,采用 d a v g d_{avg} davg。
‘single’:单链接single-linkage算法,采用 d m i n d_{min} dmin 。 -
pooling_func:即将被废弃的接口。
-
-
属性:
- labels_:一个形状为[n_samples,] 的数组,给出了每个样本的簇标记。
- n_leaves_:一个整数,给出了分层树的叶结点数量。
- n_components_:一个整数,给除了连接图中的连通分量的估计值。
- children_:一个形状为[n_samples-1,2]数组,给出了每个非叶结点中的子节点数量。
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
model = AgglomerativeClustering(
n_clusters=4, linkage="average", affinity=metric
)
model.fit(X)
print("%s Silhouette Coefficient: %0.3f"
% (metric, metrics.silhouette_score(X, model.labels_, metric='sqeuclidean')))
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c in zip(np.arange(model.n_clusters), "rgbk"):
row_ix = np.where(l == model.labels_)
plt.scatter(X[row_ix, 0], X[row_ix, 1])
plt.axis("tight")
plt.axis("off")
plt.suptitle("AgglomerativeClustering(affinity=%s)" % metric, size=20)
plt.show()
展示结果如下:
cosine Silhouette Coefficient: 0.370
euclidean Silhouette Coefficient: 0.816
cityblock Silhouette Coefficient: 0.820
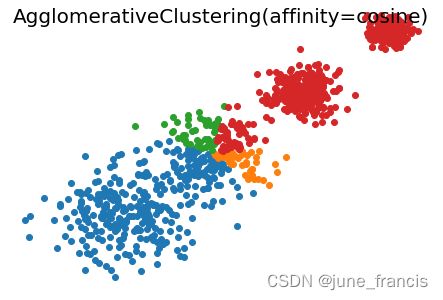
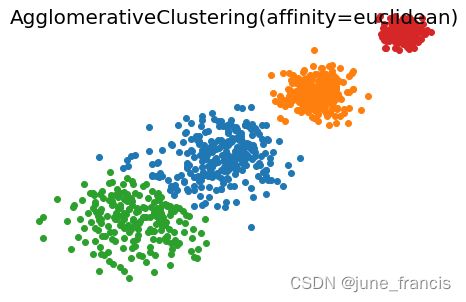
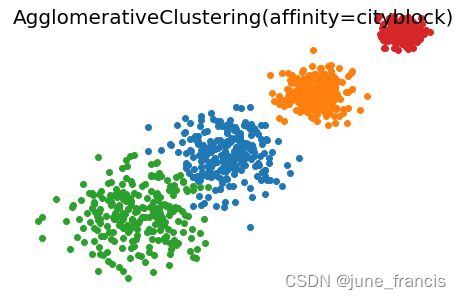
不难看出,当前场景使用 linkage='average' 和 affinity='euclidean' 的参数组合与 linkage='average' 和 affinity='cityblock' 时聚类效果更好。
接下来我们使用更为复杂的数据进行测试(以下示例来自于官方):
# Author: Gael Varoquaux
# License: BSD 3-Clause or CC-0
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import AgglomerativeClustering
from sklearn.metrics import pairwise_distances
np.random.seed(0)
# Generate waveform data
n_features = 2000
t = np.pi * np.linspace(0, 1, n_features)
def sqr(x):
return np.sign(np.cos(x))
X = list()
y = list()
for i, (phi, a) in enumerate([(0.5, 0.15), (0.5, 0.6), (0.3, 0.2)]):
for _ in range(30):
phase_noise = 0.01 * np.random.normal()
amplitude_noise = 0.04 * np.random.normal()
additional_noise = 1 - 2 * np.random.rand(n_features)
# Make the noise sparse
additional_noise[np.abs(additional_noise) < 0.997] = 0
X.append(
12
* (
(a + amplitude_noise) * (sqr(6 * (t + phi + phase_noise)))
+ additional_noise
)
)
y.append(i)
X = np.array(X)
y = np.array(y)
n_clusters = 3
labels = ("Waveform 1", "Waveform 2", "Waveform 3")
# Plot the ground-truth labelling
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c, n in zip(range(n_clusters), "rgb", labels):
lines = plt.plot(X[y == l].T, c=c, alpha=0.5)
lines[0].set_label(n)
plt.legend(loc="best")
plt.axis("tight")
plt.axis("off")
plt.suptitle("Ground truth", size=20)
# Plot the distances
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
avg_dist = np.zeros((n_clusters, n_clusters))
plt.figure(figsize=(5, 4.5))
for i in range(n_clusters):
for j in range(n_clusters):
avg_dist[i, j] = pairwise_distances(
X[y == i], X[y == j], metric=metric
).mean()
avg_dist /= avg_dist.max()
for i in range(n_clusters):
for j in range(n_clusters):
plt.text(
i,
j,
"%5.3f" % avg_dist[i, j],
verticalalignment="center",
horizontalalignment="center",
)
plt.imshow(avg_dist, interpolation="nearest", cmap=plt.cm.gnuplot2, vmin=0)
plt.xticks(range(n_clusters), labels, rotation=45)
plt.yticks(range(n_clusters), labels)
plt.colorbar()
plt.suptitle("Interclass %s distances" % metric, size=18)
plt.tight_layout()
# Plot clustering results
for index, metric in enumerate(["cosine", "euclidean", "cityblock"]):
model = AgglomerativeClustering(
n_clusters=n_clusters, linkage="average", affinity=metric
)
model.fit(X)
plt.figure()
plt.axes([0, 0, 1, 1])
for l, c in zip(np.arange(model.n_clusters), "rgbk"):
plt.plot(X[model.labels_ == l].T, c=c, alpha=0.5)
plt.axis("tight")
plt.axis("off")
plt.suptitle("AgglomerativeClustering(affinity=%s)" % metric, size=20)
plt.show()
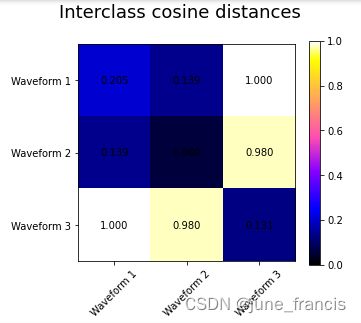
展示效果如下:
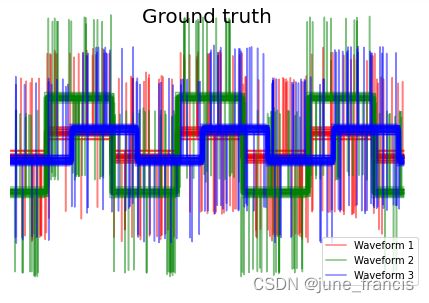
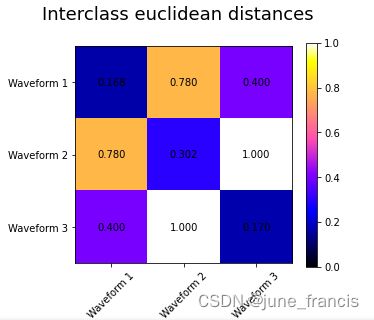
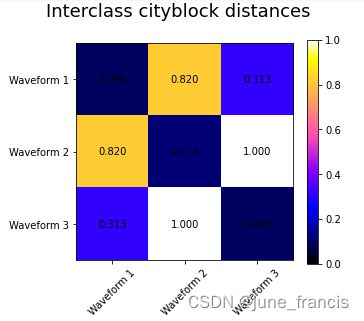
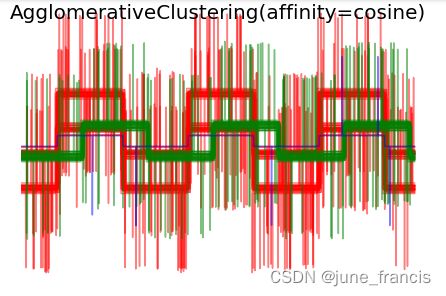
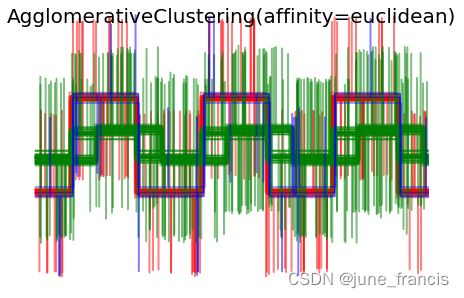
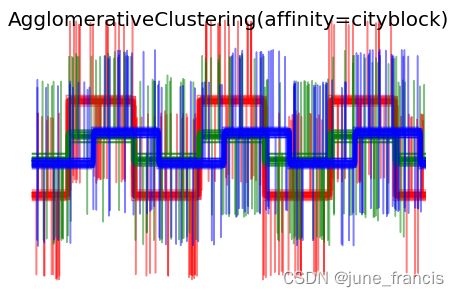
为了演示不同的距离计算方式对层次聚类结果的影响,官方示例中特意构建了波形样本集,这样的样本集可被看作高维的向量,当然了,官方提供的这些距离计算方式也通常用在高维数据场景下(特别是欧氏距离 euclidean 和曼哈顿距离 cityblock )。
示例中构建的波形数据一共有三组,第一组和第二组实际上其中一组是由另一组等比缩放得到的。而余弦距离 cosine distance 对于缩放前后的数据进行处理得到的结果是一样的,所以,它无法区分这两组波形数据,对于没有噪声的缩放数据,余弦距离同样无法分辨。
在这些波形数据中被加入了观测噪声,这些噪声都是比较稀疏的,仅有 6% 的数据包含噪声,所以,L1范数(例如 cityblock 距离)的噪声要比 L2范数(例如 euclidean 距离)的噪声要大很多。这些都能从上面的类内距离矩阵中看出来:在对角线上的数值,表明了类的分布的广度。
当我们对这些数据进行聚类时,聚类的效果反映在了距离矩阵中。在应用了欧式距离方法的类中,由于噪声的原因样本被聚类的不是很好,所以这种方式无法很好的区分这些波形样本。而对于曼哈顿距离,所有的波形数据都被很好的区分出来。然而余弦距离则无法区分波形数据1和波形数据2,应为它俩被分为了同一个类簇。
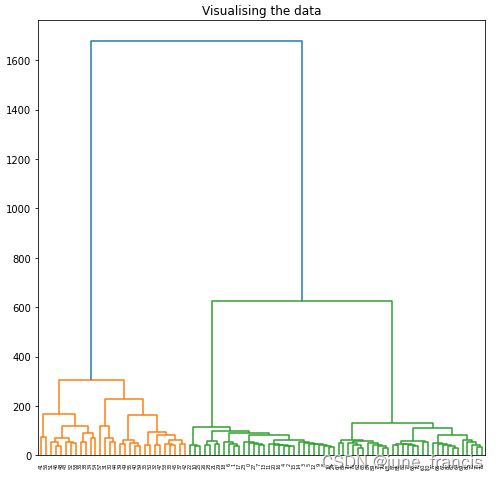
将上述构建的数据进行层次的可视化:
import scipy.cluster.hierarchy as shc
plt.figure(figsize =(8, 8))
plt.title('Visualising the data')
Dendrogram = shc.dendrogram((shc.linkage(X, method ='ward')))
参考文献
- https://scikit-learn.org/stable/auto_examples/cluster/plot_agglomerative_clustering_metrics.html#sphx-glr-auto-examples-cluster-plot-agglomerative-clustering-metrics-py
- https://www.datanovia.com/en/lessons/agglomerative-hierarchical-clustering/
- https://www.geeksforgeeks.org/implementing-agglomerative-clustering-using-sklearn/