yolov5转hisi的nnie(c and c++)
yolov5转hisi的nnie(c and c++)
-
- 总述
- 一. 训练前修改网络
- 二. 导出模型
- 三. 后处理
-
- 1. c++版
- 2. 基于hisi SDK的纯c版(后续更新)
总述
刚躺了坑,记录一下,目的是将yolov5(6.1)转成海思可以推理的wk文件,并完成后处理,实现在板子上进行推理并拿到正确结果。我的设备是3516dv300.,本文会梳理c++版本和c版本,其中c++版本可以方便调用opencv进行读图,show图,但不便于集成在hisi工程中,所以我也尝试了跟着hisi SDK sample中的yolov3改了一版纯c的v5,最终结果可以和c++版的完全对上。
ps: 附一张v5m在hisi的推理结果(size:608),效果还是很不错的:)

一. 训练前修改网络
1. 修改MaxPool层
nnie中:pooling层采用的是ceil mode(其实是因为caffe不支持floor mode)
在models/common.py如下图修改,开启ceil_model:

2. 修改Upsample层为反卷积
因为torch中的upsample是最近邻插值(nearest),而海思只支持uppooling方式,
[-1, 1, nn.Upsample, [None, 2, ‘nearest’]] 改为 [-1, 1, nn.ConvTranspose2d, [256,256, 2, 2]] 和 [-1, 1, nn.ConvTranspose2d, [128,128, 2, 2]], 其中的256与128数字是根据网络通道数来的,仅限于s模型,比如m模型需要改为384和192.
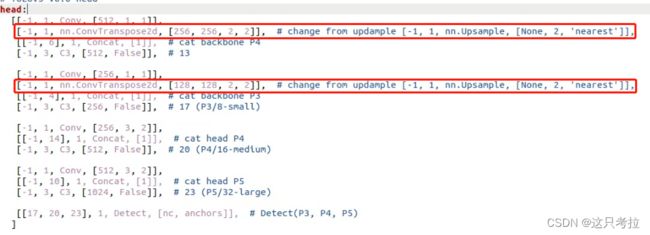
(注:6.1版yolov5没有focus层,不需要修改;6.1的激活函数是silu,即x*sigmod,也是可支持的,不需 要任何修改)
以上修改好后训练模型~
二. 导出模型
1. 导出onnx模型:
(1) 在export中opset改为9
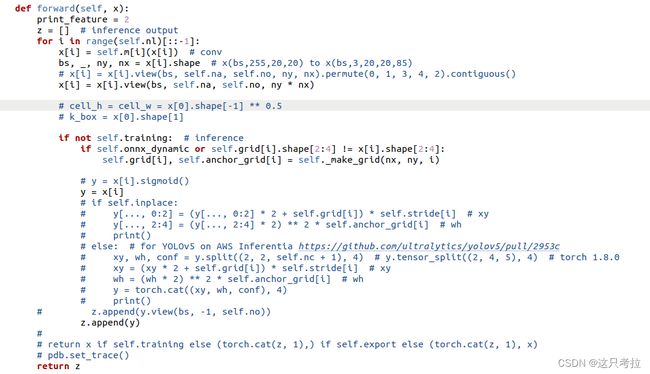
(2) 在models/yolo.py中修改detect中代码如下:
原代码:
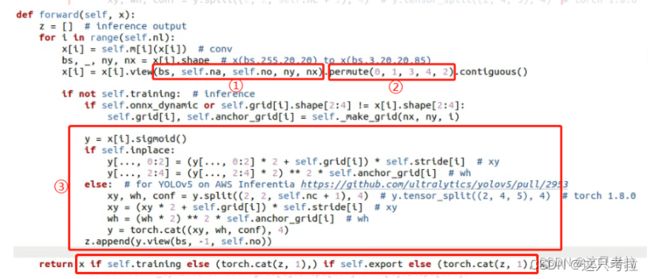
改动有以下几点:
<1>:去掉了原先的permute;
<2>:将view原来的输出维度(bs, na, no, ny, nx) 改为 (bs, na, no, ny * nx);
<3>:去除了后处理坐标点和宽高decode代码,去除cat操作
现在来分析下为什么这么改:
<1>:nnie不支持5个维度的permute(即transpose),且只支持0231的方式,过于局限,我们不妨删掉这一层,在后处理中按照合适的读取方式去找结果就好了。
<2>:nnie的reshape也只支持4维,且第一维必须是0,为了能用nnie的reshape,我们不得不把x和y共享一个维度,这导致的结果是输出结果中,x和y在同一行,我们只需按个数取值即可。
<3>:后处理中,对三个检测层分别处理,所以不需要concat
2. 导出onnx-sim模型:python3 onnxsim xxx.onnx xxx-sim.onnx
3. 导出caffe model和prototex(网上有很多教程,没什么坑)
4. 导出wk:ruyistiodio中导出(网上很多教程,没什么坑)
三. 后处理
1. c++版
参考nnie-lite工程:https://github.com/mxsurui/NNIE-lite
在此基础上添加yolov5的功能,基于里面的yolov3修改
-
yolo.cpp中yolov3DetectDemo拷贝一份,命名为yolov5DetectDemo,修改其中
feature_index0 = 2;
feature_index1 = 1;
feature_index2 = 0;
(亦可在导出onnx时交换append的顺序); -
在parseYolov3Feature函数所在位置,拷贝一份命名parseYolov5Feature,并在yolo.cpp中yolov5DetectDemo中调用此函数,
这里贴出我修改的parseYolov5Feature,其中需要考虑到数据读取方式以及后处理的方式
inline void parseYolov5Feature(int img_width,
int img_height,
int num_classes,
int kBoxPerCell,
int feature_index,
float conf_threshold,
const std::vector<cv::Size2f> &anchors,
const nnie::Mat feature,
std::vector<int> &ids,
std::vector<cv::Rect> &boxes,
std::vector<float> &confidences,
int print_level)
{
const float downscale = static_cast<float>(std::pow(2, feature_index) / 32); // downscale, 1/32, 1/16, 1/8
int cell_w = (int)std::pow(feature.width, 0.5);
int cell_h = cell_w;
for (int cy = 0; cy < cell_h; ++cy)
{
for (int cx = 0; cx < cell_w; ++cx)
{
for (int b = 0; b < kBoxPerCell; ++b)
{
int channel = b * (num_classes + 5);
float tc = feature.data[cx + (cy * cell_w) + (channel + 4) * cell_h * cell_w];
float confidence = Sigmoid(tc);
if (confidence >= conf_threshold)
{
float tx = feature.data[cx + (cy * cell_w) + channel * cell_h * cell_w];
float ty = feature.data[cx + (cy * cell_w) + (channel + 1) * cell_h * cell_w];
float tw = feature.data[cx + (cy * cell_w) + (channel + 2) * cell_h * cell_w];
float th = feature.data[cx + (cy * cell_w) + (channel + 3) * cell_h * cell_w];
float tc = feature.data[cx + (cy * cell_w) + (channel + 4) * cell_h * cell_w];
tx = Sigmoid(tx);
ty = Sigmoid(ty);
tw = Sigmoid(tw);
th = Sigmoid(th);
float x = ((float)cx - 0.5f + 2.0f * tx) / cell_w;
float y = ((float)cy - 0.5f + 2.0f * ty) / cell_h;
float w = (2.0f * tw) * (2.0f * tw) * anchors[b].width * downscale / cell_w;
float h = (2.0f * th) * (2.0f * th) * anchors[b].height * downscale / cell_h;
std::vector<float> classes(num_classes);
for (int i = 0; i < num_classes; ++i)
{
float tc_by_class = feature.data[cx + (cy * cell_w) + (channel + 5 + i) * cell_h * cell_w];
float tc_by_class_sigmoid = Sigmoid(tc_by_class);
classes[i] = tc_by_class_sigmoid;
}
auto max_itr = std::max_element(classes.begin(), classes.end());
int index = static_cast<int>(max_itr - classes.begin());
if (num_classes > 1){
confidence = confidence * classes[index];
}
int center_x = (int) (x * img_width);
int center_y = (int) (y * img_height);
int width = (int) (w * img_width);
int height = (int) (h * img_height);
int left = static_cast<int>(center_x - (width - 1.0f) * 0.5f);
int top = static_cast<int>(center_y - (height - 1.0f) * 0.5f);
if (confidence > conf_threshold){
ids.push_back(index);
boxes.emplace_back(left, top, width, height);
confidences.push_back(confidence);
}
}
}
}
}
2. 基于hisi SDK的纯c版(后续更新)
。 。 。
参考:
- https://blog.csdn.net/tangshopping/article/details/110038605