OPTICS聚类算法详解
欢迎关注”生信修炼手册”!
DBSCAN算法对于邻域半径eps和最小样本数minPoints这两个参数比较敏感,不同的参数取值会产生不同的聚类效果。为了降低参数设置对聚类结果造成的不稳定性,在DBSCAN算法的基础上,提出了OPTICS算法,全称如下
Ordering Points to identify the clustering structure
通过对样本点排序来识别聚类结构,为了搞清楚该算法,首先要理解以下两个基本概念
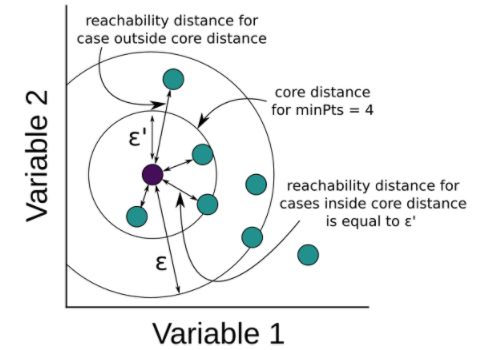
1. core distance,核心距离是使一个样本点成为core points的最小半径,在给定邻域半径eps和minPoints参数的前提下,核心距离可以比给定的eps更小
2. reachability distance,可达距离指的是样本与core point的距离
上述两种距离的定义可以参考下图来理解
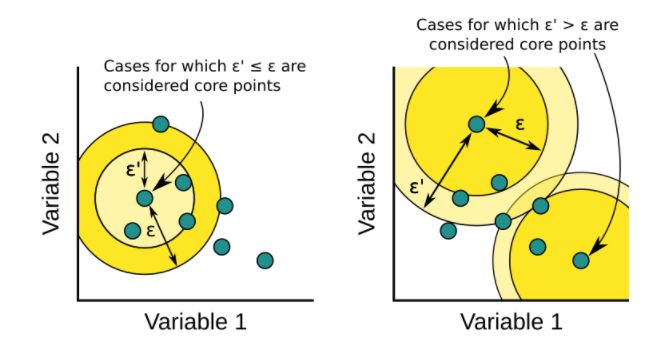
核心距离的作用用于判断一个样本是否为core points, 如果核心距离小于等于eps, 则样本为核心样本点;如果核心距离大于eps, 则样本点不是核心样本点,图示如下
可达距离则用于对样本点进行排序,这也是OPTICS算法中Ordering Points的由来。该算法的具体过程如下
1. 定义两个队列,有序队列和结果队列,有序队列用于存储core points及其密度直达points, 并按照可达距离升序排列;结果队列用于存储样本点的输出次序;有序队列中的points为待处理样本,结果队列中的points为处理之后的样本;
2. 选取一个未处理的core point, 将其放入结果队列,同时计算邻域内样本点的可达距离,按照可达距离升序将样本点依次放入有序队列;
3. 从有序队列中提取第一个样本,如果为core point, 则计算可达距离,将可达距离最小的点放入结果队列,如果不是core point, 则跳过该点,选取新的core point, 重复步骤2
4. 不断迭代第二步和第三步,直到所有样本点都处理完毕,然后输出结果队列中的样本点及其可达距离
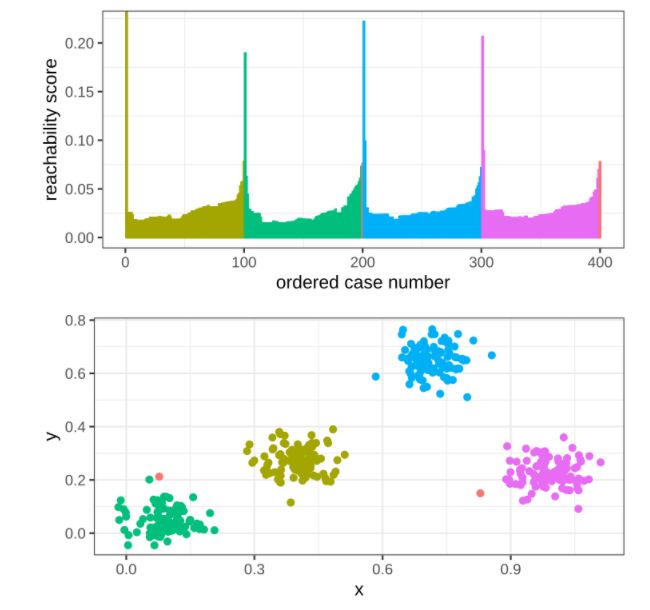
处理完毕之后,根据样本的输出顺序和可达距离,可以绘制如下所示的柱状图,其中不同的峰谷对应不同不同的cluster, 红色表示噪声点
在scikit-learn中,使用OPTICS聚类的代码如下
>>> from sklearn.cluster import OPTICS
>>> import numpy as np
>>> X = np.array([[1, 2], [2, 5], [3, 6],[8, 7], [8, 8], [7, 3]])
>>> clustering = OPTICS(min_samples=2).fit(X)
>>> clustering.labels_
array([0, 0, 0, 1, 1, 1])该算法继承了DBSCAN算法的优点,同时增强了其稳定性。
·end·
—如果喜欢,快分享给你的朋友们吧—
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。
更多精彩
KEGG数据库,除了pathway你还知道哪些
全网最完整的circos中文教程
DNA甲基化数据分析专题
突变检测数据分析专题
mRNA数据分析专题
lncRNA数据分析专题
circRNA数据分析专题
miRNA数据分析专题
单细胞转录组数据分析专题
chip_seq数据分析专题
Hi-C数据分析专题
HLA数据分析专题
TCGA肿瘤数据分析专题
基因组组装数据分析专题
CNV数据分析专题
GWAS数据分析专题
2018年推文合集
2019年推文合集
2020推文合集
写在最后
转发本文至朋友圈,后台私信截图即可加入生信交流群,和小伙伴一起学习交流。
扫描下方二维码,关注我们,解锁更多精彩内容!
一个只分享干货的
生信公众号