这图怎么画| 批量小提琴图+箱线图+散点+差异分析
写在前面
【这图怎么画】系列的图都来自VIP群里同学的提问。推文只是对图片的复现,不代表作者对图片展现形式的认同。欢迎同学们在群里分析有意思的图片。
本期图片
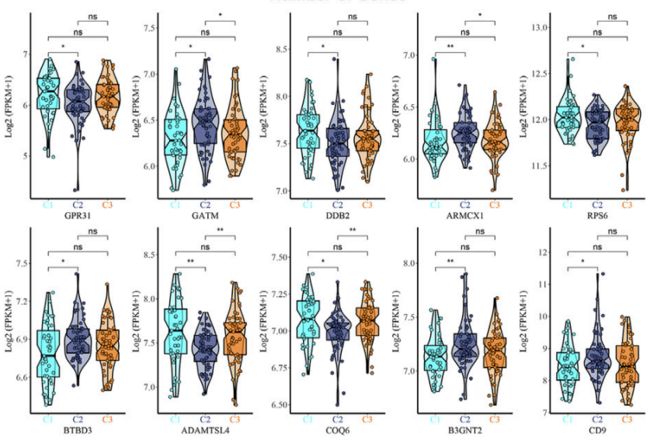
❝「Title:」Typing characteristics of metabolism-related genes in osteoporosis
「Journal:」Front. Pharmacol.
「Doi:」https://doi.org/10.1016/j.eja.2022.126692
❞
读图
❝The expression difference box plot represents the difference in expression levels of GPR31, GATM, DDB2, ARMCX1, RPS6, BTBD3, ADAMTSL4, COQ6, B3GNT2, and CD9 genes among the three isoforms.
❞
没有什么特殊。之前画过的箱线图:
跟着 Cell 学作图 | 3.箱线图+散点+差异显著性检验
跟着Nat Commun学作图 | 1.批量箱线图+散点+差异分析
跟着Nat Commun学作图 | 4.配对箱线图+差异分析
R实战 | 对称云雨图 + 箱线图 + 配对散点 + 误差棒图 +均值连线
跟着Nature学作图 | 质控箱线图
跟着 Cell 学作图 | 箱线图+散点(组间+组内差异分析)
复现结果
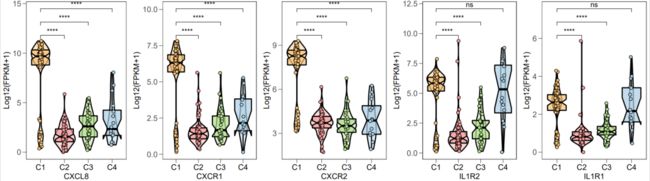 row1
row1  all
all
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞30个并保留30分钟,截图发至微信mzbj0002领取。
「木舟笔记2022年度VIP可免费领取」。
❝注:2022马上过去了,为了方便各位读者朋友,现推出
❞木舟笔记永久VIP,售价169¥。2022VIP仅需支付差价进行升级。木舟笔记永久VIP享本号所有资源(限定课程除外),后续不再推出VIP企划。
木舟笔记2022年度VIP企划
「权益:」
「2022」年度木舟笔记所有推文示例数据及代码(「在VIP群里实时更新」)。
 data+code
data+code 木舟笔记「科研交流群」。
「半价」购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
「收费:」
「99¥/人」。可添加微信:mzbj0002 转账,或直接在文末打赏。

绘图
# loda data ana preprocess
mRNA <- read.csv("All_mRNA_FPKM.csv",header=T,row.names=1)
#log2
bar_mat <- t(log2(mRNA+1))
# group info
anno <- read.csv("sample_index.csv",header=T,row.names=1)
anno$type2 <- anno$Type
anno <- anno[rownames(bar_mat),]
bar_mat <- bar_mat[rownames(anno),]
bar_mat<- as.data.frame(bar_mat)
bar_mat$sam <- anno$Type
## plot
library(RColorBrewer)
library(ggpubr)
library(ggplot2)
bar_mat$sam<-factor(bar_mat$sam,levels=c("C1","C2","C3","C4"))
# comparisons
my_comparisons <- list(c("C1", "C2"),
c("C1", "C3"),
c("C1", "C4"))
# gene list
gc <- head( colnames(bar_mat), -1)
#开始批量绘制
plist<-list()
for (i in 1:length(gc)){
bar_tmp<-bar_mat[,c(gc[i],"sam")]
colnames(bar_tmp)<-c("Expression","sam")
pb1<- ggplot(data = bar_tmp,aes(x = sam,
y = Expression ,
fill = sam))+
scale_fill_manual(values = mycol[c(7,5,3,1)]) +
geom_violin(alpha = 0.4, position = position_dodge(width = .75),
size = 0.8, color="black") +
geom_boxplot(notch = TRUE, outlier.size = -1,
color="black", lwd=0.8, alpha = 0.7) +
geom_point(shape = 21, size=2,
position = position_jitterdodge(),
color="black", alpha = 1) +
theme_bw() +
ylab("Log12(FPKM+1)") +
xlab(gc[i]) +
theme(axis.text.x = element_text(size = 12, color = "black"),
axis.ticks = element_line(size=0.2, color="black"),
axis.ticks.length = unit(0.2, "cm"),
legend.position = "none",
panel.background = element_blank(),
panel.grid = element_blank(),
axis.title = element_text(size = 12),
axis.text = element_text(size = 12)) +
stat_compare_means(method="t.test",hide.ns = F,comparisons =my_comparisons,label="p.signif")
plist[[i]]<-pb1
}
# cowplot
library(cowplot)
p <- plot_grid(plotlist = plist, ncol = 5)
## save
ggsave("boxplot1208.pdf",width = 14,height = 20)
往期内容
CNS图表复现|生信分析|R绘图 资源分享&讨论群!
这图怎么画| 有点复杂的散点图
这图怎么画 | 相关分析棒棒糖图
组学生信| Front Immunol |基于血清蛋白质组早期诊断标志筛选的简单套路
(免费教程+代码领取)|跟着Cell学作图系列合集
Q&A | 如何在论文中画出漂亮的插图?
跟着 Cell 学作图 | 桑葚图(ggalluvial)
R实战 | Lasso回归模型建立及变量筛选
跟着 NC 学作图 | 互作网络图进阶(蛋白+富集通路)(Cytoscape)
R实战 | 给聚类加个圈圈(ggunchull)
R实战 | NGS数据时间序列分析(maSigPro)
跟着 Cell 学作图 | 韦恩图(ggVennDiagram)
 木舟笔记矩阵
木舟笔记矩阵
