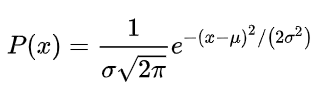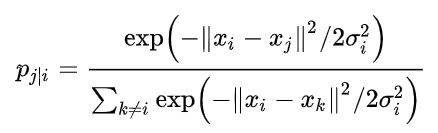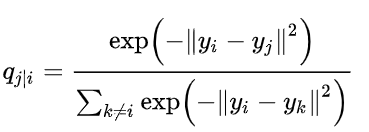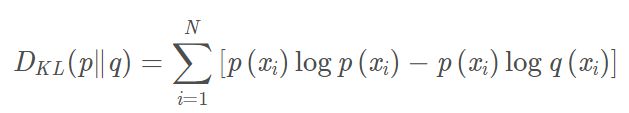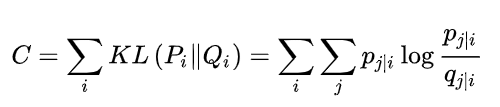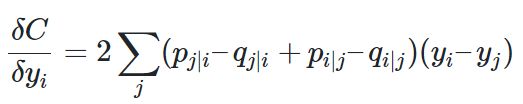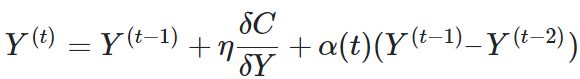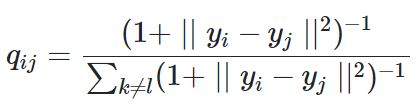使用t-SNE算法进行可视化
欢迎关注”生信修炼手册”!
t-SNE全称如下
t-Distributed Stochastic Neighbor Emdedding
是一种非线性的降维算法,常用于将数据降维到二维或者三维空间进行可视化,来观察数据的结构。
在MDS算法中,降维的基本思想是保持高维和低维空间样本点的距离不变,而t-SNE由SNE算法延伸而来,基本思想是保持降维前后概率分布不变。基于高维分布来构建概率
首先看下SNE算法,初始高维空间下两个样本点的条件概率如下
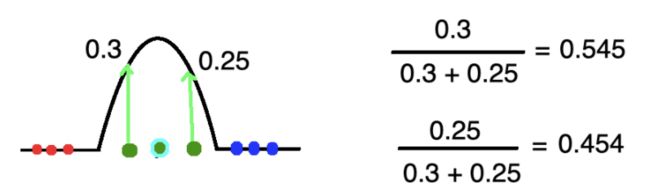
这个公式是用某个事件的概率除以所有事件的概率得出的,类似下图
降维到低维空间之后,两个样本点的条件概率如下
和高维空间相比,直接将低维空间高斯分布的方差指定为高维空间方差的1/√2。构建好两个概率分布之后,通过引入KL散度来衡量两个分布之间的差异。
KL散度又叫做相对熵,通过真实分布和理论分布之间熵的差异来衡量两个分布之间的差异,公式如下
其中p(x)表示真实的概率分布,q(x)表示理论的概率分布,在SNE算法中,对应的目标函数如下
对于该目标函数而言,由于KL函数是非对称函数,低维空间的不同距离会导致不同的权重,比如p为0.8,q为0.2, 对应的损失为
而当p为0.2,q为0.8时,cost为-0.277, 差别很大。 在求解的过程中,首先需要解决的问题就是每个样本对应的方差的设值问题,在SNE中,通过困惑度这个超参数来帮助寻找最佳的方差,困惑度的公式如下
其中H指的是样本对应的熵
通过给定一个具体的困惑度数值,通过二分查找的方式来寻找一个最佳的方差值,通常困惑度的范围为5-50。
另外,在寻找目标函数最小值的过程中,通过梯度下降法来解决问题,目标函数的梯度如下
在梯度下降的过程中,为了加速优化和避免陷入局部最优解,在梯度更新的公式中,需要引入之前梯度累加的指数衰减项,具体的公式如下
综合来看,SNE的目标函数本身存在权重问题,而且求解过程中,超参数很多,需要多次优化,比较复杂。为了克服SNE的这些问题,t-SNE被提出,其区别于SNE的地方主要是以下两点
1.使用对称的SNE
2.低维空间下使用t分布替代高斯分布
t-SNE中目标函数如下
用联合概率来替代了条件概率,从而将目标函数转换为一个对称函数。另外,低维空间的概率基于t分布来计算,公式如下
对于t-SNE算法而言,其梯度公式如下
使用t-SNE之后,解决了目标函数的非对称问题,而且t分布的处理相比高斯分布更具实际意义,如下图所示
横轴为距离,纵轴为相似度,对于较大相似度的点,在t分布中对应的距离更小,对于相似度较小的点,在t分布中对应的距离更长。对应到低维之后的样本点,就是同一簇内的点更紧密,不同簇之间的点更疏远,非常符合我们的需求。
在scikit-learn中,使用t-SNE算法的代码如下
>>> import numpy as np
>>> from sklearn.manifold import TSNE
>>> X = np.array([[0, 0, 0], [0, 1, 1], [1, 0, 1], [1, 1, 1]])
>>> X_embedded = TSNE(n_components=2).fit_transform(X)
>>> X_embedded.shape
(4, 2)t-SNE算法由于没有显示的预估部分,不能用于集合数据的直接降维,所以主要用于可视化,将数据降维到2维或者3维空间进行可视化。而且由于算法是随机的,需要多次试验选择合适的超参数,同时算法的复杂度较高,计算时间更久。
·end·
—如果喜欢,快分享给你的朋友们吧—
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。
更多精彩
KEGG数据库,除了pathway你还知道哪些
全网最完整的circos中文教程
DNA甲基化数据分析专题
突变检测数据分析专题
mRNA数据分析专题
lncRNA数据分析专题
circRNA数据分析专题
miRNA数据分析专题
单细胞转录组数据分析专题
chip_seq数据分析专题
Hi-C数据分析专题
HLA数据分析专题
TCGA肿瘤数据分析专题
基因组组装数据分析专题
CNV数据分析专题
GWAS数据分析专题
2018年推文合集
2019年推文合集
2020推文合集
写在最后
转发本文至朋友圈,后台私信截图即可加入生信交流群,和小伙伴一起学习交流。
扫描下方二维码,关注我们,解锁更多精彩内容!
一个只分享干货的
生信公众号