- 流浪地球 - 华为OD机试真题(E卷、Java)
什码情况
华为odjava数据结构算法面试机试
针对刷题难,效率慢,我们提供一对一算法辅导,针对个人情况定制化的提高计划(全称1V1效率更高)。有兴趣的同学可以扫码添加我们的微信(code5bug)了解,免费试课一下。题目描述流浪地球计划在赤道上均匀部署了N个转向发动机,按位置顺序编号为0~N。1).初始状态下所有的发动机都是未启动状态;2).发动机启动的方式分为”手动启动”和”关联启动”两种方式;3).如果在时刻1一个发动机被启动,下一个时刻
- C# Serilog配置和使用
ryan68888
c#开发语言
1.安装NuGet安装2.LogSerilog.cs类代码如下:usingSerilog;usingSerilog.Events;usingSystem;usingSystem.Collections.Generic;usingSystem.Linq;usingSystem.Text;usingSystem.Threading.Tasks;namespaceWinFormPro{publiccla
- Rust + 时序数据库 TDengine:打造高性能时序数据处理利器
涛思数据(TDengine)
时序数据库rusttdengine
引言:为什么选择TDengine与Rust?TDengine是一款专为物联网、车联网、工业互联网等时序数据场景优化设计的开源时序数据库,支持高并发写入、高效查询及流式计算,通过“一个数据采集点一张表”与“超级表”的概念显著提升性能。Rust作为一门系统级编程语言,近年来在数据库、嵌入式系统、分布式服务等领域迅速崛起,以其内存安全、高性能著称,与TDengine的高效特性天然契合,适合构建高可靠、高
- MATLAB数据的保存与读取
晚风微凉~
java前端javascript
在工程应用中,我们经常需要将未处理完的数据保存起来以便后期使用,或者在一些复杂计算中,我们需要多次计算过程中,由于系统的工作空间会随着系统的关闭而被释放掉,导致下次使用时无法快速调用,所有需要对数据进行保存与读取。1.核心代码1)数据保存基于MATALB的储存数据的常用命令是"save",使用save会将数据以二进制的方式存储在后缀名)为"文件名字.mat";savedemo01使用该命令会将数据
- MATLAB的function函数的使用
晚风微凉~
matlab开发语言
在工程应用中,我们经常会遇到算法的计算较为复杂,很多算法的过程重复次数过多的问题,针对这个问题我们可以考虑使用function函数简化代码编写的工作量。1、单个传参在使用function的函数时,我们首先需要定义function函数的结构;function[输出参数]=函数名(输入参数)%注释:function函数的使用一般是比较多的,因此需要注意注释的编写,避免后期工作的误导;主要代码:****
- 三维点云重建的原理及代码
晚风微凉~
matlab图像处理
点云重建是将来自各种传感器(如激光雷达、相机等)采集的离散点云数据转换为具有结构和几何形状的物体模型的过程。在这个过程中,算法的核心任务是从大量的离散点中提取出具有几何意义的特征,并将这些特征组合成相应的物体模型。在实际应用中,无法获得物体所有表面的三维坐标数据,因此点云重建算法必须处理部分点云数据,尽可能准确地还原物体的几何结构。点云重建的目标是通过对描述物体表面形状的点数据进行处理,根据它们的
- 编译链接过程
YancyKahn
编译链接编译链接GCC
编译链接过程C/C++程序从文本到可执行文件之间是一个复杂的过程.对于源代码(.c/.cpp)文件我们是不能直接运行的,必须经过一系列的处理才能转化为机器语言,再通过链接相应的文件转化为可执行程序.这个过程称为编译链接过程.本文篇幅较长,想直接看分析过程点击这里下面是从源代码到可执行文件的整个编译链接的过程:整个编译链接过程无非就分为编译过程和链接过程1.编译过程C文件编译过程又可分为:编译和汇编
- Android端ReactNative环境搭建——上
hzulwy
reactnativereactnativeandroidreact.js
前言最近一年,因为公司业务需要,部门引入了rn这门跨段技术来开发业务需求。从去年部门大佬调研rn这个框架到现在已有超过一年的时间了。而我从当时毕业不到1年的小白成长到现在负责维护项目的Android端代码的主力。同时,自己对rn相关的技术有了不少理解。因此,想要分享一些知识点,希望可以帮助到大家。我会以一个专栏的方式述说在这一年当中使用rn开发需求遇到的困难。大家可以借鉴参考下,共同进步!!!使用
- nvm管理多版本node,nvm 配置国内镜像,npm配置国内镜像
奇纳尼
npm前端node.js
目录前言nvm是什么?安装nvm配置nvm国内镜像nvm常用命令npm配置国内镜像:淘宝镜像nrm源管理命令:可以不用实际项目中会遇到的问题前言公司多个项目需要配置不同版本的node,按照传统方式一直切换下载安装不同版本的node太麻烦了,发现nvm可以管理多版本nodenvm是什么?nvm(node.jsversionmanagement),是一个nodejs的版本管理工具。nvm和n都是nod
- autojs之乐旅商城自动报名自动约
恶猫
javascript前端开发语言autojs安卓脚本
之前用的。现在能不能用不知道了啊。自己测试吧。//一键亮屏device.wakeUp();device.wakeUpIfNeeded();sleep(1000);//滑开swipe(device.width/2,device.height*0.8,device.width/2,device.height*0.1,2000);sleep(2000);//一键开微信launchApp("微信");sl
- C语言指针入门(一)
悄悄敲敲敲
c语言开发语言
1.什么是指针?内存会划分为一个个内存单元(一个内存单元大小为1字节)每个内存单元都有一个编号即地址也被称为指针,我们可以理解为指针就是地址。通过指针可以找到其所指向的内存单元。就像我们第一次去一个朋友家时可以通过门牌号找到她的家一样,指针就是所存储数据的“门牌号”但不同的地方是在C语言中不同类型的数据在内存中所占用的字节数和存储方式是不一样的,所以我们在存取数据时不仅要知道位置信息还要知道该数据
- C语言指针入门(二)
悄悄敲敲敲
c语言开发语言算法c++
1.二级指针二级指针就是指针的指针,即存放指针变量地址的一个变量。#includeintmain(){inta=10;int*p=&a;int**pp=&p;printf("%d\n",a);printf("%d\n",*p);printf("%d\n",**pp);//打印结果全为10printf("%p\n",a);printf("%p\n",*p);printf("%p\n",**pp);/
- 基于 STC89C52 的 8x8 点阵显示数字
@小张要努力
单片机嵌入式硬件stm3251单片机proteus
一、引言在电子设计领域,信息的有效展示是众多项目的关键环节。8x8点阵作为一种经济且实用的显示模块,能够呈现数字、简单字母及图形等信息,在电子时钟、简易游戏机等产品中广泛应用。STC89C52单片机凭借其低成本、丰富的I/O资源与稳定的性能,成为驱动8x8点阵的理想选择。本文将深入探讨如何利用STC89C52单片机结合74LS245芯片,实现8x8点阵的数字显示功能,并借助Proteus软件进行仿
- 基于 STC89C52 的智能秒表
@小张要努力
单片机stm3251单片机proteusmcuc++c语言
引言秒表作为一种常见的计时工具,在体育赛事、实验测量等众多场景中有着广泛应用。随着电子技术的发展,基于单片机的智能秒表凭借其高精度、多功能等优势逐渐取代传统机械秒表。本文将详细介绍一款基于STC89C52单片机的智能秒表设计,该秒表通过两位数码管进行时间显示。STC89C52单片机特性回顾STC89C52是一款性能卓越的8位CMOS微控制器。它拥有8K字节的系统可编程Flash存储器,可方便地存储
- pyspark学习rdd处理数据方法——学习记录
亭午
学习
python黑马程序员"""文件,按JSON字符串存储1.城市按销售额排名2.全部城市有哪些商品类别在售卖3.上海市有哪些商品类别在售卖"""frompysparkimportSparkConf,SparkContextimportosimportjsonos.environ['PYSPARK_PYTHON']=r"D:\anaconda\envs\py10\python.exe"#创建Spark
- uboot(bootrom的作用)
花落已飘
ubootlinuxuboot
BootROM详细讲解1.什么是BootROM?BootROM(BootRead-OnlyMemory)是一种固化在芯片内部的只读存储器(ROM),用于存放设备启动时执行的第一阶段引导代码。由于它是只读的,意味着其内容在芯片出厂时已经写入,无法更改。作用:当设备上电或复位时,BootROM负责最早的引导过程,它会初始化硬件环境,并决定从哪个存储介质加载操作系统或用户程序。2.BootROM的作用B
- 回归任务中的评价指标MAE,MSE,RMSE,R-Squared
旺旺棒棒冰
统计学习方法机器学习回归评价指标r2mse
转自博客。仅供自己学习使用,如有侵权,请联系删除分类任务的评价指标有准确率,P值,R值,F1值,而回归任务的评价指标就是MSE,RMSE,MAE、R-SquaredMSE均方误差MSE是真实值与预测值的差值的平方和然后求平均。通过平方的形式便于求导,所以常被用作线性回归的损失函数。MSE=1m∑i=1m(yi−y^i)2MSE=\frac{1}{m}\sum_{i=1}^{m}\left(y_{i
- 使用AIOps进行更好的事件管理
茵赛飞3D CAD数据转换软件
pagerdutydevops人工智能运维
DevOps为科技界带来了更加协作和高效的工作流程。随着AIOps的集成,自动化更进一步,使用人工智能为团队提供更快的根本原因分析和算法降噪。主要从采用AIOps中受益的主要领域之一是事件管理。AIOps可以帮助DevOps团队自动化工作流程,以实现更智能、更高效的事件管理,从而腾出时间让IT运营团队成员专注于创新以改善用户体验。在本文中,我们将了解AIOps如何从检测和识别到响应改进事件管理,以
- 第十八章:模板的多态力量_《C++ Templates》notes
郭涤生
c/c++c++开发语言笔记
模板的多态力量一、动态多态vs静态多态二、奇异递归模板模式(CRTP)三、策略模式(编译期策略选择)关键要点总结第一部分:多选题(10题)第二部分:设计题(5题)答案与详解多选题答案:设计题参考答案1.编译期策略选择器2.类型安全访问者模式3.概念约束数学库4.编译期工厂模式5.静态多态容器测试说明一、动态多态vs静态多态核心概念:动态多态:基于虚函数和继承体系,函数调用在运行时决定(通过虚函数表
- AI大模型编程能力对比:Deepseek&Claude&Gemini
黑夜路人(heiyeluren)
AI人工智能人工智能aiAIGC语言模型
在当今快速发展的技术领域,人工智能(AI)模型在编程和数据处理方面的应用越来越广泛。不同的AI模型因其独特的设计理念和技术优势,适用于不同的编程任务和场景。本文将对三种主流的AI模型——DeepSeekv3、GeminiFlash2.0和Claude3.5Sonnet的编程能力进行详细对比,帮助读者根据具体需求选择最合适的工具。同时对DeepSeekv3、GeminiFlash2.0和Claude
- React Native 迁移的阵痛
Ethan. L
ReactNative&JSreactnativereact.jsandroidios
背景由于我们的移动应用程序已经存在多年,经历了许多开发者的更替,因此变得越来越臃肿和难以维护。此外,我们团队中的Android开发人员一直很短缺,这导致我们在两个平台上的开发进度和质量存在巨大差异。因此,我们决定采用ReactNative技术,将原生工程迁移到该平台上,以提高应用程序的可维护性和整体性能。我在《ReactNative技术选型分析》中,阐述了对现有原生工程集成ReactNative的
- Android React Native应用逆向分析初探
byc6352
androidandroid
随着移动互联网时代的到来,用户在移动设备上花费的时间越来越多,不仅是因为移动设备方便携带,而且还因为层出不穷的大量应用提供为用户使用,以往在电脑上才能做的事情,现在仅靠一部手机就可以解决了。当前的移动设备厂商很多,但是被广泛使用的主流系统却只有两个,Android和iOS,因此现在大多数应用都会有两个版本,Android版本和iOS版本。然而这两种应用的开发方式却完全不同,移动客户端开发人员不得不
- 实时光线追踪技术:Ray Tracing_2024-07-21_02-55-16.Tex
chenjj4003
游戏开发python算法人工智能矩阵线性代数骨骼绑定开发语言
实时光线追踪技术:RayTracing实时光线追踪技术教程基础知识光线追踪原理光线追踪是一种渲染技术,它通过模拟光线在场景中的传播和反射来生成图像。在实时光线追踪中,这一过程被优化以在有限的时间内完成,通常用于游戏和实时动画。其核心原理是逆向追踪,即从观察者(摄像机)发出光线,而不是从光源发出,这样可以减少计算量。示例:光线追踪的基本算法#Python示例代码,展示如何计算光线与场景中物体的交点c
- DeepSeek:智能搜索与分析的新纪元
XRC2231
学习
在人工智能浪潮席卷全球的今天,DeepSeek如同一颗璀璨的新星,以其独特的魅力和强大的功能,在AI领域脱颖而出。DeepSeek,这一基于深度学习和数据挖掘技术的智能搜索与分析系统,不仅重新定义了搜索引擎的边界,更以其卓越的性能和广泛的应用场景,为全球用户带来了前所未有的智能体验。本文将从DeepSeek的定义、特点、应用场景、优势等方面进行全面而深入的介绍,带您领略这一新兴技术的独特魅力。一、
- 图像质量评价学习笔记02:IQA模型性能评价指标(PLCC、SROCC、KROCC、RMSE)
可靠的豆包蟹同志
图像质量评估IQA图像处理计算机视觉人工智能算法
性能好的图像质量评价(IQA)算法,其质量评测分数会与主观质量分数高度一致,IQA有许多评价指标,为了衡量方法测试结果与主观评价之间的一致性,视频质量专家组VQEG(VideoQualityExpertsGroup,目前国际上对视频质量进行标准化及性能测试的权威组织)提出了四个可以验证客观评价结果和主观评价结果之间的紧密程度的四个指标:PLCC、SROCC、KROCC和RMSE,也是目前最常用的I
- 【005安卓开发方案调研】之Flutter+Dart技术开发安卓
ThinkPet
移动app开发androidflutterdart跨平台
基于2025年国内移动开发环境现状,结合多份行业分析报告和技术文档,对Flutter+Dart开发安卓应用的技术成熟度和生态适配性分析如下:一、技术成熟度评估1.跨平台能力达到生产级标准Flutter的Skia自渲染引擎和Dart的AOT/JIT双编译模式,实现了90%以上的原生性能表现,在电商、社交、工具类应用中已无明显性能瓶颈。实测数据显示,列表滚动帧率稳定在55-60FPS,与原生开发差距小
- PyQt和PySide的区别和比较
PgosOcaml
pyqtmfcc++
PyQt和PySide的区别和比较PyQt和PySide是两个用于创建图形用户界面(GUI)的Python库。它们都是基于Qt框架,Qt是一个跨平台的应用程序和UI开发框架。本文将介绍PyQt和PySide之间的区别和比较,并提供相应的源代码示例。开发者许可证:PyQt的开发者许可证是商业许可证,因此如果您想在商业项目中业项目中使用PyQt,您需要购买相应的许可证。而Py业项目中使用PyQt,您需
- 10.PE导出表
蓝屏达人
PE文件结构windows
一:定位导出表PIMAGE_NT_HEADERS->OptionalHeader->DataDirectory[0]typedefstruct_IMAGE_DATA_DIRECTORY{DWORDVirtualAddress;//导出表的RVADWORDSize;//导出表大小(没用)}IMAGE_DATA_DIRECTORY,*PIMAGE_DATA_DIRECTORY;该结构的VirtualA
- 哈尔滨工业大学DeepSeek公开课人工智能:大模型原理 技术与应用-从GPT到DeepSeek|附视频下载方法
你觉得205
人工智能机器学习大数据ai知识图谱python运维
导读INTRODUCTION今天继续哈尔滨工业大学车万翔教授带来了一场主题为“DeepSeek技术前沿与应用”的报告。本报告深入探讨了大语言模型在自然语言处理(NLP)领域的核心地位及其发展历程,从基础概念出发,延伸至语言模型在机器翻译、拼音输入法、语音识别等任务中的关键作用。强调了语言模型不仅辅助其他NLP任务,本身也蕴含大量知识,如地理信息、语义理解和推理能力。随着技术的发展,尤其是trans
- Node.js 包管理配置文件详解:package.json、npmrc、package-lock.json 全面解析
还是鼠鼠
node.jsnode.jsjsonjavascript前端vscode
目录Node.js包管理配置文件详解1.package.json:Node.js项目的核心配置文件示例:完整的package.json配置关键字段解析2.package-lock.json:锁定依赖版本示例:部分package-lock.json作用如果package-lock.json出现问题3..npmrc:npm的自定义配置文件示例:修改npm源应用.npmrc配置4..nvmrc:Node
- java短路运算符和逻辑运算符的区别
3213213333332132
java基础
/*
* 逻辑运算符——不论是什么条件都要执行左右两边代码
* 短路运算符——我认为在底层就是利用物理电路的“并联”和“串联”实现的
* 原理很简单,并联电路代表短路或(||),串联电路代表短路与(&&)。
*
* 并联电路两个开关只要有一个开关闭合,电路就会通。
* 类似于短路或(||),只要有其中一个为true(开关闭合)是
- Java异常那些不得不说的事
白糖_
javaexception
一、在finally块中做数据回收操作
比如数据库连接都是很宝贵的,所以最好在finally中关闭连接。
JDBCAgent jdbc = new JDBCAgent();
try{
jdbc.excute("select * from ctp_log");
}catch(SQLException e){
...
}finally{
jdbc.close();
- utf-8与utf-8(无BOM)的区别
dcj3sjt126com
PHP
BOM——Byte Order Mark,就是字节序标记 在UCS 编码中有一个叫做"ZERO WIDTH NO-BREAK SPACE"的字符,它的编码是FEFF。而FFFE在UCS中是不存在的字符,所以不应该出现在实际传输中。UCS规范建议我们在传输字节流前,先传输 字符"ZERO WIDTH NO-BREAK SPACE"。这样如
- JAVA Annotation之定义篇
周凡杨
java注解annotation入门注释
Annotation: 译为注释或注解
An annotation, in the Java computer programming language, is a form of syntactic metadata that can be added to Java source code. Classes, methods, variables, pa
- tomcat的多域名、虚拟主机配置
g21121
tomcat
众所周知apache可以配置多域名和虚拟主机,而且配置起来比较简单,但是项目用到的是tomcat,配来配去总是不成功。查了些资料才总算可以,下面就跟大家分享下经验。
很多朋友搜索的内容基本是告诉我们这么配置:
在Engine标签下增面积Host标签,如下:
<Host name="www.site1.com" appBase="webapps"
- Linux SSH 错误解析(Capistrano 的cap 访问错误 Permission )
510888780
linuxcapistrano
1.ssh -v
[email protected] 出现
Permission denied (publickey,gssapi-keyex,gssapi-with-mic,password).
错误
运行状况如下:
OpenSSH_5.3p1, OpenSSL 1.0.1e-fips 11 Feb 2013
debug1: Reading configuratio
- log4j的用法
Harry642
javalog4j
一、前言: log4j 是一个开放源码项目,是广泛使用的以Java编写的日志记录包。由于log4j出色的表现, 当时在log4j完成时,log4j开发组织曾建议sun在jdk1.4中用log4j取代jdk1.4 的日志工具类,但当时jdk1.4已接近完成,所以sun拒绝使用log4j,当在java开发中
- mysql、sqlserver、oracle分页,java分页统一接口实现
aijuans
oraclejave
定义:pageStart 起始页,pageEnd 终止页,pageSize页面容量
oracle分页:
select * from ( select mytable.*,rownum num from (实际传的SQL) where rownum<=pageEnd) where num>=pageStart
sqlServer分页:
- Hessian 简单例子
antlove
javaWebservicehessian
hello.hessian.MyCar.java
package hessian.pojo;
import java.io.Serializable;
public class MyCar implements Serializable {
private static final long serialVersionUID = 473690540190845543
- 数据库对象的同义词和序列
百合不是茶
sql序列同义词ORACLE权限
回顾简单的数据库权限等命令;
解锁用户和锁定用户
alter user scott account lock/unlock;
//system下查看系统中的用户
select * dba_users;
//创建用户名和密码
create user wj identified by wj;
identified by
//授予连接权和建表权
grant connect to
- 使用Powermock和mockito测试静态方法
bijian1013
持续集成单元测试mockitoPowermock
实例:
package com.bijian.study;
import static org.junit.Assert.assertEquals;
import java.io.IOException;
import org.junit.Before;
import org.junit.Test;
import or
- 精通Oracle10编程SQL(6)访问ORACLE
bijian1013
oracle数据库plsql
/*
*访问ORACLE
*/
--检索单行数据
--使用标量变量接收数据
DECLARE
v_ename emp.ename%TYPE;
v_sal emp.sal%TYPE;
BEGIN
select ename,sal into v_ename,v_sal
from emp where empno=&no;
dbms_output.pu
- 【Nginx四】Nginx作为HTTP负载均衡服务器
bit1129
nginx
Nginx的另一个常用的功能是作为负载均衡服务器。一个典型的web应用系统,通过负载均衡服务器,可以使得应用有多台后端服务器来响应客户端的请求。一个应用配置多台后端服务器,可以带来很多好处:
负载均衡的好处
增加可用资源
增加吞吐量
加快响应速度,降低延时
出错的重试验机制
Nginx主要支持三种均衡算法:
round-robin
l
- jquery-validation备忘
白糖_
jquerycssF#Firebug
留点学习jquery validation总结的代码:
function checkForm(){
validator = $("#commentForm").validate({// #formId为需要进行验证的表单ID
errorElement :"span",// 使用"div"标签标记错误, 默认:&
- solr限制admin界面访问(端口限制和http授权限制)
ronin47
限定Ip访问
solr的管理界面可以帮助我们做很多事情,但是把solr程序放到公网之后就要限制对admin的访问了。
可以通过tomcat的http基本授权来做限制,也可以通过iptables防火墙来限制。
我们先看如何通过tomcat配置http授权限制。
第一步: 在tomcat的conf/tomcat-users.xml文件中添加管理用户,比如:
<userusername="ad
- 多线程-用JAVA写一个多线程程序,写四个线程,其中二个对一个变量加1,另外二个对一个变量减1
bylijinnan
java多线程
public class IncDecThread {
private int j=10;
/*
* 题目:用JAVA写一个多线程程序,写四个线程,其中二个对一个变量加1,另外二个对一个变量减1
* 两个问题:
* 1、线程同步--synchronized
* 2、线程之间如何共享同一个j变量--内部类
*/
public static
- 买房历程
cfyme
2015-06-21: 万科未来城,看房子
2015-06-26: 办理贷款手续,贷款73万,贷款利率5.65=5.3675
2015-06-27: 房子首付,签完合同
2015-06-28,央行宣布降息 0.25,就2天的时间差啊,没赶上。
首付,老婆找他的小姐妹接了5万,另外几个朋友借了1-
- [军事与科技]制造大型太空战舰的前奏
comsci
制造
天气热了........空调和电扇要准备好..........
最近,世界形势日趋复杂化,战争的阴影开始覆盖全世界..........
所以,我们不得不关
- dateformat
dai_lm
DateFormat
"Symbol Meaning Presentation Ex."
"------ ------- ------------ ----"
"G era designator (Text) AD"
"y year
- Hadoop如何实现关联计算
datamachine
mapreducehadoop关联计算
选择Hadoop,低成本和高扩展性是主要原因,但但它的开发效率实在无法让人满意。
以关联计算为例。
假设:HDFS上有2个文件,分别是客户信息和订单信息,customerID是它们之间的关联字段。如何进行关联计算,以便将客户名称添加到订单列表中?
&nbs
- 用户模型中修改用户信息时,密码是如何处理的
dcj3sjt126com
yii
当我添加或修改用户记录的时候对于处理确认密码我遇到了一些麻烦,所有我想分享一下我是怎么处理的。
场景是使用的基本的那些(系统自带),你需要有一个数据表(user)并且表中有一个密码字段(password),它使用 sha1、md5或其他加密方式加密用户密码。
面是它的工作流程: 当创建用户的时候密码需要加密并且保存,但当修改用户记录时如果使用同样的场景我们最终就会把用户加密过的密码再次加密,这
- 中文 iOS/Mac 开发博客列表
dcj3sjt126com
Blog
本博客列表会不断更新维护,如果有推荐的博客,请到此处提交博客信息。
本博客列表涉及的文章内容支持 定制化Google搜索,特别感谢 JeOam 提供并帮助更新。
本博客列表也提供同步更新的OPML文件(下载OPML文件),可供导入到例如feedly等第三方定阅工具中,特别感谢 lcepy 提供自动转换脚本。这里有导入教程。
- js去除空格,去除左右两端的空格
蕃薯耀
去除左右两端的空格js去掉所有空格js去除空格
js去除空格,去除左右两端的空格
>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>&g
- SpringMVC4零配置--web.xml
hanqunfeng
springmvc4
servlet3.0+规范后,允许servlet,filter,listener不必声明在web.xml中,而是以硬编码的方式存在,实现容器的零配置。
ServletContainerInitializer:启动容器时负责加载相关配置
package javax.servlet;
import java.util.Set;
public interface ServletContainer
- 《开源框架那些事儿21》:巧借力与借巧力
j2eetop
框架UI
同样做前端UI,为什么有人花了一点力气,就可以做好?而有的人费尽全力,仍然错误百出?我们可以先看看几个故事。
故事1:巧借力,乌鸦也可以吃核桃
有一个盛产核桃的村子,每年秋末冬初,成群的乌鸦总会来到这里,到果园里捡拾那些被果农们遗落的核桃。
核桃仁虽然美味,但是外壳那么坚硬,乌鸦怎么才能吃到呢?原来乌鸦先把核桃叼起,然后飞到高高的树枝上,再将核桃摔下去,核桃落到坚硬的地面上,被撞破了,于是,
- JQuery EasyUI 验证扩展
可怜的猫
jqueryeasyui验证
最近项目中用到了前端框架-- EasyUI,在做校验的时候会涉及到很多需要自定义的内容,现把常用的验证方式总结出来,留待后用。
以下内容只需要在公用js中添加即可。
使用类似于如下:
<input class="easyui-textbox" name="mobile" id="mobile&
- 架构师之httpurlconnection----------读取和发送(流读取效率通用类)
nannan408
1.前言.
如题.
2.代码.
/*
* Copyright (c) 2015, S.F. Express Inc. All rights reserved.
*/
package com.test.test.test.send;
import java.io.IOException;
import java.io.InputStream
- Jquery性能优化
r361251
JavaScriptjquery
一、注意定义jQuery变量的时候添加var关键字
这个不仅仅是jQuery,所有javascript开发过程中,都需要注意,请一定不要定义成如下:
$loading = $('#loading'); //这个是全局定义,不知道哪里位置倒霉引用了相同的变量名,就会郁闷至死的
二、请使用一个var来定义变量
如果你使用多个变量的话,请如下方式定义:
. 代码如下:
var page
- 在eclipse项目中使用maven管理依赖
tjj006
eclipsemaven
概览:
如何导入maven项目至eclipse中
建立自有Maven Java类库服务器
建立符合maven代码库标准的自定义类库
Maven在管理Java类库方面有巨大的优势,像白衣所说就是非常“环保”。
我们平时用IDE开发都是把所需要的类库一股脑的全丢到项目目录下,然后全部添加到ide的构建路径中,如果用了SVN/CVS,这样会很容易就 把
- 中国天气网省市级联页面
x125858805
级联
1、页面及级联js
<%@ page language="java" import="java.util.*" pageEncoding="UTF-8"%>
<!DOCTYPE HTML PUBLIC "-//W3C//DTD HTML 4.01 Transitional//EN">
&l
![]() 来寻找SimpleITK库的channel。应该会在结果当中找到SimpleITK/SimpleITK这个"channel/package"的项目。然后:
来寻找SimpleITK库的channel。应该会在结果当中找到SimpleITK/SimpleITK这个"channel/package"的项目。然后:![]() 来寻找SimpleITK库的channel。应该会在结果当中找到SimpleITK/SimpleITK这个"channel/package"的项目。然后:
来寻找SimpleITK库的channel。应该会在结果当中找到SimpleITK/SimpleITK这个"channel/package"的项目。然后:![]() 这样会显示如何安装这个包的代码,在命令行里复制它并运行即可安装。
这样会显示如何安装这个包的代码,在命令行里复制它并运行即可安装。![]()

![]() 如此一来就读取完成了。一般来讲这两个过程一气呵成,所以为了方便可以直接封装成函数:
如此一来就读取完成了。一般来讲这两个过程一气呵成,所以为了方便可以直接封装成函数: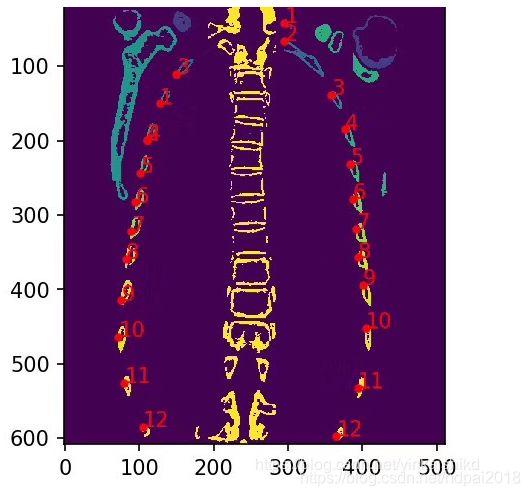 三、Pydicom的初步使用
三、Pydicom的初步使用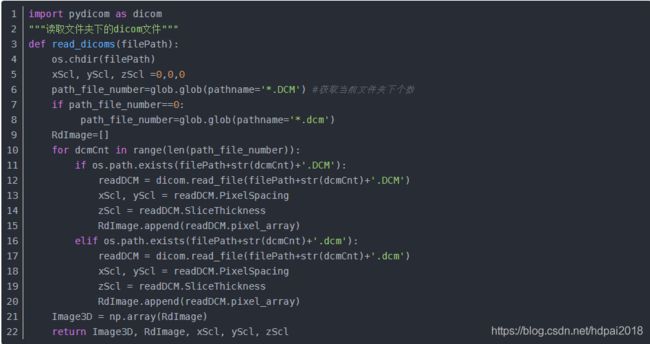 函数的输入与simpleik不同,后者输入一个文件名,而前者需要一个具体的路径。那么细心的朋友可能已经发现了程序对不同大小写的文件后缀做了区分,并且返回了numpy数据以及原始读入数据。三维矩阵的操作与simpleitk是一样的。
函数的输入与simpleik不同,后者输入一个文件名,而前者需要一个具体的路径。那么细心的朋友可能已经发现了程序对不同大小写的文件后缀做了区分,并且返回了numpy数据以及原始读入数据。三维矩阵的操作与simpleitk是一样的。