帕尔默企鹅数据集探索性分析
提示:文章写完后,目录可以自动生成,如何生成可参考右边的帮助文档
文章目录
- 前言
- 一、数据预处理
-
- 1.读取数据集(Gdrive)
- 2.四个ratio数据的预处理
- 3.sex数据字段预处理
-
- sex的相关系数
- 总结
前言
本文主要使用python对帕尔默企鹅数据集进行了EDA,欢迎交流讨论,共同进步。
前段时间老师布置了一份对帕尔默企鹅数据进行EDA的作业,结果网上一查基本全是什么“23个优秀的机器学习训练公共数据集”之类的文章,点进去也千篇一律让我无语,经过一段时间的摸鱼后也算是成功完成了作业,现写下来予以保存,因为我自己是个记忆力很差的人,所以重新捋一遍思路还是很有必要的。
本文主要分为数据预处理和数据可视化分析两节。
一、数据预处理
本次实验主要对阿德利企鹅属的三种企鹅进行探索性分析,由于三种企鹅同出一属,所以具有较高的相关性,本次实验将对相关数据集进行数据分析,以期能挖掘出数据背后隐藏的信息。
本次数据探索性分析将从数据预处理开始,并通过数据填充以消除数据集中ratio type data 的数据空值,而对于nominal type data——sex来说,则先后尝试了SVM算法和Knn算法对缺失值预测,最终选定Knn算法预测结果作为填充内容。 在获取到完整的,清洁的数据集后,开始对数据进行探索性分析。
首先我们导入本次实验过程中所需要的库
from pydrive.auth import GoogleAuth
from pydrive.drive import GoogleDrive
from google.colab import auth
from oauth2client.client import GoogleCredentials
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings("ignore")
import missingno as msno
from scipy import stats
from sklearn.svm import SVC
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import KFold
from sklearn.model_selection import cross_val_score
%matplotlib inline
import seaborn as sns
1.读取数据集(Gdrive)
然后我们读取prnguins_size.csv文件,该步骤我所使用的方式需要先将数据文件上传至Gdrive中,在通过获取sharelink获取fileID,并将数据文件存入Colab memory。由于老师要求交ipynb的文件,所以这样做,其实pycharm读取本地文件会更简单一些,不用这个ID。
auth.authenticate_user()
gauth = GoogleAuth()
gauth.credentials = GoogleCredentials.get_application_default()
drive = GoogleDrive(gauth)
# Download a file based on its file ID.
file_id1 = '1GPQb6nHNkzJqyglvNikBy7L4Eys62YF1' # Check your own ID in GDrive
downloaded = drive.CreateFile({'id': file_id1})
# Save file in Colab memory
downloaded.GetContentFile('penguins_size.csv')
通过head函数我们可以看见已经成功读取到了数据文件,并且不难发现数据中有NaN,即空值。所以我们首先检查缺失值的数量以及分布情况。
df = pd.read_csv('penguins_size.csv')
df.head(5)
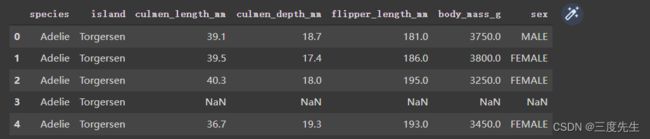
不难看到第四行都是NaN,所以接下来开始统计各个数据字段的缺失值有多少。
2.四个ratio数据的预处理
print(df.isnull().sum())

可以看到sex数据字段的缺失值最高,本次实验对缺失值的处理主要采取众数填充,且本步骤只对culmen_length_mm,culmen_depth_mm,flipper_length_mm,body_mass_g四个数据字段进行处理,对于sex数据字段来说,由于其并非属于ratio data,而是作为nominal data,所以采取众数填充并不是一个好的选择,作为标签来说,如果选用众数填充的方式处理缺失值,则很有可能会导致过采样的发生,即可能数据集中的female的数量较多,而众数填充后sex的缺失值部分全部变成了female标签,导致数据集整体偏向于female企鹅,从而导致对female企鹅数据过采样而male企鹅数据欠采样。
freq_port = df.culmen_length_mm.dropna().mode()[0] # mode返回出现最多的数据,col_name为列名
df['culmen_length_mm'] = df['culmen_length_mm'].fillna(freq_port) #采用出现最频繁的值插补
freq_port = df.culmen_depth_mm.dropna().mode()[0] # mode返回出现最多的数据,col_name为列名
df['culmen_depth_mm'] = df['culmen_depth_mm'].fillna(freq_port) #采用出现最频繁的值插补
freq_port = df.flipper_length_mm.dropna().mode()[0] # mode返回出现最多的数据,col_name为列名
df['flipper_length_mm'] = df['flipper_length_mm'].fillna(freq_port) #采用出现最频繁的值插补
freq_port = df.body_mass_g.dropna().mode()[0] # mode返回出现最多的数据,col_name为列名
df['body_mass_g'] = df['body_mass_g'].fillna(freq_port) #采用出现最频繁的值插补
一顿cv大法之后,我们来看看效果如何。
print(df.isnull().sum())
可以发现除却sex数据字段外,其余数据字段缺失值以被填充。接下来通过describe函数我们可以得知数据集目前的一些基础信息。
df.describe()

可以看到数据集中的数据个数是344个,还有其他一些基础信息,这里就不再赘述了。
3.sex数据字段预处理
本次预处理的重点其实是对sex的缺失值处理,由于sex是nominal类型的数据,所以不能简单的众数填充等等,我们先以sex数据字段分组的方式,看看sex有几个值。(其实这点是缺失值都处理完了才发现原来还有害群之马,应该先分析该数据字段的数据组成的,引以为戒。)
print(df.groupby('sex').agg({'unique'}))
可以看见除却缺失值问题之外,sex字段还有数据是“.”,很显然这也是错误的输入,应该予以修改。我们首先将sex值为“.”的这一行筛选出来进行存储,以用于之后的算法预测sex。
需要注意的是此处需要另外的变量名作为列表来存储sexPoint ,而不能直接将原sexPoint转换为列表,因为之后取sexPoint中的相关特征值训练预测sex值时,其数据结构不能是列表。
sexPoint = df.iloc[::][df.sex=='.']
print(sexPoint)
sexPointList = sexPoint.values.tolist()
print(sexPointList)
print(type(sexPoint))
print(type(sexPointList))
sex的相关系数
接下来首先计算相关系数,以确定哪些数据字段对sex数据字段具有统计学意义上的相关性。由于sex数据字段是二分称名变量,所以我们要选取Point-biserial做为相关系数。二分称名变量就是指如生死,男女这样只有两个值的变量。
# 打印出不完整样本在数据集中的下标
print(df.isna().any(axis=1).where(lambda not_exist: not_exist).dropna().index)
# 打印出缺失特征值个数统计
print(df.isna().astype(int, False).sum())
msno.matrix(df)
# 剔除数据集中不完整样本
df_com = df.drop([3, 8, 9, 10, 11, 47, 246, 286, 324, 336, 339], inplace=False)
# 计算 Point-biserial 相关系数
series_sex = df_com.loc[:,'sex'].copy()
series_sex[series_sex == 'MALE'] = 0
series_sex[series_sex == 'FEMALE'] = 1
print('Point-biserial')
for column in df_com.columns[2:6]:
cor, pvalue = stats.pointbiserialr(series_sex, df_com[column])
print(column, "<=>", 'sex')
print("correlation: ", cor)
print("pvalue: ", pvalue)
print()
结果如图:
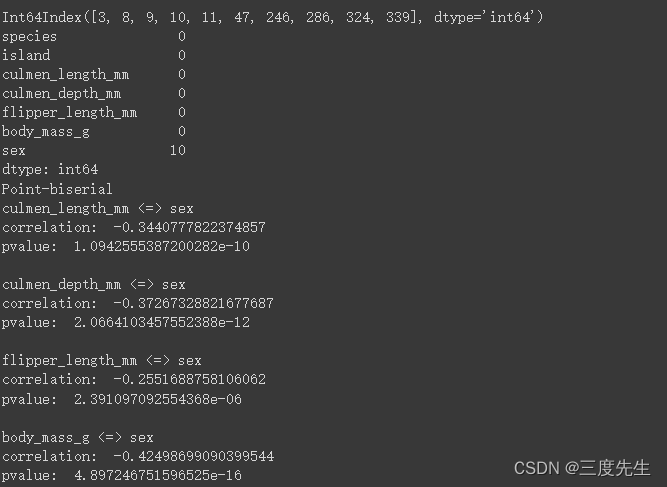

可以看到correlation值为负值,也就是说当特征(culmen_length_mm、culmen_depth_mm、flipper_length_mm、body_mass_g)值越高,则sex数据字段越趋向于male,即负相关。且Pvalue均小于0.05,因此在统计意义上是显著的。
接着计算离散型特征同目标数据字段的相关性,这里采用了卡方检测进行相关计算。
def chi_significance(x, y):
index = x.unique()
columns = y.unique()
r, c = len(index), len(columns)
count_matrix = np.zeros((r, c))
for i in range(r):
counts = y[x==index[i]].value_counts()
for j in range(c):
if columns[j] in counts.index:
count_matrix[i][j] = counts[columns[j]]
rows_total = np.sum(count_matrix, axis=1)
cols_total = np.sum(count_matrix, axis=0)
total = count_matrix.sum()
estimated_count_matrix = np.zeros((r, c))
for i in range(r):
for j in range(c):
estimated_count_matrix[i][j] = rows_total[i]*cols_total[j]/total
chi = (np.power(count_matrix - estimated_count_matrix, 2) / estimated_count_matrix).sum()
degree = (r - 1) * (c - 1)
return 1 - stats.chi2.cdf(x=chi, df=degree)
for column in ['species', 'island']:
print(column, "<=>", "sex")
print("pvalue: ", chi_significance(df_com[column], df_com['sex']))

根据species和island两特征的p值不难发现,其与目标数据字段sex的相关性并不显著,所以本次实验并不将species和island作为预测sex的关键字段。
总结
以上就是今天要讲的内容,主要对ratio类型的数据进行了缺失值填充,并对接下来的重点目标sex字段进行了相关性分析,接下来的部分就是对sex缺失值预测了,期间使用了svm算法和knn算法,并成功对sex的值进行了预测以及缺失值填充。多谢各位支持!
源码链接