IF:8+ 小儿感染性休克的特征基因及与免疫细胞浸润的关系分析
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
桓峰基因公众号推出转录组分析教程整理如下:
RNA 1. 基因表达那些事--基于 GEO
RNA 2. SCI文章中基于GEO的差异表达基因之 limma
RNA 3. SCI 文章中基于T CGA 差异表达基因之 DESeq2
RNA 4. SCI 文章中基于TCGA 差异表达之 edgeR
RNA 5. SCI 文章中差异基因表达之 MA 图
RNA 6. 差异基因表达之-- 火山图 (volcano)
RNA 7. SCI 文章中的基因表达——主成分分析 (PCA)
RNA 8. SCI文章中差异基因表达--热图 (heatmap)
RNA 9. SCI 文章中基因表达之 GO 注释
RNA 10. SCI 文章中基因表达富集之--KEGG
RNA 11. SCI 文章中基因表达富集之 GSEA
RNA 12. SCI 文章中肿瘤免疫浸润计算方法之 CIBERSORT
RNA 13. SCI 文章中差异表达基因之 WGCNA
RNA 14. SCI 文章中差异表达基因之 蛋白互作网络 (PPI)
RNA 15. SCI 文章中的融合基因之 FusionGDB2
RNA 16. SCI 文章中的融合基因之可视化
RNA 17. SCI 文章中的筛选 Hub 基因 (Hub genes)
RNA 18. SCI 文章中基因集变异分析 GSVA
RNA 19. SCI 文章中无监督聚类法 (ConsensusClusterPlus)
RNA 20. SCI 文章中单样本免疫浸润分析 (ssGSEA)
RNA 21. SCI 文章中单基因富集分析
RNA 22. SCI 文章中基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(ESTIMATE)
RNA 23. SCI文章中表达基因模型的风险因子关联图(ggrisk)
RNA 24. SCI文章中基于TCGA的免疫浸润细胞分析 (TIMER)
RNA 25. SCI文章中估计组织浸润免疫细胞和基质细胞群的群体丰度(MCP-counter)
RNA 26. SCI文章中基于转录组数据的基因调控网络推断 (GENIE3)
RNA 27 SCI文章中转录因子结合motif富集到调控网络 (RcisTarget)
桓峰基因公众号推出临床预测模型教程,整理如下:
Topic 1. 临床标志物生信分析常规思路
Topic 2. 生存分析之 Kaplan-Meier
Topic 3. SCI文章第一张表格--基线表格
Topic 4. 临床预测模型构建 Logistic 回归
Topic 5. 样本量确定及分割
Topic 6 计数变量泊松回归
Topic 7. 临床预测模型--Cox回归
Topic 8. 临床预测模型-Lasso回归
Topic 9. SCI 文章第二张表—单因素回归分析表
Topic 10. 单因素 Logistic 回归分析—单因素分析表格
Topic 11. SCI中多元变量筛选—单/多因素表
Topic 12 临床预测模型—列线表 (Nomogram)
Topic 13. 临床预测模型—一致性指数 (C-index)
Topic 14. 临床预测模型之校准曲线 (Calibration curve)
Topic 15. 临床预测模型之决策曲线 (DCA)
Topic 16. 临床预测模型之接收者操作特征曲线 (ROC)
Topic 17. 临床预测模型之缺失值识别及可视化
Topic 18. 临床预测模型之缺失值插补方法
Topic 19. 临床预测模型之输出每个患者列线图得分 (nomogramFormula)
Topic 20. 临床预测模型之竞争风险模型及计算生存概率
基于机器学习构建临床预测模型整理如下:
MachineLearning 1. 主成分分析(PCA)
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
这期分享一篇2022年11月发表在 Front Immunol(IF=8.786)的文章,作者研究小儿感染性休克的特征基因及与免疫细胞浸润的关系分析,为非肿瘤结合免疫提供了新的研究方向。
摘 要
背景: 儿童感染性休克的早期诊断对预后至关重要。本研究旨在探讨儿童感染性休克的特征基因及其与免疫细胞的关系。
方法: 我们从GEO数据库中筛选感染性休克儿童数据集,并分析差异表达基因(DEGs)。对这些DEG进行了功能富集分析。采用加权基因共表达网络分析(WCGNA)对关键模块进行筛选。最后应用最小绝对收缩选择算子(LASSO)和随机森林分析对特征基因进行鉴定。然后利用基因集富集分析(GSEA)探讨与中枢基因相关的信号通路。利用CIBERSORT对免疫细胞浸润情况进行分类。
结果: 从GSE26440中共筛选出534个DEG。然后通过WGCNA将数据聚为17个模块,其中MEgrey模块与小儿感染性休克显著相关(cor=−0.62,p<0.0001)。采用LASSO和随机森林算法选取特征基因,包括UPP1、S100A9、KIF1B、S100A12、SLC26A8。这些特征基因的受体工作特征曲线(ROC)分别为0.965、0.977、0.984、0.991和0.989,并在GSE13904的外部数据集中得到验证。GSEA分析表明,这些特征基因参与果糖和甘露糖代谢及淀粉和蔗糖代谢信号通路的正相关。CIBERSORT认为这些特征基因可能参与免疫细胞的浸润。
结论: UPP1、S100A9、KIF1B、S100A12、SLC26A8对小儿感染性休克有显著的诊断作用,并参与免疫细胞浸润。
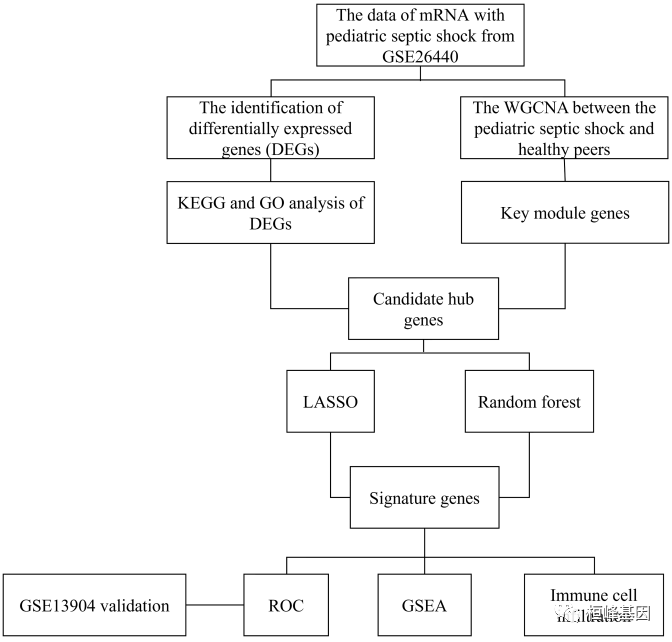
生信分析流程
相关数据准备
数据集选择:测试集:GSE26440;验证集:GSE13904
生信分析方法:
我们从文章的分析流程中提取所有的分析内容,整理出来就 7 个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于生信分析文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
1. 利用 limma 包筛选 DEGs
2. GO功能和KEGG通路富集分析
3. WGCNA 加权基因共表达网络分析
4. 筛选候选标记基因LASSO 和 Random forest构建模型
5. 外部数据验证 ROC
6. 对候选基因进行GSEA富集分析
7. CIBERSORT 免疫细胞浸润分析
研究结果
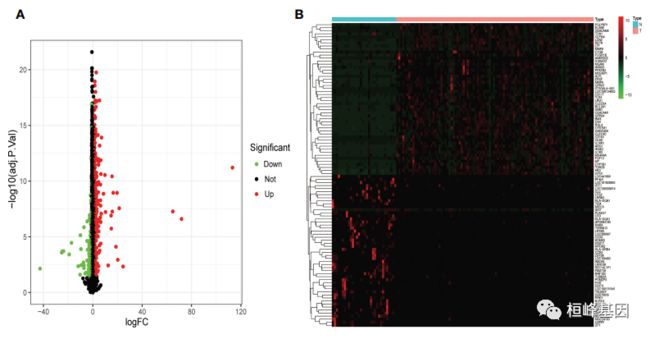
1. 小儿感染性休克中DEG的鉴定
(A) Volcano在儿童感染性休克和健康人群中显示了DEGs的表达;
(B)热图显示了前50个上调和50个下调的DEGs。
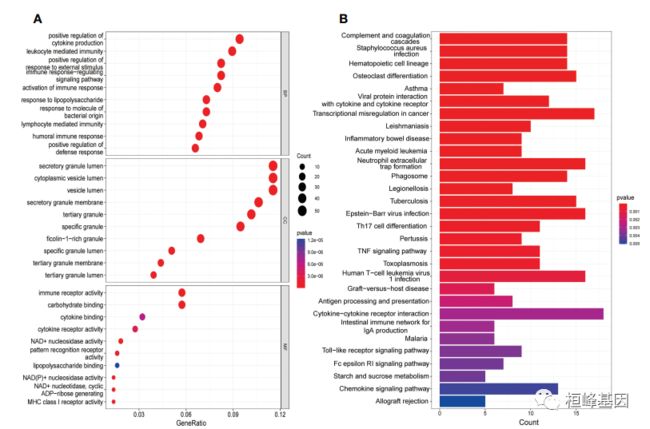
2. DEGs的功能富集分析
(A) BP、CC、MF分析中功能富集前10位;
(B) DEGs的KEGG分析
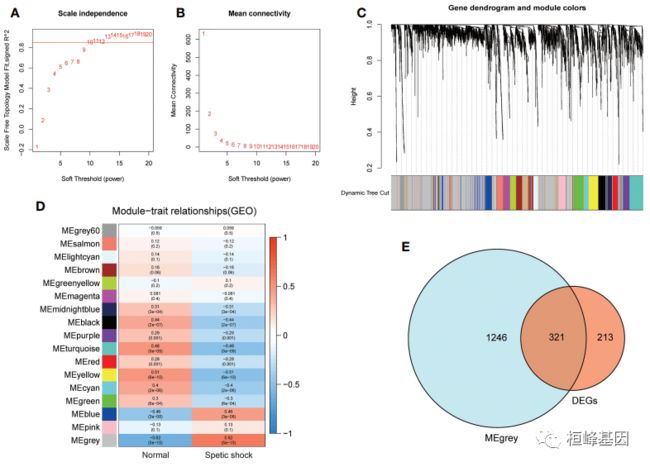
3. GSE26440的WGCNA分析及hub候选基因的鉴定
(A) WGCNA的软阈值功率;
(B) WGCNA的平均连通性;
(C) WGCNA聚类树状图;
(D) WGCNA的集群模块;
(E) MEgrey模块中DEGs与基因的相互作用图。
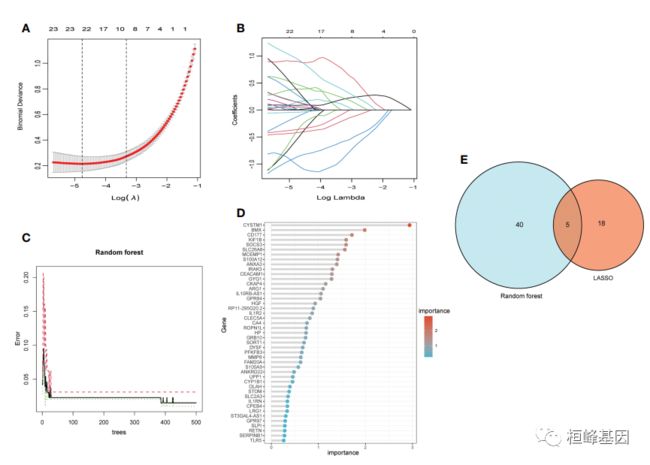
4. 机器学习对标记基因进行筛选
(A) LASSO模型的惩罚图,误差条表示标准误差;
(B) LASSO图显示,随着k罚值的增加,参数的系数大小变化幅度缩小;
(C)随机森林模型的误差率置信区间;
(D)随机森林模型中基因的相对重要性大于0.25;
(E) LASSO和随机森林算法的相互作用。
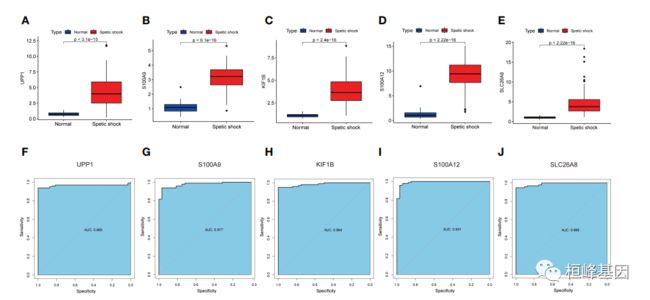
5. GSE26440中特征基因的表现
(A-E)感染性休克患儿与健康人群的特征基因表达;
(F-J) ROC显示特征基因的诊断性能。
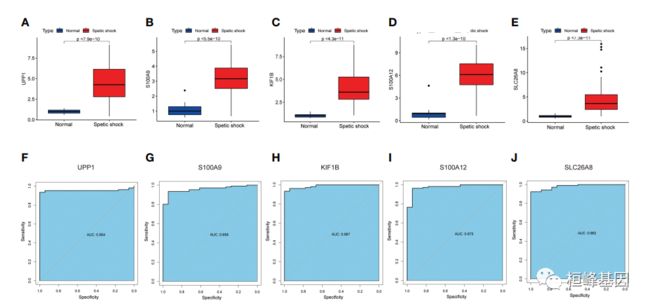
6. GSE13904中特征基因的表现
(A-E)感染性休克患儿与健康人群的特征基因表达;
(F-J) ROC显示特征基因的诊断性能。
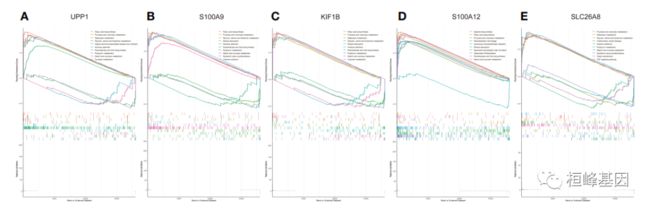
7. 儿科特征基因的GSEA
(A)小儿感染性休克中UPP1的GSEA;
(B) S100A9在小儿感染性休克中的GSEA;
(C) KIF1B在小儿感染性休克中的GSEA;
(D) S100A12在小儿感染性休克中的GSEA;
(E)小儿感染性休克中SLC26A8的GSEA。
8. 免疫细胞浸润与特征基因相关
(A)儿童感染性休克与健康人群之间的免疫细胞浸润;
(B)特征基因与显著不同的免疫细胞浸润之间的关联。
这篇文章利用桓峰基因公众号上的教程完全可以实现这些图表的绘制,过程需要自己细致的探究,毕竟不是很简单的分析,能发到8+也需要有一定的生信分析和结果解读的能力,有需要此类分析的老师可以联系桓峰基因!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
References:
Fan J, Shi S, Qiu Y, Liu M, Shu Q. Analysis of signature genes and association with immune cells infiltration in pediatric septic shock. Front Immunol. 2022;13:1056750. Published 2022 Nov 10. doi:10.3389/fimmu.2022.1056750