生物信息学|用于预测药物-药物相互作用事件的多模态深度学习框架
本篇推文引自:A multimodal deep learning framework for predicting drug–drug interaction events
1. 摘要
动机:药物-药物相互作用(DDIs)是药物研究的主要关注之一。很多基于机器学习的方法被提出用于DDI的预测,但大多数都是预测两种药物是否相互作用。研究发现DDIs可引起不同的后续事件,预测DDI相关事件更有助于探讨联合用药或不良反应背后隐藏的机制。
结果:在本文中,我们从DrugBank数据库中收集DDIs,通过依赖分析和事件调整,提取出65类DDI事件。我们提出了一个名为DDIMDL的多模态深度学习框架,该框架将多种药物特性与深度学习相结合,以构建预测DDI相关事件的模型。DDIMDL首先利用四种药物特征:化学子结构、靶点、酶和通路,分别构建基于深度神经网络(DNN)的子模型,然后采用联合DNN框架对子模型进行组合,以学习药物-药物对的交叉模态表征,预测DDI事件。在计算实验中,DDIMDL具有较高的精度和效率。此外,DDIMDL优于最先进的DDI事件预测方法和基线方法。在药物的所有特征中,化学子结构似乎是最能提供信息的。结合子结构、靶标和酶,DDIMDL的准确率为0.8852,在精确-召回曲线下的面积为0.9208。
可用性和实现:在https://github.com/YifanDengWHU/DDIMDL可以获得源代码和数据。
2. 介绍
DrugBank是一个可靠的数据源,对DDIs的描述具有统一的语法,因此我们可以通过标准描述提取DDI事件并进行进一步的研究。第二,药库具有多种药物特征。Ryu等人(2018)只使用了药物化学子结构。综合研究和提高性能需要考虑更多的特性。第三,特征之间存在冗余,如何有效的将不同的特征组合在一起是一个挑战。直接合并不同的特征向量是一种常用的方法,但是我们需要更强大和有用的方法来合并不同的特征。为了解决上述问题,我们提出了一种名为DDIMDL的计算方法,该方法结合多种药物特征和深度学习来预测DDI事件。我们定义了一个标准方案来分析药库的DDI事件,并选择65种主要事件进行分析。此外,我们还收集了药物的四个特征:化学亚结构、靶标、酶和建模途径。在DDIMDL中,根据每种药物特征构造四个子模型,并使用联合DNN框架将子模型组合起来,以学习药物-药物对的交叉模态表示。最后,我们利用学习到的交叉模态表征预测与DDI相关的事件。综上所述,本文的主要贡献如下:
我们关注于DrugBank中已知DDIs的细粒度描述,并通过对这些描述应用语义分析来构建交互事件数据集。
我们提出了一个名为DDIMDL的多模态深度学习框架,它利用深度学习和药物的不同特性来预测DDI事件。
实验结果表明,DDIMDL具有较高的效率和精度,性能优于所比较的方法。
3. 材料和方法
3.1 数据集
DrugBank (Knox et al., 2011)是一个提供12 151种药物的综合信息的资源,其中包括3844种FDA批准的药物和5867种实验药物。在研究中,我们从DrugBank中收集了DDIs以及药物的四个特征:化学亚结构、靶标、途径和酶。我们选择那些与其它药物相互作用且具有上述四个特征的药物,得到641种药物,其中双DDIs为105,824。
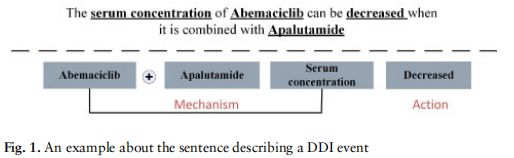
此外,我们关注药品库中的DDIs和它们的描述,并观察到这些交互是由几种句子类型描述的。图1显示了“阿培aciclib与阿帕鲁胺联合使用可降低血清浓度”的例子。这句话描述了Abemaciclib和Apalutamide之间的DDI事件。为了更好地理解DDI事件,我们将DDI事件的表示定义为四元组结构:(药物a、药物B、机制、作用),其中“机制”指的是药物在代谢、血清浓度、治疗效果等方面的作用。“动作”表示词元化后的增减。由于DrugBank中关于DDIs的描述具有固定的语法,我们使用StanfordNLP工具(Qi et al., 2018)获取依赖关系。通过使用该工具,我们可以为每个单词获得一个元组。我们在这里使用NLP术语的缩写,它们的全名可以在补充表S2中找到。由于预先训练的模型没有在生物医学文本上进行训练,因此它无法为一些专业药物的名称安排合适的词性。为了解决该模型的不足,我们建立了药品名称列表,并进行命名实体识别来获取药品。事件的动作总是依赖树的根,因此我们从动作出发,寻找与动作有特殊依赖关系的机制的根,然后对其子树进行遍历,得到整个机制。我们将影响药效的药物命名为drugA,其余的命名为drugB。这样构建了我们的四元结构fdrugA,drugB,mechanism,actiong。具体过程如算法1所示。通过这种方式,我们总共获得了110种类型的事件来描述DDIs。99%以上的交互只与一个事件关联,因此我们删除与多个事件关联的DDIs。为了便于分析,我们删除罕见事件,并选择具有超过10个DDIs的事件。
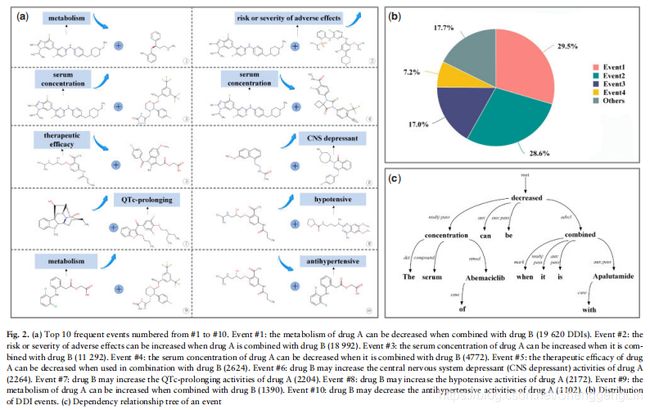
因此,我们获得了572种药物和74 528对DDIs,它们与65种类型的事件相关。我们根据这些事件的发生频率降序排列,将它们从1号排列到65号。图2a中给出了编号为#1到#10的事件作为示例,图2b中显示了事件占所有事件的百分比。所有事件的详情见补充表S1。
3.2 DDIMDL方法概述
在本研究中,我们设计了一个名为DDIMDL的多模态深度神经网络(DNN),它结合药物的不同特性来预测ddi相关事件。
3.2.1 特征提取模块
多模态深度学习预测方法将药物的不同特征作为模态,并结合它们进行预测。特征提取和表示是模型构建的关键。
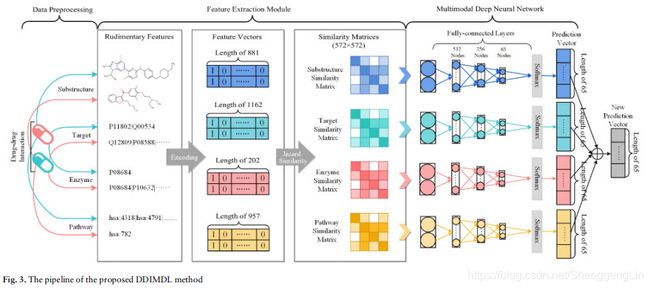
如2.1节所示,我们有药物的四个特征:化学亚结构、靶点、酶和途径,这些特征带来了药物的不同信息。每个特征对应于一组描述符,因此药物可以用一个二进制特征向量表示,其值(1或0)表示对应描述符的存在或不存在。这里,我们以化学子结构为例。Pubchem定义了881种子结构,即分子指纹。根据化学子结构,药物的表示是881维位向量,值1或0表示某种子结构类型的存在或不存在。同样,与药物相关的靶点有1162种,靶点特征可以将药物i编码为1162维位向量。这样,一个药物可以用四个特征表示为四种类型的向量。
这些特征向量具有高维数,且多数维数为0,从而对特征进行了压缩,降低了稀疏性。采用Jaccard相似度度量方法从位向量中计算药物-药物的两两相似度,而不是使用位向量作为输入。Jaccard相似度计算公式(1)。
3.2.2 DDIMDL的构建和模型优化
由于我们有几个特征,我们用DNN基于每个特征构造子模型。
不同子模型的组合对于DDIMDL很重要。在这里,子模型的输出通过平均算子组合产生最终的预测结果。如图3所示,我们根据不同的特征构造子模型,然后将子模型结合起来构建预测模型。
采用交叉熵作为损失函数,对DDIMDL模型进行了经验训练和优化。我们使用提前停止策略(Prechelt, 1998),如果在10个周期内没有观察到任何改进,就会自动停止训练。该策略可以有效地防止过拟合,同时大大加快训练速度。我们使用批大小为256的Adam作为优化算法来训练网络。
4. 结论
本研究从DrugBank获取DDI数据,运用NLP技术根据描述语法将DDI相关事件划分为65种类型,编译了包含572种药物、74 528种交互和65种DDI相关事件的数据集。提出了一种基于深度学习的多模式深度学习框架DDIMDL,该框架将多种药物特性与深度学习相结合,用于DDI事件预测。使用5-CV评估,DDIMDL优于现有的DDI事件预测方法和基线方法。综上所述,语义分析的使用使我们能够对DrugBank事件进行显著的分类,而多模式学习为整合不同特征和花费合理的训练时间提供了有力的途径。多模态深度学习框架是一种很有前途的DDI事件预测工具。
由于文章在服务器上,详细内容见;http://bbit.vip/service/main.php?version=1&type=article&id=154