- 【机器学习笔记Ⅰ】7 向量化
巴伦是只猫
机器学习机器学习笔记人工智能
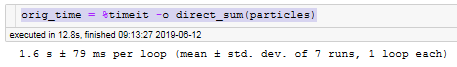
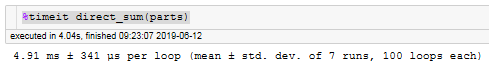
向量化(Vectorization)详解向量化是将数据或操作转换为向量(或矩阵)形式,并利用并行计算高效处理的技术。它是机器学习和数值计算中的核心优化手段,能显著提升代码运行效率(尤其在Python中避免显式循环)。1.为什么需要向量化?(1)传统循环的缺陷低效:Python的for循环逐元素操作,速度慢。代码冗长:需手动处理每个元素。示例:计算两个数组的点积(非向量化)a=[1,2,3]b=[4
- 【Python】python_jwt
宅男很神经
python开发语言
1.1传统会话(Session)机制的黄金时代与黄昏在Web应用的黎明时期,身份验证的范式几乎完全由**基于服务器端会话(Session-BasedAuthentication)**的机制所主导。这是一个直观且在单体应用时代极其有效的模型,其工作流程如同一场精密的双人舞:凭证交换与“储物柜钥匙”的签发:用户在登录页面输入用户名和密码。这些凭证被发送到服务器。服务器验证其有效性后,会在自己的“储物间
- 【Python】simulink与python联合仿真
1.1Simulink的边界:事件驱动、算法复杂性与AI集成瓶颈Simulink的核心优势在于其强大的微分方程求解器和对连续时间系统、离散时间系统的精确描述能力。其基于“信号流”和“框图”的建模范式,使得工程师可以直观地构建与物理现实高度对应的数学模型。然而,这种优势也带来了其天然的局限性:基于时间的驱动核心(Time-BasedCoreEngine):Simulink的“心脏”是一个时间驱动的仿
- python profile_python程序之profile分析
操作系统:CentOS7.3.1611_x64python版本:2.7.5问题描述1、Python开发的程序在使用过程中很慢,想确定下是哪段代码比较慢;2、Python开发的程序在使用过程中占用内存很大,想确定下是哪段代码引起的;解决方案使用profile分析分析cpu使用情况可以使用profile和cProfile对python程序进行分析,这里主要记录下cProfile的使用,profile参
- Python知识点:如何使用memory_profiler进行内存分析
开篇,先说一个好消息,截止到2025年1月1日前,翻到文末找到我,赠送定制版的开题报告和任务书,先到先得!过期不候!如何使用memory_profiler进行Python代码内存分析在开发高性能的Python应用程序时,理解和优化内存使用是至关重要的。memory_profiler是一个强大的工具,它可以帮助你监控Python代码的内存使用情况。本文将介绍如何使用memory_profiler来分
- 【Python】memory_profiler
宅男很神经
python开发语言
1.1引用计数与垃圾回收:Python的“贴身管家”与“清洁工”Python,特别是其标准实现CPython,其内存管理的核心是建立在一个优雅而高效的组合机制之上的:以引用计数为主,分代垃圾回收为辅。1.引用计数(ReferenceCounting):主要的内存管家这是CPython内存管理的基石。其原理极其简单:CPython中的每一个对象(一个整数、一个列表、一个自定义类的实例),其内部都维护
- Python 数据分析实践:车辆行驶数据处理心得
lzzy-lt-0415
python数据分析开发语言
在数据驱动决策的大趋势下,Python凭借其丰富的数据分析库,成为处理各类数据的得力工具。近期我围绕车辆行驶数据展开分析,过程中收获诸多实战经验,在此分享用Python进行数据处理与分析的心得,也结合代码讲讲实际运用思路。一、数据导入与初步探索:开启分析第一步importpandasaspd#导入数据df=pd.read_excel(r'../../数据层/数据集合/车辆行驶记录表单2.xlsx'
- Pillow 安装使用教程
小奇JAVA面试
安装使用教程pillowmicrosoft深度学习
一、Pillow简介Pillow是Python图像处理库PIL(PythonImagingLibrary)的友好分支,是图像处理的事实标准。它支持打开、编辑、转换、保存多种图像格式,常用于图像批量处理、验证码识别、缩略图生成等应用场景。二、安装Pillow2.1使用pip安装(推荐)pipinstallPillow2.2验证安装importPILprint(PIL.__version__)若无报错
- python炫酷烟花表白源代码-python炫酷烟花表白源代码
weixin_37988176
天天敲代码的朋友,有没有想过代码也可以变得很酷炫又浪漫?今天就教大家用Python模拟出绽放的烟花,工作之余也可以随时让程序为自己放一场烟花秀。python炫酷烟花表白源代码这个有趣的小项目并不复杂,只需一点可视化技巧,100余行Python代码和程序库Tkinter,最后我们就能达到下面这个效果:学完本教程后,你也能做出这样的烟花秀。整体概念梳理我们的整个理念比较简单。如上图示,我们这里通过让画
- Python实例题:基于 Flask 的在线聊天系统
目录Python实例题题目要求:解题思路:代码实现:Python实例题题目基于Flask的在线聊天系统要求:使用Flask框架构建一个实时在线聊天系统,支持以下功能:用户注册、登录和个人资料管理一对一实时聊天功能群聊功能消息通知和未读消息提示在线用户状态显示使用Flask-SocketIO实现实时通信。使用SQLite数据库存储用户、聊天记录等信息。添加美观的前端界面,支持响应式设计。解题思路:使
- pickle.dump()
ddfa1234
java开发语言
pickle.dump()pickle.dump()是Python标准库中的一个函数,用于将Python对象序列化并保存到文件中。函数签名:pickle.dump(obj,file,protocol=None,*,fix_imports=True)参数说明:obj:要序列化的Python对象。file:要保存到的文件对象。可以是一个文件名的字符串,也可以是一个已经打开的文件对象。protocol:
- python炫酷烟花表白源代码,html代码烟花特效python
liuyifan0
pygamepython开发语言
大家好,小编来为大家解答以下问题,python绘制烟花特定爆炸效果,python炫酷烟花表白源代码,今天让我们一起来看看吧!代码实现:importpygameimportrandomimportmath#屏幕宽度SCREEN_WIDTH=1350SCREEN_HEIGHT=800#烟花颜色COLORS=[(255,0,0),(0,255,0),(0,0,255),(255,255,0),(255,
- Flask 安装使用教程
小奇JAVA面试
安装使用教程flaskpython后端
一、Flask简介Flask是一个使用Python编写的轻量级Web应用框架,核心设计理念是简单易用、模块化扩展性强。Flask提供了路由、模板、请求响应等基本功能,适合构建中小型网站、RESTfulAPI、微服务架构等。二、环境准备2.1安装Python确保已安装Python3.7或以上版本:python--version如未安装,可前往:https://www.python.org/downl
- Python炫酷烟花
Want595
pythonpygame开发语言
系列文章序号直达链接Tkinter1Python李峋同款可写字版跳动的爱心2Python跳动的双爱心3Python蓝色跳动的爱心4Python动漫烟花5Python粒子烟花Turtle1Python满屏飘字2Python蓝色流星雨3Python金色流星雨4Python漂浮爱心5Python爱心光波①6Python爱心光波②7Python满天繁星8Python五彩气球9Python白色飘雪10Pyt
- C++之利用红黑树作为底层,实现对set和map的封装(难)
模拟封装map和set一.回顾红黑树二.模拟实现map和set2.1复⽤红⿊树,实现insert结构体SetKeyOfT结构体MapKeyOfT2.2⽀持iterator的实现迭代器具体实现部分上层迭代器操作普通迭代器与const迭代器map与set红黑树1.前缀递增操作符`++`2.前缀递减操作符`--`示例前缀递增操作符`++`前缀递减操作符`--`2.3map操作符[]代码解析成员变量和函数
- 第一章Pandas快速入门
Hajo_
深入浅出Pandas学习代码pythonpandas
《深入浅出Pandas》第一章代码数据来源:https://www.gairuo.com/file/data/dataset/team.xlsximportnumpyasnpimportpandasaspdfile_path='E:\\Data_python\\anconda_code\\Dive_into_Pandas\\data_files\\'team_path='team.xlsx'tea
- Python实例题:基于 Python 的简单文件管理器
狐凄
实例python开发语言前端
目录Python实例题题目要求:解题思路:代码实现:Python实例题题目基于Python的简单文件管理器要求:使用Python构建一个简单的文件管理器,支持以下功能:浏览文件和目录创建、删除、重命名文件和目录复制、移动文件和目录查看文件属性和内容搜索文件和目录使用tkinter构建图形用户界面。支持基本的文件操作权限检查。解题思路:使用os和shutil模块进行文件操作。通过tkinter构建用
- go Lock Sleep
贵哥的编程之路(热爱分享 为后来者)
golang
packagemainimport("fmt""sync""time")//Goods结构体,包含一个map[int]int用于存储商品编号和库存数量,以及一个互斥锁typeGoodsstruct{vmap[int]int//商品编号到库存数量的映射msync.Mutex//互斥锁,保证并发安全}//Inc方法,增加指定商品编号的库存数量func(g*Goods)Inc(keyint,numint
- c++ python 共享内存
qianbo_insist
音视频和c++java物联网c++c++python开发语言
一、目的是为了c++来读取并解码传递给python,Python做测试非常方便,c++和python之间必须定好协议,整体使用c++来解码,共享内存传递给python二、主类主类,串联decoder,注意decoder并没有直接在显存里面穿透,是解码以后传递给内存,从内存传给python#pragmaonce#define__STDC_CONSTANT_MACROS#defineSDL_MAIN_
- Python 数据分析与可视化 Day 14 - 建模复盘 + 多模型评估对比(逻辑回归 vs 决策树)
蓝婷儿
pythonpython数据分析逻辑回归
✅今日目标回顾整个本周数据分析&建模流程学会训练第二种模型:决策树(DecisionTree)掌握多模型对比评估的方法与实践输出综合对比报告:准确率、精确率、召回率、F1等指标为后续模型调优与扩展打下基础一、本周流程快速回顾步骤内容第1天高级数据操作(索引、透视、变形)第2天缺失值和异常值处理第3天多表合并与连接第4天特征工程(编码、归一化、时间)第5天数据集拆分(训练集/测试集)第6天逻辑回归模
- Django5.1(91)—— 如何删除一个 Django 应用
小天的铁蛋儿
djangoPythondjangopython后端
如何删除一个Django应用Django提供了将一组功能组织成名为应用程序的Python包的能力。当需求发生变化时,应用程序可能会变得过时或不再需要。以下步骤将帮助你安全地删除一个应用程序。删除所有与该应用程序相关的引用(导入、外键等)。从相应的models.py文件中删除所有模型。通过运行makemigrations来创建相关的迁移。这一步会生成一个迁移,用于删除已删除模型的表,以及与这些模型相
- 【python实用小脚本-125】基于 Python 的 Gmail 邮件发送工具:实现高效邮件自动化
Kyln.Wu
Pythonpython自动化网络
引言在现代办公和开发环境中,邮件通信是一种重要的沟通方式。自动化发送邮件可以大大提高工作效率,例如发送通知、报告或文件。本文将介绍一个基于Python的Gmail邮件发送工具,它能够通过Gmail的SMTP服务器发送邮件,并支持附件功能。该工具主要利用了Python的smtplib库和email库,结合了邮件构建和网络通信技术,为用户提供了一个简单易用的邮件发送解决方案。总体功能概述Gmail邮件
- Python之聚合函数
_AndyLau
手把手学pythonpython
Python聚合函数文章目录Python聚合函数聚合函数使用多个聚合函数结合`annotate`和`values`进行分组聚合注意事项F表达式和Q表达式F表达式Q表达式注意事项视图HTML中的表单概述Django中表单概述ModelForm关键点使用示例创建ModelForm在视图中使用ModelForm模板总结Cookie和SessionDjango中的Cookie操作Django中的Sessi
- Python报错信息归类以及处理
ʕᵔᴥᵔʔPython的错误和异常可以分为多个类别,了解这些类别有助于更好地调试和处理错误。以下是Python中常见报错信息的归类和分析。1.语法错误(SyntaxError)在代码执行前被解析器捕获的错误,通常是由于代码不符合Python语法规则。常见子类:IndentationError:缩进错误TabError:Tab和空格混用示例:#缺少冒号ifTrueprint("Hello")#Syn
- python 获取mac地址
Take_a_chestnut
python小工具python开发语言
python获取mac地址方法一:使用socket库使用了socket库中的ioctl函数和fcntl模块来获取MAC地址importsocketimportfcntlimportstructdefget_mac_address():interface='eth0'#替换为你的网络接口名称,例如eth0或en0sock=socket.socket(socket.AF_INET,socket.SOC
- LRU缓存算法在搜索引擎中的应用
数据结构与算法学习
缓存算法搜索引擎ai
LRU缓存算法在搜索引擎中的应用关键词:LRU算法、缓存淘汰、搜索引擎、哈希表、双向链表、性能优化、访问频率摘要:本文深入探讨了LRU(最近最少使用)缓存算法在搜索引擎中的关键应用。我们将从基本概念出发,通过生活化的比喻解释LRU的工作原理,分析其在搜索引擎架构中的具体实现方式,并通过Python代码示例展示如何构建一个高效的LRU缓存系统。文章还将讨论LRU算法的数学建模、实际应用场景以及未来发
- 60天python训练计划----day59
在之前的学习中,我们层层递进的介绍了时序模型的发展,从AR到MA到ARMA,再到ARIMA。本质就是把数据处理的操作和模型结合在一起了,实际上昨天提到的季节性差分也可以合并到模型中,让流程变得更加统一。季节性差分用S来表示,所以这个模型叫做SARIMA模型一、SARIMA模型SARIMA(SeasonalAutoRegressiveIntegratedMovingAverage)是标准ARIMA模
- 【python实用小脚本-128】基于 Python 的 Hacker News 爬虫工具:自动化抓取新闻数据
Kyln.Wu
Pythonpython爬虫自动化
引言在技术社区中,HackerNews是一个汇聚最新技术文章和讨论的热门平台。许多开发者和技术爱好者依赖它来获取行业动态和前沿资讯。然而,手动浏览和筛选这些文章可能耗时且低效。本文将介绍一个基于Python的HackerNews爬虫工具,它能够自动化地从HackerNews网站抓取最新文章,并将结果保存为CSV文件。该工具主要利用了Python的requests、BeautifulSoup和csv
- python系列之:使用md5和sha256完成签名认证,调用接口
快乐骑行^_^
前端和后端开发python系列使用md5和sha256完成签名认证调用接口
python系列之:使用md5和sha256完成签名认证,调用接口MD5签名和sha256签名认证md5认证代码sha256认证代码拼接签名生成签名拼接url调用接口MD5签名和sha256签名认证MD5签名认证算法特性:生成128位(16字节)的哈希值计算速度快已被证明存在碰撞漏洞(不同输入可能产生相同输出)签名认证流程:发送方对原始数据计算MD5哈希值将哈希值附加到数据中发送接收方重新计算接收
- Kafka系列之:不删除Kafka Topic,清理Kafka Topic中的数据
快乐骑行^_^
KafkaKafka系列不删除KafkaTopic清理KafkaTopic数据
Kafka系列之:不删除KafkaTopic,清理KafkaTopic中的数据一、需求二、Java删除Topic中数据三、python删除Topic中数据一、需求需要清理topic中的数据但是不能通过删除topic删除数据,则采取基于topic的offset删除topic中的数据二、Java删除Topic中数据HashMapdeleteRecords=newHashMap<>();这一行创建了一个
- 解线性方程组
qiuwanchi
package gaodai.matrix;
import java.util.ArrayList;
import java.util.List;
import java.util.Scanner;
public class Test {
public static void main(String[] args) {
Scanner scanner = new Sc
- 在mysql内部存储代码
annan211
性能mysql存储过程触发器
在mysql内部存储代码
在mysql内部存储代码,既有优点也有缺点,而且有人倡导有人反对。
先看优点:
1 她在服务器内部执行,离数据最近,另外在服务器上执行还可以节省带宽和网络延迟。
2 这是一种代码重用。可以方便的统一业务规则,保证某些行为的一致性,所以也可以提供一定的安全性。
3 可以简化代码的维护和版本更新。
4 可以帮助提升安全,比如提供更细
- Android使用Asynchronous Http Client完成登录保存cookie的问题
hotsunshine
android
Asynchronous Http Client是android中非常好的异步请求工具
除了异步之外还有很多封装比如json的处理,cookie的处理
引用
Persistent Cookie Storage with PersistentCookieStore
This library also includes a PersistentCookieStore whi
- java面试题
Array_06
java面试
java面试题
第一,谈谈final, finally, finalize的区别。
final-修饰符(关键字)如果一个类被声明为final,意味着它不能再派生出新的子类,不能作为父类被继承。因此一个类不能既被声明为 abstract的,又被声明为final的。将变量或方法声明为final,可以保证它们在使用中不被改变。被声明为final的变量必须在声明时给定初值,而在以后的引用中只能
- 网站加速
oloz
网站加速
前序:本人菜鸟,此文研究总结来源于互联网上的资料,大牛请勿喷!本人虚心学习,多指教.
1、减小网页体积的大小,尽量采用div+css模式,尽量避免复杂的页面结构,能简约就简约。
2、采用Gzip对网页进行压缩;
GZIP最早由Jean-loup Gailly和Mark Adler创建,用于UNⅨ系统的文件压缩。我们在Linux中经常会用到后缀为.gz
- 正确书写单例模式
随意而生
java 设计模式 单例
单例模式算是设计模式中最容易理解,也是最容易手写代码的模式了吧。但是其中的坑却不少,所以也常作为面试题来考。本文主要对几种单例写法的整理,并分析其优缺点。很多都是一些老生常谈的问题,但如果你不知道如何创建一个线程安全的单例,不知道什么是双检锁,那这篇文章可能会帮助到你。
懒汉式,线程不安全
当被问到要实现一个单例模式时,很多人的第一反应是写出如下的代码,包括教科书上也是这样
- 单例模式
香水浓
java
懒汉 调用getInstance方法时实例化
public class Singleton {
private static Singleton instance;
private Singleton() {}
public static synchronized Singleton getInstance() {
if(null == ins
- 安装Apache问题:系统找不到指定的文件 No installed service named "Apache2"
AdyZhang
apachehttp server
安装Apache问题:系统找不到指定的文件 No installed service named "Apache2"
每次到这一步都很小心防它的端口冲突问题,结果,特意留出来的80端口就是不能用,烦。
解决方法确保几处:
1、停止IIS启动
2、把端口80改成其它 (譬如90,800,,,什么数字都好)
3、防火墙(关掉试试)
在运行处输入 cmd 回车,转到apa
- 如何在android 文件选择器中选择多个图片或者视频?
aijuans
android
我的android app有这样的需求,在进行照片和视频上传的时候,需要一次性的从照片/视频库选择多条进行上传
但是android原生态的sdk中,只能一个一个的进行选择和上传。
我想知道是否有其他的android上传库可以解决这个问题,提供一个多选的功能,可以使checkbox之类的,一次选择多个 处理方法
官方的图片选择器(但是不支持所有版本的androi,只支持API Level
- mysql中查询生日提醒的日期相关的sql
baalwolf
mysql
SELECT sysid,user_name,birthday,listid,userhead_50,CONCAT(YEAR(CURDATE()),DATE_FORMAT(birthday,'-%m-%d')),CURDATE(), dayofyear( CONCAT(YEAR(CURDATE()),DATE_FORMAT(birthday,'-%m-%d')))-dayofyear(
- MongoDB索引文件破坏后导致查询错误的问题
BigBird2012
mongodb
问题描述:
MongoDB在非正常情况下关闭时,可能会导致索引文件破坏,造成数据在更新时没有反映到索引上。
解决方案:
使用脚本,重建MongoDB所有表的索引。
var names = db.getCollectionNames();
for( var i in names ){
var name = names[i];
print(name);
- Javascript Promise
bijian1013
JavaScriptPromise
Parse JavaScript SDK现在提供了支持大多数异步方法的兼容jquery的Promises模式,那么这意味着什么呢,读完下文你就了解了。
一.认识Promises
“Promises”代表着在javascript程序里下一个伟大的范式,但是理解他们为什么如此伟大不是件简
- [Zookeeper学习笔记九]Zookeeper源代码分析之Zookeeper构造过程
bit1129
zookeeper
Zookeeper重载了几个构造函数,其中构造者可以提供参数最多,可定制性最多的构造函数是
public ZooKeeper(String connectString, int sessionTimeout, Watcher watcher, long sessionId, byte[] sessionPasswd, boolea
- 【Java命令三】jstack
bit1129
jstack
jstack是用于获得当前运行的Java程序所有的线程的运行情况(thread dump),不同于jmap用于获得memory dump
[hadoop@hadoop sbin]$ jstack
Usage:
jstack [-l] <pid>
(to connect to running process)
jstack -F
- jboss 5.1启停脚本 动静分离部署
ronin47
以前启动jboss,往各种xml配置文件,现只要运行一句脚本即可。start nohup sh /**/run.sh -c servicename -b ip -g clustername -u broatcast jboss.messaging.ServerPeerID=int -Djboss.service.binding.set=p
- UI之如何打磨设计能力?
brotherlamp
UIui教程ui自学ui资料ui视频
在越来越拥挤的初创企业世界里,视觉设计的重要性往往可以与杀手级用户体验比肩。在许多情况下,尤其对于 Web 初创企业而言,这两者都是不可或缺的。前不久我们在《右脑革命:别学编程了,学艺术吧》中也曾发出过重视设计的呼吁。如何才能提高初创企业的设计能力呢?以下是 9 位创始人的体会。
1.找到自己的方式
如果你是设计师,要想提高技能可以去设计博客和展示好设计的网站如D-lists或
- 三色旗算法
bylijinnan
java算法
import java.util.Arrays;
/**
问题:
假设有一条绳子,上面有红、白、蓝三种颜色的旗子,起初绳子上的旗子颜色并没有顺序,
您希望将之分类,并排列为蓝、白、红的顺序,要如何移动次数才会最少,注意您只能在绳
子上进行这个动作,而且一次只能调换两个旗子。
网上的解法大多类似:
在一条绳子上移动,在程式中也就意味只能使用一个阵列,而不使用其它的阵列来
- 警告:No configuration found for the specified action: \'s
chiangfai
configuration
1.index.jsp页面form标签未指定namespace属性。
<!--index.jsp代码-->
<%@taglib prefix="s" uri="/struts-tags"%>
...
<s:form action="submit" method="post"&g
- redis -- hash_max_zipmap_entries设置过大有问题
chenchao051
redishash
使用redis时为了使用hash追求更高的内存使用率,我们一般都用hash结构,并且有时候会把hash_max_zipmap_entries这个值设置的很大,很多资料也推荐设置到1000,默认设置为了512,但是这里有个坑
#define ZIPMAP_BIGLEN 254
#define ZIPMAP_END 255
/* Return th
- select into outfile access deny问题
daizj
mysqltxt导出数据到文件
本文转自:http://hatemysql.com/2010/06/29/select-into-outfile-access-deny%E9%97%AE%E9%A2%98/
为应用建立了rnd的帐号,专门为他们查询线上数据库用的,当然,只有他们上了生产网络以后才能连上数据库,安全方面我们还是很注意的,呵呵。
授权的语句如下:
grant select on armory.* to rn
- phpexcel导出excel表简单入门示例
dcj3sjt126com
PHPExcelphpexcel
<?php
error_reporting(E_ALL);
ini_set('display_errors', TRUE);
ini_set('display_startup_errors', TRUE);
if (PHP_SAPI == 'cli')
die('This example should only be run from a Web Brows
- 美国电影超短200句
dcj3sjt126com
电影
1. I see. 我明白了。2. I quit! 我不干了!3. Let go! 放手!4. Me too. 我也是。5. My god! 天哪!6. No way! 不行!7. Come on. 来吧(赶快)8. Hold on. 等一等。9. I agree。 我同意。10. Not bad. 还不错。11. Not yet. 还没。12. See you. 再见。13. Shut up!
- Java访问远程服务
dyy_gusi
httpclientwebservicegetpost
随着webService的崛起,我们开始中会越来越多的使用到访问远程webService服务。当然对于不同的webService框架一般都有自己的client包供使用,但是如果使用webService框架自己的client包,那么必然需要在自己的代码中引入它的包,如果同时调运了多个不同框架的webService,那么就需要同时引入多个不同的clien
- Maven的settings.xml配置
geeksun
settings.xml
settings.xml是Maven的配置文件,下面解释一下其中的配置含义:
settings.xml存在于两个地方:
1.安装的地方:$M2_HOME/conf/settings.xml
2.用户的目录:${user.home}/.m2/settings.xml
前者又被叫做全局配置,后者被称为用户配置。如果两者都存在,它们的内容将被合并,并且用户范围的settings.xml优先。
- ubuntu的init与系统服务设置
hongtoushizi
ubuntu
转载自:
http://iysm.net/?p=178 init
Init是位于/sbin/init的一个程序,它是在linux下,在系统启动过程中,初始化所有的设备驱动程序和数据结构等之后,由内核启动的一个用户级程序,并由此init程序进而完成系统的启动过程。
ubuntu与传统的linux略有不同,使用upstart完成系统的启动,但表面上仍维持init程序的形式。
运行
- 跟我学Nginx+Lua开发目录贴
jinnianshilongnian
nginxlua
使用Nginx+Lua开发近一年的时间,学习和实践了一些Nginx+Lua开发的架构,为了让更多人使用Nginx+Lua架构开发,利用春节期间总结了一份基本的学习教程,希望对大家有用。也欢迎谈探讨学习一些经验。
目录
第一章 安装Nginx+Lua开发环境
第二章 Nginx+Lua开发入门
第三章 Redis/SSDB+Twemproxy安装与使用
第四章 L
- php位运算符注意事项
home198979
位运算PHP&
$a = $b = $c = 0;
$a & $b = 1;
$b | $c = 1
问a,b,c最终为多少?
当看到这题时,我犯了一个低级错误,误 以为位运算符会改变变量的值。所以得出结果是1 1 0
但是位运算符是不会改变变量的值的,例如:
$a=1;$b=2;
$a&$b;
这样a,b的值不会有任何改变
- Linux shell数组建立和使用技巧
pda158
linux
1.数组定义 [chengmo@centos5 ~]$ a=(1 2 3 4 5) [chengmo@centos5 ~]$ echo $a 1 一对括号表示是数组,数组元素用“空格”符号分割开。
2.数组读取与赋值 得到长度: [chengmo@centos5 ~]$ echo ${#a[@]} 5 用${#数组名[@或
- hotspot源码(JDK7)
ol_beta
javaHotSpotjvm
源码结构图,方便理解:
├─agent Serviceab
- Oracle基本事务和ForAll执行批量DML练习
vipbooks
oraclesql
基本事务的使用:
从账户一的余额中转100到账户二的余额中去,如果账户二不存在或账户一中的余额不足100则整笔交易回滚
select * from account;
-- 创建一张账户表
create table account(
-- 账户ID
id number(3) not null,
-- 账户名称
nam