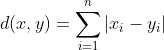【机器学习笔记8】K-mean聚类实现与应用
目录
K-mean聚类算法
距离计算
欧氏距离
曼哈顿距离
余弦相似度
K-mean算法实现
步骤1:随机选择初始质心
步骤2:欧式距离计算以及寻找最近质心
步骤3:根据分好的簇重新计算质心
步骤4:组合K-mean各模块
绘图
主函数(含数据标准化)
运行结果:
使用K-mean进行图像压缩
数据预处理
压缩图像
绘制图像
使用sklearn实现K-mean
K-mean算法的缺点
前言:请确保已知晓无监督学习和聚类的定义。
K-mean聚类算法
K-mean又称K均值算法,意思是将数据集分为K簇,每个簇以属于该簇的数据集均值为该簇的中心(或质心)。K-mean算法流程大致为:
-
首先随机选择K个初始聚类中心。
-
计算各个点到每个聚类中心的距离,将各个点归到距离最近的簇中。
-
用每个簇的数据重新计算各自的中心。
-
重复2、3过程,直到簇中心不再变化或者超过最大迭代次数。
可以看出,K-mean算法相对来说很简单,容易实现。但也导致它有一些缺点,关于K-mean算法的优缺点将在后面讨论。
在上述过程中,第二步中提到了计算距离,接下来先讨论距离计算。
距离计算
不同情况的距离度量选择是不同的,下面介绍常用的三种:
欧氏距离
曼哈顿距离
余弦相似度
K-mean算法实现
需要数据集的可以私。
需要的包:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScalerStandardScal包是拿来标准化数据集的,关于手动实现Z-score标准化请见:
多元线性回归模型_Twilight Sparkle.的博客-CSDN博客
下面每一步都对照上面说到的步骤。
步骤1:随机选择初始质心
# 随机选择样本最为初始质心
def init_centroid(daraSet,K):
randIdx = np.random.permutation(daraSet.shape[0])
centroids = daraSet[randIdx[:K]]
return centroids步骤2:欧式距离计算以及寻找最近质心
# 寻找最近质心索引
def find_closed_centroid(dataSet,centroids):
K = centroids.shape[0] # 质心个数
idx = np.zeros(dataSet.shape[0],dtype=int) # 样本到最近质心的索引
for i in range(dataSet.shape[0]):
# 计算一个样本到所有质心的欧氏距离
diff = np.tile(dataSet[i],(K,1)) - centroids # tile可以复制矩阵,用法自查
squaredDist = np.sum(diff**2,axis=1) # axis = 1计算每一行的和
distance = squaredDist ** 0.5
# 选出距离最小的质点索引
idx[i] = np.argmin(distance)
return idx步骤3:根据分好的簇重新计算质心
# 计算质心
def compute_centroids(dataSet,idx,K,oldCentroids):
m,n = dataSet.shape
centroids = np.zeros((K, n))
for k in range(K):
# 提取属于同一类的样本
points = np.array(dataSet[np.where(idx == k)])
# 计算质心
centroids[k] = points.mean(axis=0) # 计算每一列的均值
# 计算变化量
changed = centroids - oldCentroids
return centroids,changed步骤4:组合K-mean各模块
# 组合K—mean
def run_Kmean(dataSet,K,max_iters = 100):
m,n = dataSet.shape
idx = []
# 初始化质点
centroids = init_centroid(dataSet,K)
for i in range(max_iters):
# 寻找最近质点(分组)
idx = find_closed_centroid(dataSet,centroids)
# 计算新质点
centroids,changed = compute_centroids(dataSet,idx,K,centroids)
if np.all(changed == 0):
break
return idx绘图
# 绘图
def Kmean_plot(dataSet,idx,K):
for k in range(K):
# 提取属于同一类的样本
points = np.array(dataSet[np.where(idx == k)])
plt.scatter(points[:, 0], points[:, 1])
plt.show()主函数(含数据标准化)
if __name__ == '__main__':
dataSet = np.load('ex7_X.npy')
# K-mean需要数据标准化
scaler = StandardScaler()
dataSet = scaler.fit_transform(dataSet)
K = 3
classification = run_Kmean(dataSet,K)
Kmean_plot(dataSet,classification,K)运行结果:
使用K-mean进行图像压缩
现在用K-mean来做一些有趣的事情吧!
原始图像:
我们需要将这张图像变为只有8个颜色构成的图像。
数据预处理
需要将图片转化为2维矩阵,这张图是![]() 的,需要变为
的,需要变为![]() 的矩阵,并且需要归一化。
的矩阵,并且需要归一化。
original_img = plt.imread('bird_small.png')
# 归一化
original_img/=255
# 将图像转化为m*3的二维矩阵,m为图像像素,这里m应为128*128。
X_img = np.reshape(original_img, (original_img.shape[0] * original_img.shape[1], 3))
# print(X_img.shape)压缩图像
需要在函数run_Kmean的返回值里多加一个centroids,其余代码不变。
K = 8
# max_iters = 10
idx,centroids = run_Kmean(X_img,K)
# 现在,将每一个像素覆盖为它所属的簇质心的值
X_recovered = centroids[idx]
X_recovered = np.reshape(X_recovered, original_img.shape)绘制图像
# Display original image
fig, ax = plt.subplots(1, 2, figsize=(8, 8))
plt.axis('off')
ax[0].imshow(original_img * 255)
ax[0].set_title('Original')
ax[0].set_axis_off() # 不显示坐标轴
# Display compressed image
ax[1].imshow(X_recovered * 255)
ax[1].set_title('Compressed with %d colours' % K)
ax[1].set_axis_off()
plt.show()结果:
使用sklearn实现K-mean
网上随便找了张图片,来压缩吧!
写代码时发现一个很奇怪的问题,这张图它居然是4个图层的,变为二维数组时需要注意一下,不是变成m*3 而是m*4 。(后来发现只要是Q截图的都是4图层的。)
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
if __name__ == '__main__':
original_img = plt.imread('color.png')
print("Shape of original_img is:", original_img.shape)
original_img /= 255
X_img = np.reshape(original_img, (original_img.shape[0] * original_img.shape[1], 4))
# 模型训练
K = 8
model = KMeans(n_clusters=K)
model.fit(X_img)
centroids = model.cluster_centers_
# labels得到的是质心索引
labels = model.predict(X_img)
# print(labels[:6])
# 替换样本
X_recovered = centroids[labels]
X_recovered = np.reshape(X_recovered, original_img.shape)
plt.imshow(X_recovered*255)
plt.axis('off')
plt.show()压缩后:
K-mean算法的缺点
-
算法中的K是事先给定的,但是K的选择在许多时候是难以估计的,很多时候不知道数据集应该分为多少类最合适。
-
K-mean初始质心选择如果选的不好,有可能无法得到有效的聚类结果。可能会局部收敛而非全局收敛。为了解决该问题,提出了二分K-mean算法,实验表明二分K-mean的聚类效果要好于普通的K-mean算法。
-
数据集较大时收敛较慢。
-
如果遇到非球状的数据,K-mean算法不适用。
该图来源:K-means聚类算法原理及python实现