- MCP-Proxy:开发多LLM & 多MCP 支持并安全访问MCP Server的秘密
IT古董
技术杂谈安全MCPMCP-Proxy
在构建多模型、多协议、可控可信的大模型接入平台时,MCP-Proxy扮演着关键中枢。它不仅要支持多个LLM接入,还要保障对后端MCPServer的安全访问、请求审计、能力切换与资源隔离。什么是MCP/MCP-Proxy?MCP(ModelCapabilityProtocol)是新一代模型能力调用协议,类似于OpenAI的API,但可支持:多厂商大模型(OpenAI、DeepSeek、Yi、Chat
- Linux I/O 文件操作详解:从系统调用到实际工程应用
平凡灵感码头
linux学习linux运维服务器
一、写在前面在Linux或任何类Unix操作系统中,文件是一切的核心——无论是硬盘上的文本文件,还是串口设备、GPIO寄存器、甚至网络接口,几乎都被抽象为“文件”。理解Linux下的I/O文件操作机制,不仅是嵌入式开发的基础,也是进行系统编程与底层控制的关键。二、I/O的本质:一切皆文件Linux将外设抽象成文件的方式,统一了对各种资源的操作模型。你可以用open打开串口设备/dev/ttyS0,
- 构建医学文献智能助手:基于 LangChain 的专业领域 RAG 系统实践
前言在当今医疗科技快速发展的时代,每天都有数以千计的医学研究成果在全球范围内发表。从临床试验报告到基础研究论文,从流行病学调查到药物研发数据,这些专业文献承载着推动医学进步的重要知识。然而,面对如此海量且专业性极强的文献资料,医疗从业者往往感到力不从心。如何在有限的时间内,准确把握文献核心价值,并将其转化为临床实践的指导?这个问题一直困扰着整个医疗行业。1.项目背景与业务价值1.1医学文献阅读的困
- JVM(9)——详解Serial垃圾回收器
十六点五
jvmjava开发语言后端
Serial垃圾回收器是JVM最古老、最基础、最简单的垃圾回收器,也是理解其他更复杂回收器的基础。一、Serial回收器的定位与设计目标核心特点:单线程(Single-Threaded)这是Serial回收器最根本的特征。无论是进行垃圾标记(Marking)、清除(Sweeping)、复制(Copying)还是整理(Compacting),它都只使用一个单独的线程来执行所有垃圾回收工作。工作模式:
- Spring AI快速入门
学java的cc
spring大数据java
一、引入依赖org.springframework.aispring-ai-starter-model-openaiorg.springframework.aispring-ai-bom${spring-ai.version}pomimport二、配置模型spring:ai:openai:base-url:https://dashscope.aliyuncs.com/compatible-mode
- Python爬虫实战:全方位爬取知乎学习板块问答数据
Python爬虫项目
2025年爬虫实战项目python爬虫学习开发语言scrapy游戏
1.项目背景与爬取目标知乎是中国最大的知识问答社区,聚集了大量高质量的学习资源和经验分享。爬取知乎“学习”板块的问答数据,可以为学习资料整理、舆情分析、推荐系统开发等提供数据支持。本项目目标:爬取“学习”话题下的热门问答列表抓取每个问答的标题、作者、回答内容、点赞数、评论数等详细信息实现动态加载内容的抓取,包含图片和富文本避免被反爬机制限制,保证数据采集稳定结合数据分析,为后续应用打基础2.知乎“
- Python爬虫实战:爬取知乎问答与用户信息
Python爬虫项目
python爬虫php数据分析开发语言开源
简介随着网络信息量的爆炸,如何有效获取有价值的内容,成为了数据分析、机器学习等领域的基础之一。爬虫作为数据采集的基本工具之一,常常被用来获取互联网上的公开数据。在这篇博客中,我们将结合最新的Python爬虫技术,详细讲解如何爬取知乎问答与用户信息。本文将会介绍:Python爬虫的基础知识知乎问答网页结构分析使用Python进行知乎数据爬取爬取知乎问答内容与用户信息如何处理和存储爬取的数据使用最新的
- 【机器学习&深度学习】反向传播机制
目录一、一句话定义二、类比理解三、为什重要?四、用生活例子解释:神经网络=烹饪机器人4.1第一步:尝一口(前向传播)4.2第二步:倒着推原因(反向传播)五、换成人工智能流程说一遍六、图示类比:找山顶(最优参数)七、总结一句人话八、PyTorch代码示例:亲眼看到每一层的梯度九、梯度=损失函数对参数的偏导数十、类比总结反向传播(Backpropagation)是神经网络中训练过程的核心机制,它就像“
- 人脸识别算法赋能园区无人超市安防升级
智驱力人工智能
算法人工智能边缘计算人脸识别智慧园区智慧工地智慧煤矿
人脸识别算法赋能园区无人超市安防升级正文在园区无人超市的运营管理中,传统安防手段依赖人工巡检或基础监控设备,存在响应滞后、误报率高、环境适应性差等问题。本文从技术背景、实现路径、功能优势及应用场景四个维度,阐述如何通过人脸识别检测、人员入侵算法及疲劳检测算法的协同应用,构建高效、精准的智能安防体系。一、技术背景:视觉分析算法的核心支撑人脸识别算法基于深度学习的卷积神经网络(CNN)模型,通过提取面
- .NET C# async/定时任务的异步线程池调度方案最大线程数 = 处理器核心数 × 250
专注VB编程开发20年
.netc#开发语言
关于.NET中Threading.Timer的线程机制,结合线程池特性和异步协作原理分析如下:一、线程复用机制共享进程级线程池Threading.Timer的回调任务不会每次新建线程,而是提交到.NET进程全局线程池统一调度,该线程池与async/await任务共享同一资源池。线程池维护可复用工作线程队列,避免频繁创建/销毁开销任务优先由空闲线程执行,无空闲线程则进入全局队列等待线程池扩
- 世赛背景下,中职物联网应用与服务赛项实训解决方案
武汉唯众智创
物联网世赛物联网应用与服务世界职业院校技能大赛技能大赛物联网应用
一、世赛背景与物联网应用赛项概述1.1世赛发展历程及对中职教育的影响世界技能大赛(WorldSkillsCompetition,简称世赛)自1950年创立以来,已经成为全球范围内展示职业技能水平的重要赛事。截至2024年,世赛已成功举办46届,参赛国家和地区数量不断增加,从最初的20多个发展到如今的80多个,参赛选手超过1000名。世赛涵盖了从传统制造业到现代信息技术等众多领域,其中物联网应用与服
- 企业级知识库私有化部署:腾讯混元+云容器服务TKE实战
大熊计算机
#腾讯云语言模型
1.背景需求分析在金融、医疗等数据敏感行业,企业需要构建完全自主可控的知识库系统。本文以某证券机构智能投研系统为原型,演示如何基于腾讯混元大模型与TKE容器服务实现:千亿级参数模型的私有化部署金融领域垂直场景微调高并发低延迟推理服务全链路安全合规方案1.1典型技术挑战#性能基准测试数据(单位:QPS)|场景|裸机部署|容器化部署|优化后||--------------------|--------
- OPC/MQTT工业通信软件OPLink
牛宝柱
OPLink是上海泗博自主研发的基于OPC数据采集及转发,OPC数据到MQTT通信的产品。它是基于上海泗博多年的OPC应用经验和工业通信产品的开发背景,推出的OPC/MQTT工业通信软件。软件设计简洁、实用、稳定。同时,OPLink还具备与KepwareLinkMaster相似的数据转发功能,可实现PLC设备间的自主数据通信。功能升级实时性增强:针对复杂的工业自动化环境,OPLink优化了数据传输
- SnowConvert:自动化数据迁移的技术解析与最佳实践
weixin_30777913
迁移学习数据库运维
SnowConvert是Snowflake生态系统的关键迁移工具,专为将传统数据仓库(如Oracle、Teradata、SQLServer等)的代码资产高效、准确地转换为Snowflake原生语法而设计。以下基于官方文档对其技术原理、工作流程及最佳实践进行深入分析:一、SnowConvert核心技术解析精准的语法映射引擎语言支持:深度解析源系统特有语法(OraclePL/SQL,TeradataB
- GitHub Actions与AWS OIDC实现安全的ECR/ECS自动化部署
ivwdcwso
运维与云原生githubaws安全ecrecsoldcCI/CD
引言在现代云原生应用开发中,实现安全、高效的CI/CD流程至关重要。本文将详细介绍如何利用GitHubActions和AWSOIDC(OpenIDConnect)构建一个无需长期凭证的安全部署管道,将容器化应用自动部署到AmazonECR和ECS服务。架构概述整个解决方案的架构包含三个主要部分:GitHub端:代码仓库和GitHubActions工作流AWS端:OIDC身份验证、ECR容器仓库和E
- ArkTS 开发学习路径全攻略:从入门到实战
码农乐园
学习
随着HarmonyOS的持续演进,ArkTS(ArkTypeScript)已成为鸿蒙系统的主力开发语言。特别是HarmonyOSNEXT推行纯鸿蒙化后,ArkTS成为构建鸿蒙原生应用的唯一选择。本文将为你梳理一套系统化的学习路径,从语法基础到实战项目,再到系统能力调用与分布式开发,一步步带你成为合格的鸿蒙开发者。第一阶段:ArkTS语言和HarmonyOS基础入门学习目标:掌握ArkTS基础语法;
- AWS Lambda与RDS连接优化之旅
t0_54manong
编程问题解决手册aws云计算个人开发
在云计算的时代,AWSLambda与RDS的结合为开发者提供了高效且灵活的解决方案。然而,在实际应用中,我们常常会遇到一些性能瓶颈。本文将通过一个真实案例,探讨如何优化AWSLambda与RDS之间的连接,以提高API的响应速度。背景介绍最近,我们在AWS上部署了一个使用Dotnet6开发的API,它通过APIGateway暴露给外部,并连接到同VPC内的MySQLAuroraRDS数据库。部署前
- “自动化失败归因”测试集-Who&When
liliangcsdn
自动化人工智能语言模型
在MAS(Multi-Agent系统)中,Agent之间自主协作、信息链条长,异常常见而且诊断困难。Who&When就是测者MAS系统异常诊断的benchmark。Who&When的prompt问题来源于GAIAandAssistantBench,包含了127个LLMMulti-Agent系统中收集的异常日志,并经过系统和人工处理。Who&When样本配有如下所示的细粒度标注:“谁”(Who):哪
- 从历史到未来:《今日简史》与《原则》的世界格局研究
喝醉酒的小白
破万卷历史
目录标题一、引言:两种视角下的世界格局二、世界观比较:历史演进与系统运行2.1赫拉利的人类中心史观2.2达利欧的系统论世界观2.3世界观的异同与互补三、方法论比较:历史叙事与系统建模3.1赫拉利的历史叙事方法3.2达利欧的系统建模方法3.3方法论的异同与互补四、核心议题比较:科技、经济与全球治理4.1科技变革:颠覆性力量的不同解读4.2经济周期:历史规律的不同阐释4.3全球治理:未来秩序的不同展望
- 【Python】 如何使用.whl文件安装Python包?
civilpy
python开发语言
基本原理在Python的世界中,.whl文件是一种分发格式,它代表“Wheel”。Wheel是一种Python包格式,旨在提供一种快速、可靠且兼容的方式,用于安装Python库。与源代码包相比,Wheel文件是预编译的,这意味着它们已经包含了编译后的扩展模块,这使得安装过程更快,更简单。代码示例以下是使用.whl文件安装Python包的示例步骤:示例1:基本安装假设你已经下载了一个名为exampl
- AIRIOT物联网低代码平台如何配置MQTT驱动?
AIRIOT
网络服务器物联网
MQTT驱动配置简介MQTT全称为消息队列遥测传输(英语:MessageQueuingTelemetryTransport),是ISO标准(ISO/IECPRF20922)下基于发布(Publish)/订阅(Subscribe)范式的消息协议,工作在TCP/IP协议族上。MQTT最大优点在于,可以用极少的数据和有限的带宽,为连接远程设备提供实时可靠的消息服务。作为一种低开销、低带宽占用的即时通讯协
- Redis配置与优化
?ccc?
redis数据库缓存
目录一:Redis介绍1:关系数据库与非关系型数据库2:Redis基础2.1Redis简介2.2Redis安装部署2.3配置参数3:Redis命令工具3.1redis-cli命令行工具3.2redis-benchmark测试工具4:Redis数据库常用命令4.1key相关命令4.2多数据库常用命令二:Redis持久化1:RDB和AOF的区别2:RDB和AOF的优缺点3:Redis持久化配置三:性能
- Shell 编程之正则表达式与文本处理器
目录一:正则表达式二:基础正则表达式1.基础正则表达式示例(1)查找特定字符(2)利用中括号“[]”来查找集合字符(3)查找行首“^”与行尾字符“$”(4)查找任意一个字符“.”与重复字符“*”(5)查找连续字符范围“{}”2.元字符总结3.扩展正则表达式二:文本处理器1.sed工具(1)输出符合条件的文本(p表示正常输出)(2)删除符合条件的文本(d)(3)替换符合条件的文本(4)迁移符合条件的
- 疲劳检测与行为分析:工厂智能化实践
智驱力人工智能
安全智慧城市行为识别人员属性识别疲劳检测抽烟检测徘徊检测
视觉分析算法赋能工厂疲劳与安全管理一、背景与需求在制造业中,疲劳作业是导致安全事故和效率下降的核心因素之一。传统人工巡检存在覆盖面不足、响应滞后等问题,而基于视觉分析的智能监控系统通过多算法协同,可实现全天候、高精度的疲劳检测与行为管理。本文围绕疲劳检测算法、人员计数算法、抽烟检测算法及徘徊检测算法,探讨其在工厂场景中的技术实现与应用价值。二、技术实现疲劳检测算法原理:基于PERCLOS(眼睑闭合
- Nordic智能楼宇自动化系统方案/nrf-knx-iot
Halfway--
Product物联网iot
1:KNXIoT通过物联网(IoT)的强大功能和灵活性扩展了KNX标准的能力。因此,它允许KNX设备与物联网设备和云服务集成,从而能够创建先进的智能楼宇自动化系统。通过KNXIoT,设备可以在IP网络上进行通信,从而在设备连接和控制方式上提供更大的灵活性2:KNXIoT由3个主要负责数据互操作性的主要元素组成:KNXIoT第三方API一个标准化的API,通过一个抽象层连接KNX特定知识和第三方应用
- 鸿蒙 ArkTS 开发知识点全体系(HarmonyOS NEXT 架构)
码农乐园
harmonyos架构华为
一、基础知识:ArkTS语言与项目结构1.ArkTS基础语法(华为增强TypeScript)类型声明与推导函数与箭头函数类、接口、枚举、泛型模块导入与导出装饰器语法(@Entry、@Component等)异步编程(async/await)2.DevEcoStudio开发环境项目创建与构建模拟器配置与真机调试工程结构(entry、pages、resources、common、config.json)
- 【开源项目】「安卓原生3D开源渲染引擎」:Sceneform‑EQR
「安卓原生3D开源渲染引擎」:Sceneform‑EQR渲染引擎“那一夜凌晨3点,第一次提交PR的手在抖……”——我深刻体会这种忐忑与激动。仓库地址:(github.com)。一、前言:开源对我意味着什么DIY的自由Vs.工业化的束缚刚入Android原生开发时,我习惯自己在项目里嵌入各种3D渲染/AR/XR模块,结构臃肿、流程混乱。当我知道GoogleSceneformSDK被弃用,起初只是出于
- GPT-4o重磅升级!只需一条指令,教你秒出SCI级专业科研图!
智写AI
AI学术写作指南信息可视化人工智能
经过数月爆肝,七哥终于完成专业的学术AI使用教程,估计也有个80万字的详细操作指南。分为多个细分的专业写作场景,跟着一步一步操作,借助ChatGPT做学术、干科研、写论文、课题申报都变得超简单。欢迎加我交流(yida985),祝你一臂之力。七哥之前写过关于用AI生成流程图的教程,不过需要借助其他软件才能搞定完美的流程图。近期GPT-4o全新推出了“生图功能”,这个生图的过程就更加方便轻松了,全能G
- RAG 调优指南:Spring AI Alibaba 模块化 RAG 原理与使用
ApacheDubbo
spring人工智能架构SpringAIRAG
>夏冬,SpringAIAlibabaContributorRAG简介什么是RAG(检索增强生成)RAG(RetrievalAugmentedGeneration,检索增强生成)是一种结合信息检索和文本生成的技术范式。核心设计理念RAG技术就像给AI装上了「实时百科大脑」,通过先查资料后回答的机制,让AI摆脱传统模型的"知识遗忘"困境。️四大核心步骤1.文档切割→建立智能档案库核心任务:将海量文档
- Python 数据挖掘实战: 关联规则与聚类分析,解锁数据价值的钥匙
清水白石008
pythonPython题库python数据挖掘动画
Python数据挖掘实战:关联规则与聚类分析,解锁数据价值的钥匙引言在数字化浪潮席卷全球的今天,数据已成为企业和组织最重要的战略资产。海量数据蕴藏着巨大的价值,等待我们去挖掘和发现。数据挖掘(DataMining),作为从海量数据中提取有价值知识和模式的关键技术,正日益受到各行各业的重视。它如同探矿者的火眼金睛,能够穿透数据的迷雾,发现隐藏在背后的规律和趋势,为商业决策、科学研究和社会发展提供强有
- 如何用ruby来写hadoop的mapreduce并生成jar包
wudixiaotie
mapreduce
ruby来写hadoop的mapreduce,我用的方法是rubydoop。怎么配置环境呢:
1.安装rvm:
不说了 网上有
2.安装ruby:
由于我以前是做ruby的,所以习惯性的先安装了ruby,起码调试起来比jruby快多了。
3.安装jruby:
rvm install jruby然后等待安
- java编程思想 -- 访问控制权限
百合不是茶
java访问控制权限单例模式
访问权限是java中一个比较中要的知识点,它规定者什么方法可以访问,什么不可以访问
一:包访问权限;
自定义包:
package com.wj.control;
//包
public class Demo {
//定义一个无参的方法
public void DemoPackage(){
System.out.println("调用
- [生物与医学]请审慎食用小龙虾
comsci
生物
现在的餐馆里面出售的小龙虾,有一些是在野外捕捉的,这些小龙虾身体里面可能带有某些病毒和细菌,人食用以后可能会导致一些疾病,严重的甚至会死亡.....
所以,参加聚餐的时候,最好不要点小龙虾...就吃养殖的猪肉,牛肉,羊肉和鱼,等动物蛋白质
- org.apache.jasper.JasperException: Unable to compile class for JSP:
商人shang
maven2.2jdk1.8
环境: jdk1.8 maven tomcat7-maven-plugin 2.0
原因: tomcat7-maven-plugin 2.0 不知吃 jdk 1.8,换成 tomcat7-maven-plugin 2.2就行,即
<plugin>
- 你的垃圾你处理掉了吗?GC
oloz
GC
前序:本人菜鸟,此文研究学习来自网络,各位牛牛多指教
1.垃圾收集算法的核心思想
Java语言建立了垃圾收集机制,用以跟踪正在使用的对象和发现并回收不再使用(引用)的对象。该机制可以有效防范动态内存分配中可能发生的两个危险:因内存垃圾过多而引发的内存耗尽,以及不恰当的内存释放所造成的内存非法引用。
垃圾收集算法的核心思想是:对虚拟机可用内存空间,即堆空间中的对象进行识别
- shiro 和 SESSSION
杨白白
shiro
shiro 在web项目里默认使用的是web容器提供的session,也就是说shiro使用的session是web容器产生的,并不是自己产生的,在用于非web环境时可用其他来源代替。在web工程启动的时候它就和容器绑定在了一起,这是通过web.xml里面的shiroFilter实现的。通过session.getSession()方法会在浏览器cokkice产生JESSIONID,当关闭浏览器,此
- 移动互联网终端 淘宝客如何实现盈利
小桔子
移動客戶端淘客淘寶App
2012年淘宝联盟平台为站长和淘宝客带来的分成收入突破30亿元,同比增长100%。而来自移动端的分成达1亿元,其中美丽说、蘑菇街、果库、口袋购物等App运营商分成近5000万元。 可以看出,虽然目前阶段PC端对于淘客而言仍旧是盈利的大头,但移动端已经呈现出爆发之势。而且这个势头将随着智能终端(手机,平板)的加速普及而更加迅猛
- wordpress小工具制作
aichenglong
wordpress小工具
wordpress 使用侧边栏的小工具,很方便调整页面结构
小工具的制作过程
1 在自己的主题文件中新建一个文件夹(如widget),在文件夹中创建一个php(AWP_posts-category.php)
小工具是一个类,想侧边栏一样,还得使用代码注册,他才可以再后台使用,基本的代码一层不变
<?php
class AWP_Post_Category extends WP_Wi
- JS微信分享
AILIKES
js
// 所有功能必须包含在 WeixinApi.ready 中进行
WeixinApi.ready(function(Api) {
// 微信分享的数据
var wxData = {
&nb
- 封装探讨
百合不是茶
JAVA面向对象 封装
//封装 属性 方法 将某些东西包装在一起,通过创建对象或使用静态的方法来调用,称为封装;封装其实就是有选择性地公开或隐藏某些信息,它解决了数据的安全性问题,增加代码的可读性和可维护性
在 Aname类中申明三个属性,将其封装在一个类中:通过对象来调用
例如 1:
//属性 将其设为私有
姓名 name 可以公开
- jquery radio/checkbox change事件不能触发的问题
bijian1013
JavaScriptjquery
我想让radio来控制当前我选择的是机动车还是特种车,如下所示:
<html>
<head>
<script src="http://ajax.googleapis.com/ajax/libs/jquery/1.7.1/jquery.min.js" type="text/javascript"><
- AngularJS中安全性措施
bijian1013
JavaScriptAngularJS安全性XSRFJSON漏洞
在使用web应用中,安全性是应该首要考虑的一个问题。AngularJS提供了一些辅助机制,用来防护来自两个常见攻击方向的网络攻击。
一.JSON漏洞
当使用一个GET请求获取JSON数组信息的时候(尤其是当这一信息非常敏感,
- [Maven学习笔记九]Maven发布web项目
bit1129
maven
基于Maven的web项目的标准项目结构
user-project
user-core
user-service
user-web
src
- 【Hive七】Hive用户自定义聚合函数(UDAF)
bit1129
hive
用户自定义聚合函数,用户提供的多个入参通过聚合计算(求和、求最大值、求最小值)得到一个聚合计算结果的函数。
问题:UDF也可以提供输入多个参数然后输出一个结果的运算,比如加法运算add(3,5),add这个UDF需要实现UDF的evaluate方法,那么UDF和UDAF的实质分别究竟是什么?
Double evaluate(Double a, Double b)
- 通过 nginx-lua 给 Nginx 增加 OAuth 支持
ronin47
前言:我们使用Nginx的Lua中间件建立了OAuth2认证和授权层。如果你也有此打算,阅读下面的文档,实现自动化并获得收益。SeatGeek 在过去几年中取得了发展,我们已经积累了不少针对各种任务的不同管理接口。我们通常为新的展示需求创建新模块,比如我们自己的博客、图表等。我们还定期开发内部工具来处理诸如部署、可视化操作及事件处理等事务。在处理这些事务中,我们使用了几个不同的接口来认证:
&n
- 利用tomcat-redis-session-manager做session同步时自定义类对象属性保存不上的解决方法
bsr1983
session
在利用tomcat-redis-session-manager做session同步时,遇到了在session保存一个自定义对象时,修改该对象中的某个属性,session未进行序列化,属性没有被存储到redis中。 在 tomcat-redis-session-manager的github上有如下说明: Session Change Tracking
As noted in the &qu
- 《代码大全》表驱动法-Table Driven Approach-1
bylijinnan
java算法
关于Table Driven Approach的一篇非常好的文章:
http://www.codeproject.com/Articles/42732/Table-driven-Approach
package com.ljn.base;
import java.util.Random;
public class TableDriven {
public
- Sybase封锁原理
chicony
Sybase
昨天在操作Sybase IQ12.7时意外操作造成了数据库表锁定,不能删除被锁定表数据也不能往其中写入数据。由于着急往该表抽入数据,因此立马着手解决该表的解锁问题。 无奈此前没有接触过Sybase IQ12.7这套数据库产品,加之当时已属于下班时间无法求助于支持人员支持,因此只有借助搜索引擎强大的
- java异常处理机制
CrazyMizzz
java
java异常关键字有以下几个,分别为 try catch final throw throws
他们的定义分别为
try: Opening exception-handling statement.
catch: Captures the exception.
finally: Runs its code before terminating
- hive 数据插入DML语法汇总
daizj
hiveDML数据插入
Hive的数据插入DML语法汇总1、Loading files into tables语法:1) LOAD DATA [LOCAL] INPATH 'filepath' [OVERWRITE] INTO TABLE tablename [PARTITION (partcol1=val1, partcol2=val2 ...)]解释:1)、上面命令执行环境为hive客户端环境下: hive>l
- 工厂设计模式
dcj3sjt126com
设计模式
使用设计模式是促进最佳实践和良好设计的好办法。设计模式可以提供针对常见的编程问题的灵活的解决方案。 工厂模式
工厂模式(Factory)允许你在代码执行时实例化对象。它之所以被称为工厂模式是因为它负责“生产”对象。工厂方法的参数是你要生成的对象对应的类名称。
Example #1 调用工厂方法(带参数)
<?phpclass Example{
- mysql字符串查找函数
dcj3sjt126com
mysql
FIND_IN_SET(str,strlist)
假如字符串str 在由N 子链组成的字符串列表strlist 中,则返回值的范围在1到 N 之间。一个字符串列表就是一个由一些被‘,’符号分开的自链组成的字符串。如果第一个参数是一个常数字符串,而第二个是type SET列,则 FIND_IN_SET() 函数被优化,使用比特计算。如果str不在strlist 或st
- jvm内存管理
easterfly
jvm
一、JVM堆内存的划分
分为年轻代和年老代。年轻代又分为三部分:一个eden,两个survivor。
工作过程是这样的:e区空间满了后,执行minor gc,存活下来的对象放入s0, 对s0仍会进行minor gc,存活下来的的对象放入s1中,对s1同样执行minor gc,依旧存活的对象就放入年老代中;
年老代满了之后会执行major gc,这个是stop the word模式,执行
- CentOS-6.3安装配置JDK-8
gengzg
centos
JAVA_HOME=/usr/java/jdk1.8.0_45
JRE_HOME=/usr/java/jdk1.8.0_45/jre
PATH=$PATH:$JAVA_HOME/bin:$JRE_HOME/bin
CLASSPATH=.:$JAVA_HOME/lib/dt.jar:$JAVA_HOME/lib/tools.jar:$JRE_HOME/lib
export JAVA_HOME
- 【转】关于web路径的获取方法
huangyc1210
Web路径
假定你的web application 名称为news,你在浏览器中输入请求路径: http://localhost:8080/news/main/list.jsp 则执行下面向行代码后打印出如下结果: 1、 System.out.println(request.getContextPath()); //可返回站点的根路径。也就是项
- php里获取第一个中文首字母并排序
远去的渡口
数据结构PHP
很久没来更新博客了,还是觉得工作需要多总结的好。今天来更新一个自己认为比较有成就的问题吧。 最近在做储值结算,需求里结算首页需要按门店的首字母A-Z排序。我的数据结构原本是这样的:
Array
(
[0] => Array
(
[sid] => 2885842
[recetcstoredpay] =&g
- java内部类
hm4123660
java内部类匿名内部类成员内部类方法内部类
在Java中,可以将一个类定义在另一个类里面或者一个方法里面,这样的类称为内部类。内部类仍然是一个独立的类,在编译之后内部类会被编译成独立的.class文件,但是前面冠以外部类的类名和$符号。内部类可以间接解决多继承问题,可以使用内部类继承一个类,外部类继承一个类,实现多继承。
&nb
- Caused by: java.lang.IncompatibleClassChangeError: class org.hibernate.cfg.Exten
zhb8015
maven pom.xml关于hibernate的配置和异常信息如下,查了好多资料,问题还是没有解决。只知道是包冲突,就是不知道是哪个包....遇到这个问题的分享下是怎么解决的。。
maven pom:
<dependency>
<groupId>org.hibernate</groupId>
<ar
- Spark 性能相关参数配置详解-任务调度篇
Stark_Summer
sparkcachecpu任务调度yarn
随着Spark的逐渐成熟完善, 越来越多的可配置参数被添加到Spark中来, 本文试图通过阐述这其中部分参数的工作原理和配置思路, 和大家一起探讨一下如何根据实际场合对Spark进行配置优化。
由于篇幅较长,所以在这里分篇组织,如果要看最新完整的网页版内容,可以戳这里:http://spark-config.readthedocs.org/,主要是便
- css3滤镜
wangkeheng
htmlcss
经常看到一些网站的底部有一些灰色的图标,鼠标移入的时候会变亮,开始以为是js操作src或者bg呢,搜索了一下,发现了一个更好的方法:通过css3的滤镜方法。
html代码:
<a href='' class='icon'><img src='utv.jpg' /></a>
css代码:
.icon{-webkit-filter: graysc
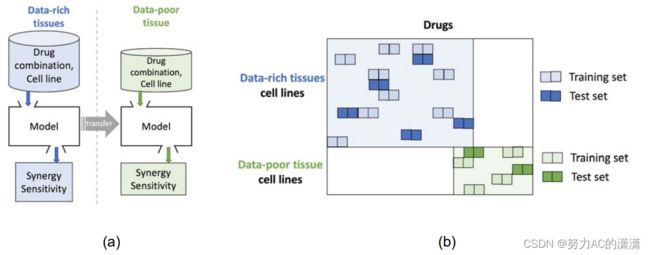
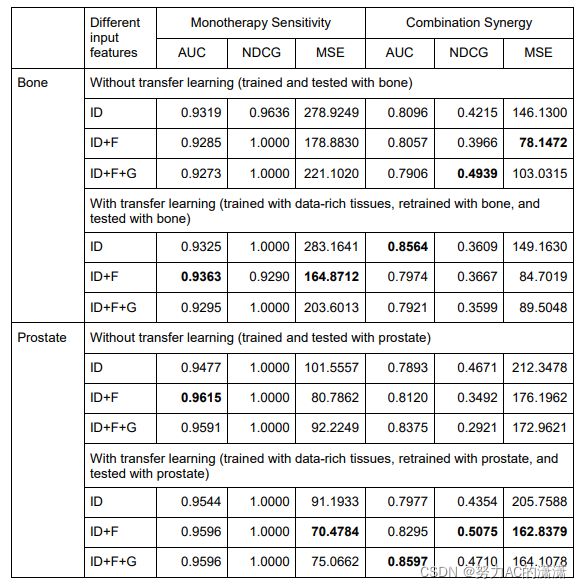
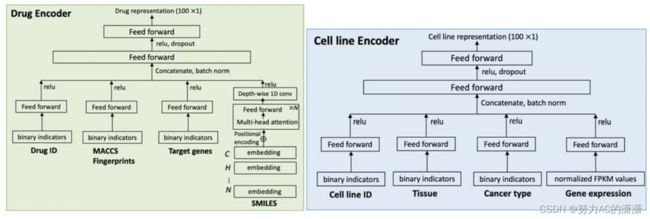
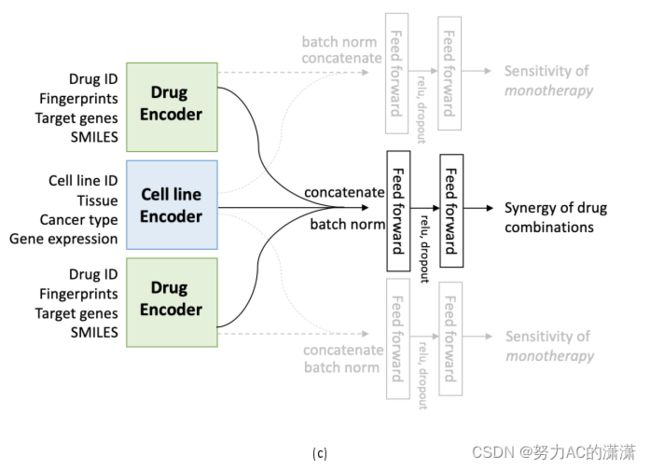
![将其表述为一个预测问题。 给定一个实验块[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-fdFTtCmM-1643093400306)(E:\DBLab\MyNote\pictures\image-20220125134238828.png)]药物组合[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-tBiJ1rpi-1643093400318)(E:\DBLab\MyNote\pictures\image-20220125134253435.png)] 和细胞系[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-C6OrjkgE-1643093400322)(E:\DBLab\MyNote\pictures\image-20220125134302151.png)], 预测模型是一个函数 [外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-86U2w60n-1643093400335)(E:\DBLab\MyNote\pictures\image-20220125134310997.png)]【联合用药协同评分】](http://img.e-com-net.com/image/info8/e7c19ee80509402cab71f6475c384f5c.jpg)
