geo差异表达分析_GEO实操|limma分析差异基因
 欢迎关注医科研公众号,这里是白介素2的读书笔记:)跟我一起聊临床与科研的故事, 生物医学数据挖掘,R语言,TCGA、GEO,SEER数据挖掘。点 这里 跳转到原文,关注我藕~
欢迎关注医科研公众号,这里是白介素2的读书笔记:)跟我一起聊临床与科研的故事, 生物医学数据挖掘,R语言,TCGA、GEO,SEER数据挖掘。点 这里 跳转到原文,关注我藕~
---limma分析差异基因---
在经过了前两期中的数据下载,数据基本处理之后,解决了一个探针对应多个基因数的以及多个探针对应一个基因求平均值,在此基础上运用limma包分析差异基因;
除此以外,包括绘制火山图,热图,PCA等,都在本文中解决
数据载入
1if(T){
2Sys.setlocale(\'LC_ALL\',\'C\')
3library(dplyr)
4##
5if(T){
6load("expma.Rdata")
7load("probe.Rdata")
8}
9expma[1:5,1:5]
10boxplot(expma)##看下表达情况
11metdata[1:5,1:5]
12head(probe)
13
14## 查看Gene Symbol是否有重复
15table(duplicated(probe$`Gene Symbol`))##12549 FALSE
16
17## 整合注释信息到表达矩阵
18ID19expma1,function(x){log2(x+1)})
20expma21esetin% probe$ID,] %>% cbind(probe)
22eset[1:5,1:5]
23colnames(eset)
24}
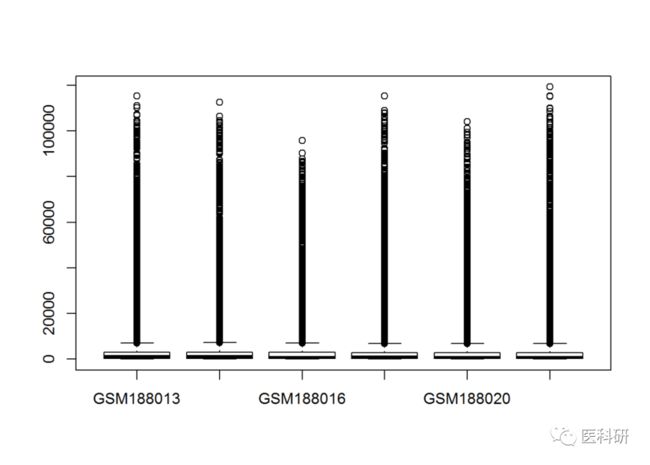 image.png
image.png
1## [1] "GSM188013" "GSM188014" "GSM188016" "GSM188018"
2## [5] "GSM188020" "GSM188022" "ID" "Gene Symbol"
3## [9] "ENTREZ_GENE_ID"
多个探针求平均值
1test1 2test1[1:5,1:5]##与去重结果相吻合
3## Group.1 GSM188013 GSM188014 GSM188016 GSM188018
4## 1 8.438846 8.368513 7.322442 7.813573
5## 2 A1CF 10.979025 10.616926 9.940773 10.413311
6## 3 A2M 6.565276 6.422112 8.142194 5.652593
7## 4 A4GALT 7.728628 7.818966 8.679885 7.048563
8## 5 A4GNT 10.243388 10.182382 9.391991 8.779887
9dim(test1)##
10## [1] 12549 7
11colnames(test1)
12## [1] "Group.1" "GSM188013" "GSM188014" "GSM188016" "GSM188018" "GSM188020"
13## [7] "GSM188022"
14rownames(test1)15test116eset_datPCA plot
Principal Component Analysis (PCA)分析使用的是基于R语言的 prcomp() and princomp()函数.
完成PCA分析一般有两种方法:princomp()使用的是一种的spectral decomposition方法
prcomp() and PCA()[FactoMineR]使用的是SVD法
1data 2data[1:5,1:5]##表达矩阵
3## GSM188013 GSM188014 GSM188016 GSM188018 GSM188020
4## 8.438846 8.368513 7.322442 7.813573 7.244615
5## A1CF 10.979025 10.616926 9.940773 10.413311 9.743305
6## A2M 6.565276 6.422112 8.142194 5.652593 5.550033
7## A4GALT 7.728628 7.818966 8.679885 7.048563 5.929258
8## A4GNT 10.243388 10.182382 9.391991 8.779887 9.431585
9metdata[1:5,1:5]
10## title
11## GSM188013 DMSO treated MCF7 breast cancer cells [HG-U133A] Exp 1
12## GSM188014 Dioxin treated MCF7 breast cancer cells [HG-U133A] Exp 1
13## GSM188016 DMSO treated MCF7 breast cancer cells [HG-U133A] Exp 2
14## GSM188018 Dioxin treated MCF7 breast cancer cells [HG-U133A] Exp 2
15## GSM188020 DMSO treated MCF7 breast cancer cells [HG-U133A] Exp 3
16## geo_accession status submission_date
17## GSM188013 GSM188013 Public on May 12 2007 May 08 2007
18## GSM188014 GSM188014 Public on May 12 2007 May 08 2007
19## GSM188016 GSM188016 Public on May 12 2007 May 08 2007
20## GSM188018 GSM188018 Public on May 12 2007 May 08 2007
21## GSM188020 GSM188020 Public on May 12 2007 May 08 2007
22## last_update_date
23## GSM188013 May 11 2007
24## GSM188014 May 11 2007
25## GSM188016 May 11 2007
26## GSM188018 May 11 2007
27## GSM188020 May 11 2007
28##构建group_list
29group_list30colnames(data)31library(factoextra)
32## Warning: package \'factoextra\' was built under R version 3.5.3
33## Loading required package: ggplot2
34## Welcome! Related Books: `Practical Guide To Cluster Analysis in R` at https://goo.gl/13EFCZ
35## 计算PCA
36data37data38res.pca 39##展示主成分对差异的贡献
40fviz_eig(res.pca)
 Fig2
Fig2
1## 可视化结果
2fviz_pca_ind(res.pca,
3 col.ind = group_list, # 颜色对应group信息
4 palette = c("#00AFBB", "#FC4E07"),
5 addEllipses = TRUE, # Concentration ellipses
6 ellipse.type = "confidence",
7 legend.title = "Group",## Legend名称
8 repel = TRUE
9 )
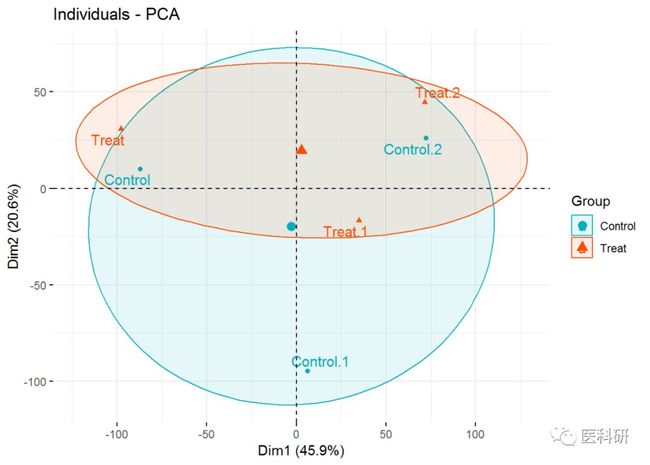 Fig3
Fig3
层次聚类图-聚类结果也与PCA相似,结果并不好
聚类分析的结果也同样可以进一步美化,但这里不做
计算距离时同样需进行转置,但在前一步PCA分析中的data已经经过转置,故未重复
1dd "euclidean")##data是经过行列转换的
2hc "ward.D2")
3plot(hc)
 Fig4
Fig4
1##对结果进行美化
2# Convert hclust into a dendrogram and plot
3hcd as.dendrogram(hc)
4# Define nodePar
5nodePar list(lab.cex = 0.6, pch = c(NA, 19),
6 cex = 0.7, col = "blue")
7# Customized plot; remove labels
8plot(hcd, ylab = "Height", nodePar = nodePar, leaflab = "none")
 Fig5
Fig5
limma分析差异基因
limma包的具体用法参考 limma Users Guide
构建分组信息,构建好比较矩阵是关键
注意这里的表达矩阵信息 eset_dat是经过处理后的,为转置,行为gene,列为sample
1library(limma)
2library(dplyr)
3group_list
4## [1] "Treat" "Control" "Treat" "Control" "Treat" "Control"
5design 6colnames(design)=levels(factor(group_list))
7design
8## Control Treat
9## 1 0 1
10## 2 1 0
11## 3 0 1
12## 4 1 0
13## 5 0 1
14## 6 1 0
15## attr(,"assign")
16## [1] 1 1
17## attr(,"contrasts")
18## attr(,"contrasts")$`factor(group_list)`
19## [1] "contr.treatment"
20## 比较信息
21contrast.matrix22 levels = design)
23contrast.matrix##查看比较矩阵的信息,这里我们设置的是Treat Vs. control
24## Contrasts
25## Levels Treat-Control
26## Control -1
27## Treat 1
28## 拟合模型
29fit 30fit2 31fit2 32DEG% na.omit() ## coef比较分组 n基因数
33head(DEG)
34## logFC AveExpr t P.Value adj.P.Val B
35## ALDH3A1 -3.227263 10.302323 -10.710306 4.482850e-05 0.3134585 -4.048355
36## CYP1B1 -3.033684 13.287607 -10.505888 4.995753e-05 0.3134585 -4.049713
37## CYP1A1 -9.003353 11.481268 -8.371476 1.762905e-04 0.7374232 -4.069681
38## HHLA2 -1.550587 6.595658 -7.443431 3.337672e-04 0.9308066 -4.083411
39## SLC7A5 -2.470333 13.628775 -7.298868 3.708688e-04 0.9308066 -4.085966
40## TIPARP -1.581274 12.764218 -7.024252 4.552834e-04 0.9522252 -4.091192
41dim(DEG)
42## [1] 12549 6
43save(DEG,file = "DEG_all.Rdata")
绘制火山图
火山图其实仅仅是一种可视化的方式,能够从整体上让我们对整体的差异分析情况有个了解
筛选到差异基因后,可以直接绘制出火山图
火山图的横坐标为logFC, 纵坐标为-log10(pvalue),因此其实理论上讲plot即可完成火山图绘制
最简单原始的画法
1colnames(DEG)
2## [1] "logFC" "AveExpr" "t" "P.Value" "adj.P.Val" "B"
3plot(DEG$logFC,-log10(DEG$P.Value))
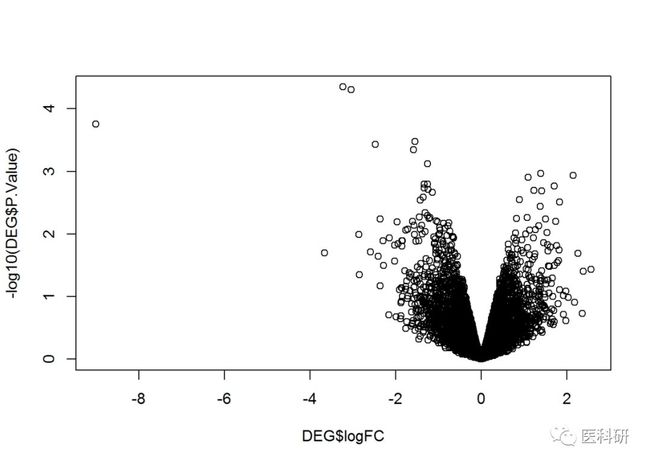 Fig6
Fig6
高级的画法
借助于有人开发的更高级的包,用于完成某些特殊的功能,或者更美观
1require(EnhancedVolcano)
2EnhancedVolcano(DEG,
3
4 lab = rownames(DEG),
5
6 x = "logFC",
7
8 y = "P.Value",
9
10 selectLab = rownames(DEG)[1:5],
11
12 xlab = bquote(~Log[2]~ "fold change"),
13
14 ylab = bquote(~-Log[10]~italic(P)),
15
16 pCutoff = 0.05,## pvalue阈值
17
18 FCcutoff = 1,## FC cutoff
19
20 xlim = c(-5,5),
21
22 transcriptPointSize = 1.8,
23
24 transcriptLabSize = 5.0,
25
26 colAlpha = 1,
27
28 legend=c("NS","Log2 FC"," p-value",
29 " p-value & Log2 FC"),
30
31 legendPosition = "bottom",
32
33 legendLabSize = 10,
34
35 legendIconSize = 3.0)
 Fig7
Fig7
绘制热图
热图的使用比较频繁,得到差异基因后可以直接绘制热图
相对简单好用的要属pheatmap包了
管道中的常规提取需要加上特殊的占位符.
1## 首先提取出想要画的数据
2head(DEG)
3## 提取FC前50
4up_50% as_tibble() %>%
5 mutate(genename=rownames(DEG)) %>%
6 dplyr::arrange(desc(logFC)) %>%
7 .$genename %>% .[1:50] ## 管道符中的提取
8## FC低前50
9down_50% as_tibble() %>%
10 mutate(genename=rownames(DEG)) %>%
11 dplyr::arrange(logFC) %>%
12 .$genename %>% .[1:50] ## 管道符中的提取
13index0,down_50)
14
15## 开始绘图-最简单的图
16library(pheatmap)
17pheatmap(eset_dat[index,],show_colnames =F,show_rownames = F)
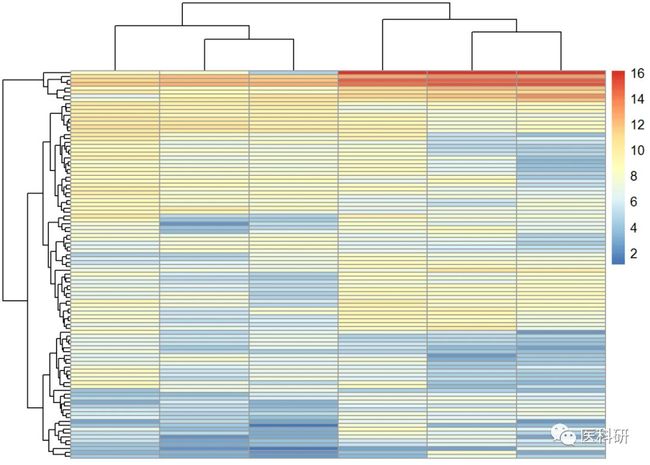 Fig8
Fig8
稍微调整细节
1index_matrix<-t(scale(t(eset_dat[index,])))##归一化 2index_matrix[index_matrix>1]=1
3index_matrix[index_matrix<-1]=-1 4head(index_matrix) 5 6## 添加注释 7anno=data.frame(group=group_list) 8rownames(anno)=colnames(index_matrix) 9anno##注释信息的数据框10pheatmap(index_matrix,11 show_colnames =F,12 show_rownames = F,13 cluster_cols = T, 14 annotation_col=anno)15
 Fig9
Fig9
本期内容就到这里,我是白介素2,下期再见
相关阅读
R语言GEO数据挖掘01-数据下载及提取表达矩阵
R语言ggplot2绘制箱线图
R语言-相关系数计算
文献精读| 28分的临床预测模型文章是什么水平
文献精读|基于SEER数据库的回顾性分析
GDCRNATools|ceRNA套路终结者Part1
TCGAbiolinks下载TCGA数据
R语言-临床三线表
SurvivalROC包绘制时间依赖的ROC曲线
R语言绘制ROC曲线
ggpubr-专为学术绘图而生
美图神器ggstatsplot-专为学术论文而生
R语言ggboxplot-一文掌握箱线图绘制所有细节
https://www.jianshu.com/p/d8e002de9fc3复制链接到浏览器打开可达我的简书博客。
本期的内容就到这里,我是老朋友白介素2,下期再见。
扫码关注我
你来或不来,我就在那里
作者:白介素2
往期系列推送:
想学R?从看R界传奇老司机的免费讲座开始吧
R语言概述
R入门教程之变量
专题 | R语言基础篇—列表,因子和数据框
专题 | R语言基础篇—矩阵
生存分析——R语言
“R”语言基础列之(一)——学渣也能搞定高大上的“R”语言啦
R语言基础系列之 (二) ————给我一个爱上你的理由
R软件基本操作,第三课 |【小白学R系列】
第四课~!安装 |【小白学R系列】
第五课 Packages |【小白学R系列】
第六课 R的内置数据集 |【小白学R系列】
第七课 送ncRNA数据库偶!|【小白学R系列】
第八课 R语言数据结构 |【小白学R系列】
第九课 向量的索引 |【小白学R系列】
第十课 向量的计算 |【小白学R系列】
第十一课 矩阵 |【小白学R系列】
第十二课 数组 |【小白学R系列】
第十三课 列表 |【小白学R系列】
第十四课 数据框 |【小白学R系列】
第十五课 因子 |【小白学R系列】
第十六课 缺失数据 |【小白学R系列】
第十七课 字符串 |【小白学R系列】
第十八课 时间日期处理 |【小白学R系列】
发送生信到本公众号(freescience联盟)后台,查看更多R系列推文~

欢迎各位亲来稿分享学习心得、笔记~
来稿请发至邮箱:[email protected]
1) 稿件必须为原创,如有抄袭,导致法律纠纷由投稿人承担;
2) 字数:字数无要求,漫画形式也可;
3) 内容:生信方法、科研技巧、科研经验、实验方法,临床案例等,只要对科研学习有帮助,都在推送范围内。
注意:没有稿费,没有稿费,没有稿费……
本公众号暂时没有任何收入,推送均为编编组业余时间的分享ღ
Freescience联盟QQ交流群:321950419
(点 这里 和 这里 认识我们哦~)
点 这里 领我们整理的软件库
点 这里 查看sci文章润色服务
点 这里 看R界传奇老司机直播录像
点 这里 进免费免安装的文献下载神器
点本文左下角的 阅读原文,手机端可关键词检索历史推送资料

