无信息变量选择(UVE)波长筛选算法--基于OpenSA开源库实现
系列文章目录
“光晰本质,谱见不同”,光谱作为物质的指纹,被广泛应用于成分分析中。伴随微型光谱仪/光谱成像仪的发展与普及,基于光谱的分析技术将不只停留于工业和实验室,即将走入生活,实现万物感知,见微知著。本系列文章致力于光谱分析技术的科普和应用。
文章目录
- 系列文章目录
- 前言
- 一、光谱数据读入
-
- 1.1 光谱数据读入
- 1.2 光谱可视化
- 二、光谱波长筛选
-
- 2.1 光谱预处理模块
- 2 .2 光谱波长筛选的使用
- 总结
前言
典型的光谱分析模型(以近红外光谱作为示意,可见光、中远红外、荧光、拉曼、高光谱等分析流程亦相似)建立流程如下所示,在建立过程中,需要使用算法对训练样本进行选择,然后使用预处理算法对光谱进行预处理,或对光谱的特征进行提取,再构建校正模型实现定量分析,最后针对不同测量仪器或环境,进行模型转移或传递。因此训练样本的选择、光谱的预处理、波长筛选、校正模型、模型传递以及上述算法的参数都影响着模型的应用效果。
![]()
针对光谱分析流程所涉及的常见的训练样本的划分、光谱的预处理、波长筛选、校正模型算法建立了完整的算法库,名为OpenSA(OpenSpectrumAnalysis)。整套算法库的架构如下所示。
![]()
样本划分模块提供随机划分、SPXY划分、KS划分三种数据集划分方法,光谱预处理模块提供常见光谱预处理,波长筛选模块提供Spa、Cars、Lars、Uve、Pca等特征降维方法,分析模块由光谱相似度计算、聚类、分类(定性分析)、回归(定量分析)构建,光谱相似度子模块计算提供SAM、SID、MSSIM、MPSNR等相似计算方法,聚类子模块提供KMeans、FCM等聚类方法,分类子模块提供ANN、SVM、PLS_DA、RF等经典化学计量学方法,亦提供CNN、AE、Transformer等前沿深度学习方法,回归子模块提供ANN、SVR、PLS等经典化学计量学定量分析方法,亦提供CNN、AE、Transformer等前沿深度学习定量分析方法。模型评估模块提供常见的评价指标,用于模型评估。自动参数优化模块用于自动进行最佳的模型设置参数寻找,提供网格搜索、遗传算法、贝叶斯概率三种最优参数寻找方法。可视化模块提供全程的分析可视化,可为科研绘图,模型选择提供视觉信息。可通过几行代码快速实现完整的光谱分析及应用(注: 自动参数优化模块和可视化模块暂不开源,等毕业后再说)
本篇针对OpenSA的光谱波长筛选模块进行代码开源和使用示意。
一、光谱数据读入
提供两个开源数据作为实列,一个为公开定量分析数据集,一个为公开定性分析数据集,本章仅以公开定量分析数据集作为演示。
1.1 光谱数据读入
# 分别使用一个回归、一个分类的公开数据集做为example
def LoadNirtest(type):
if type == "Rgs":
CDataPath1 = './/Data//Rgs//Cdata1.csv'
VDataPath1 = './/Data//Rgs//Vdata1.csv'
TDataPath1 = './/Data//Rgs//Tdata1.csv'
Cdata1 = np.loadtxt(open(CDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0)
Vdata1 = np.loadtxt(open(VDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0)
Tdata1 = np.loadtxt(open(TDataPath1, 'rb'), dtype=np.float64, delimiter=',', skiprows=0)
Nirdata1 = np.concatenate((Cdata1, Vdata1))
Nirdata = np.concatenate((Nirdata1, Tdata1))
data = Nirdata[:, :-4]
label = Nirdata[:, -1]
elif type == "Cls":
path = './/Data//Cls//table.csv'
Nirdata = np.loadtxt(open(path, 'rb'), dtype=np.float64, delimiter=',', skiprows=0)
data = Nirdata[:, :-1]
label = Nirdata[:, -1]
return data, label
1.2 光谱可视化
#载入原始数据并可视化
data, label = LoadNirtest('Rgs')
plotspc(data, "raw specturm")
采用的开源光谱如图所示:
![]()
二、光谱波长筛选
2.1 光谱预处理模块
提供常见的UVE、Cars、SPA、LARS、PCA等波长筛选算法,并进行了相应的封装,使用者仅需要改变名字,即可选择对应的光谱波长筛选算法,满足论文快速复现、算法快速建模,下面是光谱筛选模块的核心代码
第一种波长筛选算法,无信息变量选择算法(UVE)
class UVE:
def __init__(self, x, y, ncomp=1, nrep=500, testSize=0.2):
'''
X : 预测变量矩阵
y :标签
ncomp : 结果包含的变量个数
testSize: PLS中划分的数据集
return :波长筛选后的光谱数据
'''
self.x = x
self.y = y
# The number of latent components should not be larger than any dimension size of independent matrix
self.ncomp = min([ncomp, rank(x)])
self.nrep = nrep
self.testSize = testSize
self.criteria = None
self.featureIndex = None
self.featureR2 = np.full(self.x.shape[1], np.nan)
self.selFeature = None
def calcCriteria(self):
PLSCoef = np.zeros((self.nrep, self.x.shape[1]))
ss = ShuffleSplit(n_splits=self.nrep, test_size=self.testSize)
step = 0
for train, test in ss.split(self.x, self.y):
xtrain = self.x[train, :]
ytrain = self.y[train]
plsModel = PLSRegression(min([self.ncomp, rank(xtrain)]))
plsModel.fit(xtrain, ytrain)
PLSCoef[step, :] = plsModel.coef_.T
step += 1
meanCoef = np.mean(PLSCoef, axis=0)
stdCoef = np.std(PLSCoef, axis=0)
self.criteria = meanCoef / stdCoef
def evalCriteria(self, cv=3):
self.featureIndex = np.argsort(-np.abs(self.criteria))
for i in range(self.x.shape[1]):
xi = self.x[:, self.featureIndex[:i + 1]]
if i<self.ncomp:
regModel = LinearRegression()
else:
regModel = PLSRegression(min([self.ncomp, rank(xi)]))
cvScore = cross_val_score(regModel, xi, self.y, cv=cv)
self.featureR2[i] = np.mean(cvScore)
def cutFeature(self, *args):
cuti = np.argmax(self.featureR2)
self.selFeature = self.featureIndex[:cuti+1]
if len(args) != 0:
returnx = list(args)
i = 0
for argi in args:
if argi.shape[1] == self.x.shape[1]:
returnx[i] = argi[:, self.selFeature]
i += 1
return returnx
def SpctrumFeatureSelcet(method, X, y):
"""
:param method: 波长筛选/降维的方法,包括:Cars, Lars, Uve, Spa, Pca
:param X: 光谱数据, shape (n_samples, n_features)
:param y: 光谱数据对应标签:格式:(n_samples,)
:return: X_Feature: 波长筛选/降维后的数据, shape (n_samples, n_features)
y:光谱数据对应的标签, (n_samples,)
"""
if method == "None":
X_Feature = X
if method== "Cars":
Featuresecletidx = CARS_Cloud(X, y)
X_Feature = X[:, Featuresecletidx]
elif method == "Lars":
Featuresecletidx = Lar(X, y)
X_Feature = X[:, Featuresecletidx]
elif method == "Uve":
Uve = UVE(X, y, 7)
Uve.calcCriteria()
Uve.evalCriteria(cv=5)
Featuresecletidx = Uve.cutFeature(X)
X_Feature = Featuresecletidx[0]
elif method == "Spa":
Xcal, Xval, ycal, yval = train_test_split(X, y, test_size=0.2)
Featuresecletidx = SPA().spa(
Xcal= Xcal, ycal=ycal, m_min=8, m_max=50, Xval=Xval, yval=yval, autoscaling=1)
X_Feature = X[:, Featuresecletidx]
elif method == "Pca":
X_Feature = Pca(X)
else:
print("no this method of SpctrumFeatureSelcet!")
return X_Feature, y
2 .2 光谱波长筛选的使用
在example.py文件中,提供了光谱波长筛选模块的使用方法,具体如下,仅需要两行代码即可实现所有常见的光谱预处理。
示意:利用OpenSA实现UVE无信息变量选择进行波长筛选
#载入原始数据并可视化
#载入原始数据并可视化
data, label = LoadNirtest('Rgs')
plotspc(data, "raw specturm")
# #波长特征筛选并可视化
SpectruSelected, y = SpctrumFeatureSelcet('Uve', data, label)
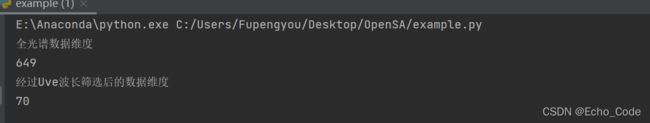
可以看到通过OpenSA一行代码,原始波长655个维度,经过Uve波长筛选后后,得到了70个关键数据点(相关筛选波长维度可以通过改变Uve对应参数修改)
若有问题,可以相互学习探讨
总结
利用OpenSA可以非常简单的实现对光谱的波长筛选,完整代码可从获得GitHub仓库 如果对您有用,请点赞!
代码现仅供学术使用,若对您的学术研究有帮助,请引用本人的论文,同时,未经许可不得用于商业化应用,欢迎大家继续补充OpenSA中所涉及到的算法,如有问题
