多标签分类评价指标HL,RL,OE, Cov的计算及Python实现
AP的计算见本人另一篇博客
HL,RL, OE, Cov是多标签分类的评价指标,通常越小表明模型越好。
一,HL计算
公式:

大意是,有N个样本的数据集,对每个样本的标签集部分,计算真实标签与预测标签部分有多少个不同的部分,
举个栗子:
样本1,y_true = [1, 0, 1, 0, 0],y_pred = [0, 1, 1, 0, 0],可以看到它有2个预测出错的部分,记样本1汉明损失为2/5 = 0.4
样本2,y_true = [1, 0, 1, 0, 1],y_pred = [1, 1, 0, 0, 0],可以看到它有3个预测出错的部分,记样本1汉明损失为3/5 = 0.6
对于总的样本集,汉明损失为(0.4+0.6)/ 2 = 0.5
程序:
import numpy as np
from sklearn import metrics
label = np.array([[1, 0, 1, 0, 0], [1, 0, 1, 0, 1]])
pred = np.array([[0, 1, 1, 0, 0], [1, 1, 0, 0, 0]])
print(metrics.hamming_loss(label, pred))
结果:

二,RL计算
公式:
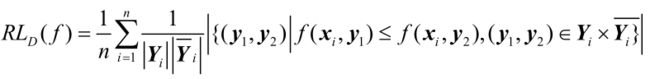
这一大坨我估计很多人第一眼看到就不想看第二眼,

翻译成人话,就是计算标签集中的排序损失。假设数据集有N个样本,对每个样本计算它的标签集中为1的标签的概率小于标签集中为0的概率的标签个数除以标签为1的标签个数乘以标签为0的标签个数。
举个栗子:
样本1,真实标签集y_true = [1, 0, 1, 0, 0],标签预测概率值y_score = [0.3, 0.4, 0.5, 0.1, 0.15],样本1有2个预测为1的标签,即概率为0.3和概率为0.5的标签,一个个来看,对概率为0.3的标签,在整个标签集中,标签为0的标签中,有概率为0.4的标签概率比它大,记为1;对概率为0.5的标签,在整个标签集中,标签为0的标签中,没有标签的概率值比它大,记为0。则样本1的所有标签的排序损失标签为1个,标签为1的标签有2个,标签为0的标签有3个,则基数对有2*3=6个,样本的排序损失为1/6=0.1666667
样本2,真实标签集y_true = [1, 0, 1, 0, 1],标签预测概率值y_score = [0.4, 0.5, 0.7, 0.2, 0.6],样本1有3个预测为1的标签,即概率为0.4、概率为0.7和概率为0.7的标签,一个个来看,对概率为0.4的标签,在整个标签集中,标签为0的标签中,有概率为0.5的标签概率比它大,记为1;对概率为0.7的标签,没有标签的概率值比它大,记为0;对概率为0.6的标签,没有标签的概率值比它大,记为0。则样本1的所有标签的排序损失标签为1个,标签为1的标签有2个,标签为0的标签有3个,则基数对有2*3=6个,样本的排序损失为1/6=0.1666667
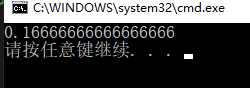
数据集总的排序损失为:(0.16667+0.16667)/ 2 = 0.16667
程序:
label = np.array([[1, 0, 1, 0, 0], [1, 0, 1, 0, 1]])
logit = np.array([[0.3, 0.4, 0.5, 0.1, 0.15], [0.4, 0.5, 0.7, 0.2, 0.6]])
print(metrics.label_ranking_loss(label, logit))
三,OE计算
公式:
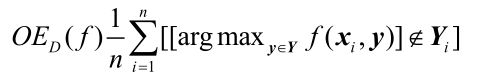
这个就是计算每个样本标签集中概率最大的那个标签在不在标签为1的部分里面,在,记为0,不在,则记为1。就是说最大的那个标签预测的对不对。
举个栗子:
样本1,真实标签集y_true = [1, 0, 1, 0, 0],标签预测概率值y_score = [0.3, 0.4, 0.5, 0.1, 0.15],概率最大的标签对应的真实标签为1,记为0。
样本2,真实标签集y_true = [1, 0, 1, 0, 1],标签预测概率值y_score = [0.4, 0.5, 0.7, 0.2, 0.6],概率最大的标签对应的真实标签为1,记为0。
总的错误率就等于 (0+0)/ 2 = 0
程序:
def One_error(label, logit):
N = len(label)
for i in range(N):
if max(label[i]) == 0:
print("该条数据哪一类都不是")
label_index = []
for i in range(N):
index = np.where(label[i] == 1)[0]
label_index.append(index)
OneError = 0
for i in range(N):
if np.argmax(logit[i]) not in label_index[i]:
OneError += 1
OneError = OneError * 1.0 / N
return OneError
label = np.array([[1, 0, 1, 0, 0], [1, 0, 1, 0, 1]])
logit = np.array([[0.3, 0.4, 0.5, 0.1, 0.15], [0.4, 0.5, 0.7, 0.2, 0.6]])
print(One_error(label, logit))
结果:

四,Cov计算
公式:

这个就是计算每个样本的标签集中,按照概率从大到小排列,到排多少的时候能把标签为1的标签覆盖完毕。为啥要减1,是因为排序是从1开始的。
举个栗子:
样本1,真实标签集y_true = [1, 0, 1, 0, 0],标签预测概率值y_score = [0.3, 0.4, 0.5, 0.1, 0.15],标签从大到小排序,0.5-》0.4-》0.3,排到第三个,把标签为1的全部覆盖了,则样本1的覆盖率为(3-1) = 2
样本2,真实标签集y_true = [1, 0, 1, 0, 1],标签预测概率值y_score = [0.4, 0.5, 0.7, 0.2, 0.6],标签从大到小排序,0.7-》0.6-》0.5-》0.4,排到第四个,把标签为1的全部覆盖了,则样本1的覆盖率为(4-1) = 3
总的覆盖率为 (2+3)/ 2 = 2.5
程序:
def Coverage(label, logit):
D = len(label[0])
N = len(label)
label_index = []
for i in range(N):
index = np.where(label[i] == 1)[0]
label_index.append(index)
cover = 0
for i in range(N):
# 从大到小排序
index = np.argsort(-logit[i]).tolist()
tmp = 0
for item in label_index[i]:
tmp = max(tmp, index.index(item))
cover += tmp
coverage = cover * 1.0 / N
return coverage
label = np.array([[1, 0, 1, 0, 0], [1, 0, 1, 0, 1]])
logit = np.array([[0.3, 0.4, 0.5, 0.1, 0.15], [0.4, 0.5, 0.7, 0.2, 0.6]])
print(Coverage(label, logit))
label = np.array([[1, 0, 1, 0, 0], [1, 0, 1, 0, 1]])
logit = np.array([[0.3, 0.4, 0.5, 0.1, 0.15], [0.4, 0.5, 0.7, 0.2, 0.6]])
print(metrics.coverage_error(label, logit))
print(Coverage(label, logit))
这个coverage方法没有在每个样本排序的时候减去1,导致最终结果如下:

本文的程序代码来自多标签评价指标python代码
如有错误,烦请指正。