生存分析系列教程(一)使用生信人工具盒进行生存分析
生信人工具盒是生信人团队的开发的一款软件,非常方便。下面我将演示一下如何通过这款软件进行生存分析。为了方便大家理解,形式依然是 数据结构-操作-结果解读。
1. 表达矩阵与生存信息矩阵
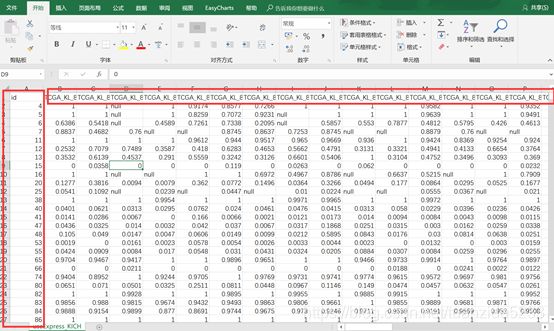
表达矩阵依然是分成两部分,基因名和样本名,分别是行名和列名。其中null数据软件会自动过滤掉。
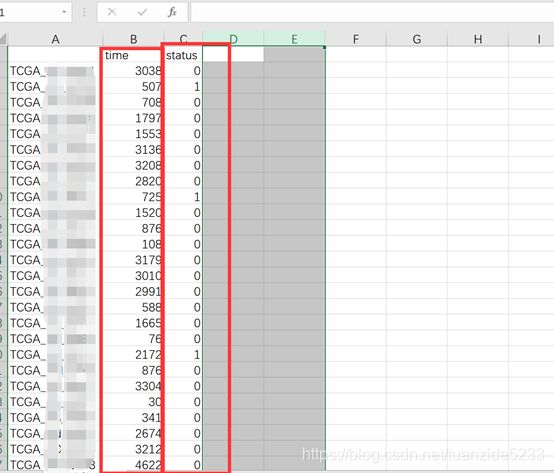
生存信息矩阵,这里包括两部分,生存时间和状态。注意这里生存时间的单位是日,死亡状态是1,生存状态是0.
重要提示!!!两个文件都需要保存成txt格式!
2. 运行生信人工具盒
生信人工具盒下载方式可以加群:244470185
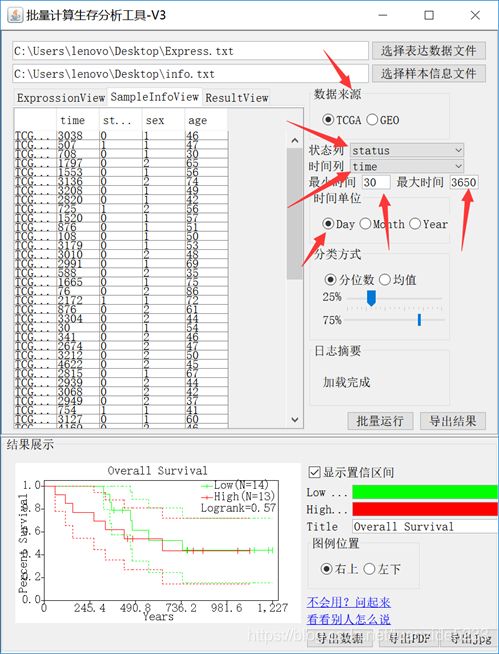
设置好参数点击批量运行,然后导出就可以啦。
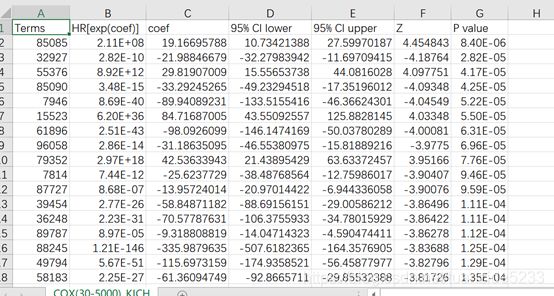
3. 结果解读
输出结果中依次是基因名,HR,coef,95置信区间,Z值和P值。
比较重要的是HR,一般HR越高表明风险越大。
Coef的正负则表示对结果的促进或者抑制。
4. 代码
希望在R语言中重现的小伙伴可以看帖:https://shengxin.ren/article/145
帖子中讲到的对应代码如下:
library(survival)
setwd("D:/Work/code/Test/GSE25065_family.xml")
data=read.csv('CoxResult.txt.matrix',sep = '\t',row.names = 1)
head(data)
time=data[,1]
status=data[,2]
cox1=coxph(Surv(time, status) ~ ACADSB,data)
cox2=coxph(Surv(time, status) ~ GATA3,data)
cox3=coxph(Surv(time, status) ~ CHMP6,data)
cox4=coxph(Surv(time, status) ~ ADCY9,data)GEO芯片数据差异表达分析时需要log2处理的原因
https://blog.csdn.net/tuanzide5233/article/details/88542805
GEO芯片数据差异表达分析时是否需要log2以及标准化的问题
https://blog.csdn.net/tuanzide5233/article/details/88542558
差异表达矩阵制作教程
https://blog.csdn.net/tuanzide5233/article/details/83659768
差异表达的热图绘制详见
https://blog.csdn.net/tuanzide5233/article/details/83659501
使用edgeR对RNAseq数据进行差异表达分析教程
https://blog.csdn.net/tuanzide5233/article/details/88785486
差异表达分析(DEG)时 row.names'里不能有重复的名字 的解决方案
https://blog.csdn.net/tuanzide5233/article/details/86568155
生存分析系列教程(一)使用生信人工具盒进行生存分析
https://blog.csdn.net/tuanzide5233/article/details/83685403
富集分析与蛋白质互作用网络(PPI)的可视化 Cystocape入门指南
https://blog.csdn.net/tuanzide5233/article/details/88048439
进阶版Venn plot:Upset plot入门实战代码详解——UpSetR包介绍
https://blog.csdn.net/tuanzide5233/article/details/83109527
使用R语言ggplot2包绘制pathway富集分析气泡图(Bubble图):数据结构及代码
https://blog.csdn.net/tuanzide5233/article/details/82141817
前段时间发现发现本文被 程序员大本营 剽窃 大概是这样的↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓