【机器学习 - 2】:数据集的处理
文章目录
- 训练集和数据集分离
- 获取最优模型
-
- 超参数
- 寻找最优模型
- 网格搜索的使用
训练集和数据集分离
训练集和数据集分离的原理:当我们获取一个数据集时,我们需要将其一小部分拿出来作为测试集,剩余的作为训练集。例如对于一个训练集,将其20%作为测试集,80%作为训练集,这20%的测试集是已经有目标值了的,将训练集进行拟合,获得模型,再通过测试集进行测试,获得最终结果,将最终结果和已知的目标值进行比对,可预测其训练模型的精确度。

以下使用sklearn中的knn算法进行预测,以识别鸢尾花为例。
- 先获取数据集,观察下图中y的值,可将0,1,2分别看做鸢尾花的不同种类。
from sklearn.datasets import load_iris
# 获取数据集
iris = load_iris()
X = iris.data
y = iris.target
- 由上图可看出数据集的目标值是有一定顺序的,我们需要将其打乱后再分出训练集和测试集,打乱用到的函数为np.random.permutation(),下图中shuffle_indexs里是0-150的随机索引
import numpy as np
shuffle_indexs = np.random.permutation(len(X))
- 打乱数据后开始取训练集和测试集,训练集取80%,测试集取20%
test_ratio = 0.2 # 取20%做测试集
test_size = int(len(X) * test_ratio)
test_indexs = shuffle_indexs[:test_size] # 测试集索引
train_indexs = shuffle_indexs[test_size:] # 训练集索引
# 获得训练集
X_train = X[train_indexs]
y_train = y[train_indexs]
# 获得测试集
X_test = X[test_indexs]
y_test = y[test_indexs]
- 调用sklearn中的knn算法,将训练集进行拟合,获得模型,测试集通过训练的模型,获得最终的预测结果,观察下图可看到y_predict和y_test(标准答案),大部分是相同的。
from sklearn.neighbors import KNeighborsClassifier
knn_clf = KNeighborsClassifier()
knn_clf.fit(X_train, y_train) # 拟合,获得模型
y_predict = knn_clf.predict(X_test) # 获取测试集的最终结果
- 根据y_predict和y_test,计算出模型的准确度,每次运行获得的准确度都不一样,但是准确率都在90%以上,说明模型的准确度较高。
np.sum(np.array(y_predict == y_test, dtype='int'))/len(y_test)
获取最优模型
参数的不同,会导致模型的不同,从而我们需要找到最合适的参数,从而训练出最优的模型。
我们可以自定义一个train_test_split函数,获取到训练集和测试集数据。根据以上的代码,编写的函数如下:
import numpy as np
def train_test_split(X, y, test_ratio=0.2, random_state=None):
if random_state:
np.random.seed(random_state) # 设置随机种子
shuffle_indexs = np.random.permutation(len(X))
test_ration = test_ration
test_size = int(len(X) * test_ratio)
test_indexs = shffle_indexs[:test_size]
train_indexs = shuffle_indexs[test_size:]
# 训练集
X_train = X[train_indexs]
y_train = y[train_indexs]
# 测试集
X_test = X[test_indexs]
y_test = y[test_indexs]
return X_train, X_test, y_train, y_test
我们也可以使用sklearn中封装好的train_test_split
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_iris
iris = load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=666)
超参数
超参数:在执行程序之前需要确定的参数。
举个例子:在knn分类器中,即KNeighborsClassifier(n_neighbors=3),n_neighbors值的不同,会导致模型的准确率不同,我们需要不断调整参数,找到某个数更加拟合我们的数据,这就是超参数。
权重问题:在【机器学习 - 1】:knn算法这一篇文章里,我们举了一个使用knn算法判断肿瘤为恶性肿瘤或良性肿瘤的例子,这个例子中我们主要以离待预测点周围最近的3个点进行判断。
而在如下图的情况中,待预测点(绿点)离红点(良性肿瘤)比较近,则它更可能为良性肿瘤,若以上篇文章中的思路来判断,因为它周围有2两个恶性肿瘤(蓝点),所以它很可能为恶性肿瘤。根据以上两种判断情况,我们需要把距离和个数这两种判断特征都考虑进来。
即绿色的点离红色的点最近,我们可以给这些距离加一个权重,这样及时周围有两个蓝点,但红点最近的距离权重大于这两个蓝点的距离权重,绿色的点可能就为良性肿瘤

在KNeighborsClassifier()中可设置权重参数:weights
当weights=uniform时,不考虑距离带来的权重问题
当weights=distance时,距离作为计算的权重
我们先看一下KNeighborsClassifier()的源码(如下图2),weights默认为uniform,p=2这个p是距离方法(如下图1),当=1时为曼哈顿距离,p=2时为欧拉距离,p增大,计算距离的方法不同。在KNeighborsClassifier()中默认为欧拉距离
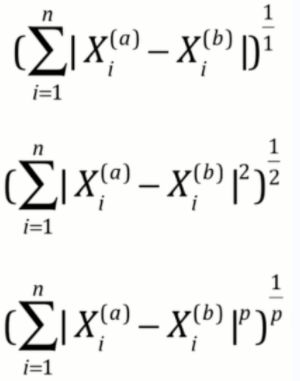

寻找最优模型
from sklearn.neighbors import KNeighborsClassifier
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
iris = load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X, y)
%%time
best_k = 0
best_score = 0.0
best_clf = None
best_method = None
best_p = 0
for p in range(1, 6):
for weight in ['uniform', 'distance']:
for k in range(1, 21):
knn_clf = KNeighborsClassifier(n_neighbors=k, weights=weight, p=p)
knn_clf.fit(X_train, y_train)
score = knn_clf.score(X_test, y_test)
if score>best_score:
best_score = score
best_k = k
best_clf = knn_clf
best_method = weight
best_p = p
print(best_k)
print(best_score)
print(best_clf)
print(best_method)
print(best_p)
网格搜索的使用
本次网格搜索的数据集以手写识别数据集为例。
- 获取数据,可以打印描述信息进行查看。
from sklearn.datasets import load_digits # 导入手写识别数据集
import numpy as np
from matplotlib import pyplot as plt
digits = load_digits()
X = digits.data
y = digits.target
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y)
- 绘制出手写数字
x = X_train[1000].reshape(8, -1)
plt.imshow(x, cmap=plt.cm.binary)
plt.show()
- 使用sklearn中的grid search
# 创建网格参数,每一组参数放在一个字典中
param_grid = [
{'weights':['uniform'],
'n_neighbors':[i for i in range(1,21)]
},
{
'weights':['distance'],
'n_neighbors': [i for i in range(1,21)],
'p':[i for i in range(1,6)]
}
]
from sklearn.model_selection import GridSearchCV
from sklearn.neighbors import KNeighborsClassifier
knn_clf = KNeighborsClassifier()
%%time
# 尝试寻找最佳参数
grid_search = GridSearchCV(knn_clf, param_grid, verbose=2, n_jobs=-1) # verbose越大越详细,n_jobs调用几个cpu进行计算,当n_jobs=-1时表示调用所有cpu进行计算
grid_search.fit(X_train, y_train)
grid_search.best_estimator_
grid_search.best_score_
grid_search.best_params_








