IF:6+ 综合分析揭示了一种炎症性癌症相关的成纤维细胞亚型在预测膀胱癌患者的预后和免疫治疗反应方面具有重要意义...
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.这期分享一篇2022年12月份发表在International Journal of Molecular Sciences (IF=6.2),作者综合分析揭示了一种炎症性癌症相关的成纤维细胞亚型在预测膀胱癌患者的预后和免疫治疗反应方面具有重要意义。
该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
单细胞测序数据分析教程整理如下:
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【5】单细胞转录组数据可视化分析 (scater)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【14】单细胞调节网络推理和聚类 (SCENIC)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
桓峰基因公众号推出转录组分析教程整理如下:
RNA 1. 基因表达那些事--基于 GEO
RNA 2. SCI文章中基于GEO的差异表达基因之 limma
RNA 3. SCI 文章中基于T CGA 差异表达基因之 DESeq2
RNA 4. SCI 文章中基于TCGA 差异表达之 edgeR
RNA 5. SCI 文章中差异基因表达之 MA 图
RNA 6. 差异基因表达之-- 火山图 (volcano)
RNA 7. SCI 文章中的基因表达——主成分分析 (PCA)
RNA 8. SCI文章中差异基因表达--热图 (heatmap)
RNA 9. SCI 文章中基因表达之 GO 注释
RNA 10. SCI 文章中基因表达富集之--KEGG
RNA 11. SCI 文章中基因表达富集之 GSEA
RNA 12. SCI 文章中肿瘤免疫浸润计算方法之 CIBERSORT
RNA 13. SCI 文章中差异表达基因之 WGCNA
RNA 14. SCI 文章中差异表达基因之 蛋白互作网络 (PPI)
RNA 15. SCI 文章中的融合基因之 FusionGDB2
RNA 16. SCI 文章中的融合基因之可视化
RNA 17. SCI 文章中的筛选 Hub 基因 (Hub genes)
RNA 18. SCI 文章中基因集变异分析 GSVA
RNA 19. SCI 文章中无监督聚类法 (ConsensusClusterPlus)
RNA 20. SCI 文章中单样本免疫浸润分析 (ssGSEA)
RNA 21. SCI 文章中单基因富集分析
RNA 22. SCI 文章中基于表达估计恶性肿瘤组织的基质细胞和免疫细胞(ESTIMATE)
RNA 23. SCI文章中表达基因模型的风险因子关联图(ggrisk)
RNA 24. SCI文章中基于TCGA的免疫浸润细胞分析 (TIMER)
RNA 25. SCI文章中估计组织浸润免疫细胞和基质细胞群的群体丰度(MCP-counter)
RNA 26. SCI文章中基于转录组数据的基因调控网络推断 (GENIE3)
RNA 27 SCI文章中转录因子结合motif富集到调控网络 (RcisTarget)
桓峰基因公众号推出临床预测模型教程,整理如下:
Topic 1. 临床标志物生信分析常规思路
Topic 2. 生存分析之 Kaplan-Meier
Topic 3. SCI文章第一张表格--基线表格
Topic 4. 临床预测模型构建 Logistic 回归
Topic 5. 样本量确定及分割
Topic 6 计数变量泊松回归
Topic 7. 临床预测模型--Cox回归
Topic 8. 临床预测模型-Lasso回归
Topic 9. SCI 文章第二张表—单因素回归分析表
Topic 10. 单因素 Logistic 回归分析—单因素分析表格
Topic 11. SCI中多元变量筛选—单/多因素表
Topic 12 临床预测模型—列线表 (Nomogram)
Topic 13. 临床预测模型—一致性指数 (C-index)
Topic 14. 临床预测模型之校准曲线 (Calibration curve)
Topic 15. 临床预测模型之决策曲线 (DCA)
Topic 16. 临床预测模型之接收者操作特征曲线 (ROC)
Topic 17. 临床预测模型之缺失值识别及可视化
Topic 18. 临床预测模型之缺失值插补方法
Topic 19. 临床预测模型之输出每个患者列线图得分 (nomogramFormula)
Topic 20. 临床预测模型之竞争风险模型及计算生存概率
基于机器学习构建临床预测模型整理如下:
MachineLearning 1. 主成分分析(PCA)
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
摘 要
炎性癌相关成纤维细胞(iCAFs)与膀胱癌(BCa)的进展、抗癌治疗耐药性和不良预后密切相关。然而,iCAFs在BCa中的功能作用研究很少。在我们的研究中,通过Seurat管道获得并集成了两个BCa scRNA-seq数据集(GSE130001和GSE146137)。基于已报道的标记(COL1A1和PDGFRA),鉴定了iCAFs,并开发了278个相关标记。在无监督共识聚类后,发现了两种TCGA-BLCA分子亚型,并以不同的癌症异常特征、免疫肿瘤微环境、预后、对化疗/免疫治疗的反应和干性为特征。随后,基于特征的聚类在预后和治疗反应预测方面的稳健性在GEO-meta队列中得到验证,该队列包含7个独立的GEO数据集(519例BCa患者)和3个免疫检查点抑制剂(ICI)治疗的队列。考虑到iCAFs的异质性,对iCAFs进行重新聚类,并鉴定出一个亚群体,命名为“LOXL2+ iCAFs”。LOXL2过表达/沉默CAFs与T24细胞共培养的CM显示,在CAFs中过表达LOXL2促进了T24细胞的增殖、迁移和侵袭,而沉默LOXL2通过IL32抑制了T24细胞。此外,LOXL2与M2巨噬细胞极化标志物CD206正相关。总的来说,整合的单细胞和体RNA测序分析揭示了icaf相关的特征,可以预测BCa的预后和免疫治疗反应。此外,Hug 基因LOXL2可能是BCa治疗的一个有前景的靶点。
生信分析流程
相关数据准备
数据集选择:
单细胞数据集:GSE130001, GSE146137
bulk表达数据集:TCGA-BLCA, GSE5287, GSE13507, GSE31684, GSE48075, GSE48277, GSE69795, and GSE70691
3个免疫检查点抑制剂(ICI)治疗的队列: IMvigor210 (BCa), GSE78220 (melanoma), and GSE135222 (lung cancer)
基因集选择:
从基于网络的工具StemChecker (http://stemchecker.sysbiolab)下载了26个与茎干相关的基因集。T细胞衰竭(TEX)相关签名和28个免疫相关签名分别来自Zhang et al.和Wang et al.的研究。
生信分析方法:
我们从文章的分析流程中提取所有的分析内容,整理出来就 11个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于生信分析+实验结合的文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
1. 根据 R 包 Seurat 逐步进行归一化、积分、降维和聚类;
2. 利用 FindMarkers 函数与 logFC 识别 iCAF 相关标记;
3. GSVA包的单样本基因集富集分析(ssGSEA);
4. 采用基因集富集分析(GSEA)评价分子亚型之间的生物学特性;
5. 使用R包clusterProfiler执行ORA和GSEA并可视化结果;
6. ConsensusClusterPlus 无监督聚类探索生物特性;
7. 通过t-SNE对聚类性能进行可视化;
8. 肿瘤免疫微环境(TIME)的组成:ESTIMATE, CIBERSORT, quanTIseq, TIMER,MCPcounter;
9. 采用BLCAsubtyping R包评估基于TCGA和UNC分类的共识分子亚型比例;
10. 使用predictic R包和geneppattern评估患者对化疗和免疫治疗的反应;
11. 基于 STRING database 利用 Cytoscape 分析识别 Hug 基因。
研究结果
1. scRNA-seq分析强调了iCAFs在BCa中的作用
(A) CAFs及其对应两个亚群(iCAFs和mCAFs)分别由先前报道的标记(COL1A1、PDGFRA和RGS5)识别。Vlinplots显示了每种基因的表达水平标记在十个簇中。t-SNE图显示聚类中各标记物的表达水平4个和7个分别被命名为iCAFs和mCAFs。
(B)热图说明了表达式iCAFs和mCAFs前10个细胞类型特异性标记的差异。
(C)GO(柱状图,左)和KEGG(点状图,左)通过iCAFs(上)和mCAFs(下)的标记,ORA展示了丰富的术语。
(D)GSEA分别在iCAFs(上)和mCAFs(下)中显示出上调的癌症特征。
2. iCAFs相关亚型具有不同的免疫基因组模式
(A)TCGA-BLCA分为两个分子亚型。
(B)通过t-SNE可见两个亚型之间的区别。
(C)簇2具有较高的iCAF特征评分。
(D) Heatmap显示了三种不同iCAF相关亚型的免疫亚型、ESTIMATE评分和iCAF特征评分。
(E)两种亚型间质、免疫、估计评分、肿瘤纯度和iCAF特征的分布。
(F)两种亚型之间22种免疫细胞的浸润丰度。
(G)TEX特征基因和M2巨噬细胞极化因子(H)在两亚型间的表达水平。
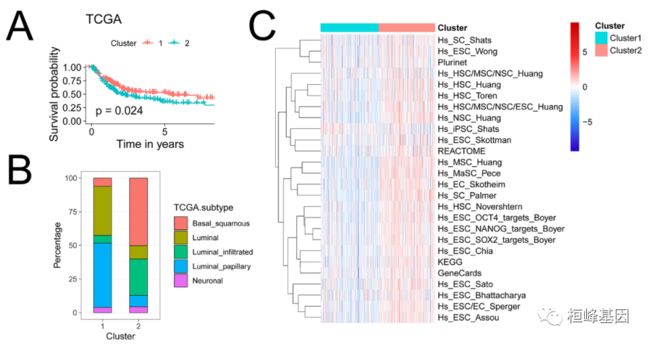
3. iCAFs相关亚型具有明显的通路失调
(A)聚类2与TCGA-BLCA预后较差有关。
(B)两个簇间TCGA分子亚型一致。
(C)两个簇间26个茎干相关特征的富集活性。
(D)簇1(左)和簇2(右)的癌症特征上调。
(E)簇2通路上调。
(F)免疫治疗反应预测。
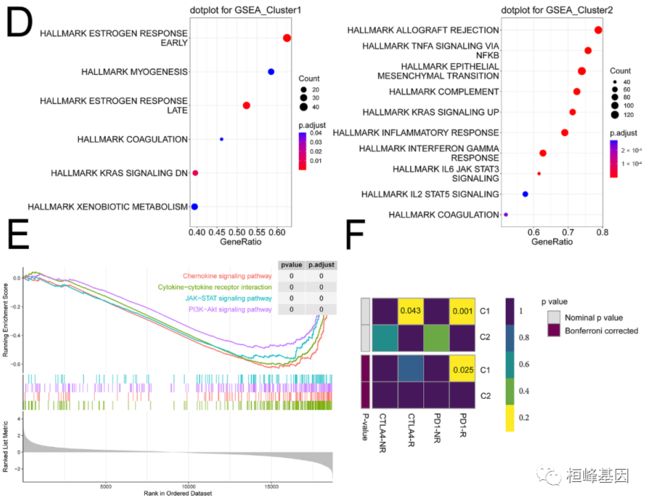
4. 在IMvigor210中, iCAFs相关亚型预测预后和免疫治疗反应
(A)通过基于icaf相关特征的共识聚类确定了两个聚类。与聚类2的患者相比,聚类1的患者预后较差。
(B)堆叠条形图显示集群1与集群2相比有更高比例的应答者。p值由卡方检验确定。R:应答器,NR:非应答器。
(C)聚类1的icaf相关评分显著高于聚类2。每个IMvigor210样本的icaf相关评分由ssGSEA确定。
(D)簇1与高TMB有关
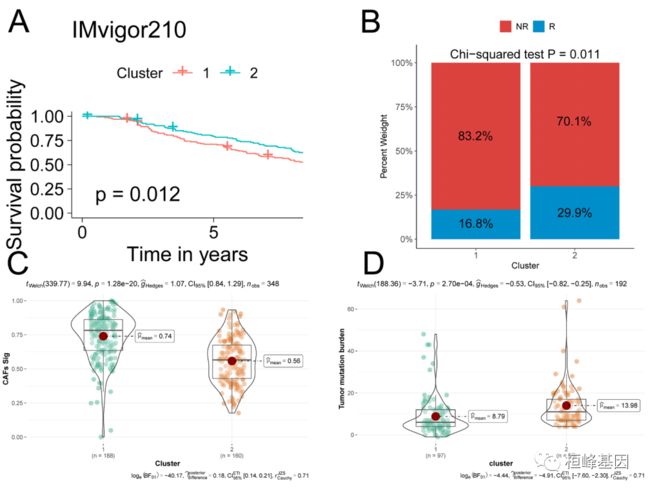
5. LOXL2+ iCAFs预测预后不良
(A)通过分析PPI网络鉴定出LOXL2 Hub基因。
(B) LOXL2高表达与不良预后相关。
(C)过表达LOXL2组M2巨噬细胞浸润水平明显升高。
(D)高表达的LOXL2与几种下调的免疫治疗预测通路有关。
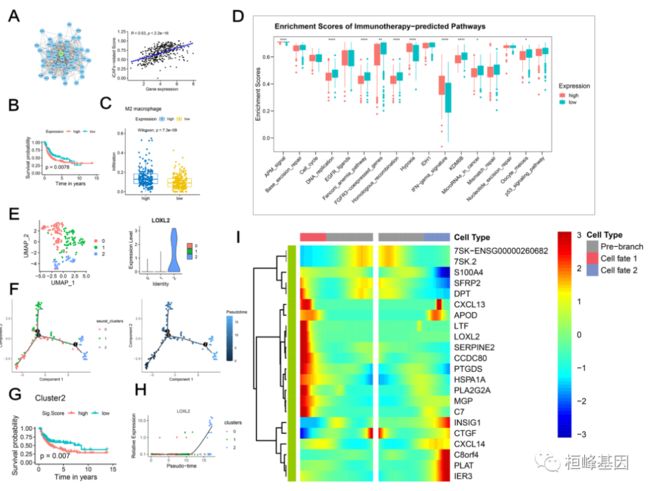
(E) iCAFs的重新聚类鉴定出三个亚群,LOXL2在亚簇2中高度表达。
(F)伪时间分析表明,子簇0投影到根上,子簇1和子簇2分别投影到两个分支上。
(G)亚簇2与预后较差有关。
(H)LOXL2表达水平随轨迹树变化显著。
(I)Heatmap显示分支依赖基因。
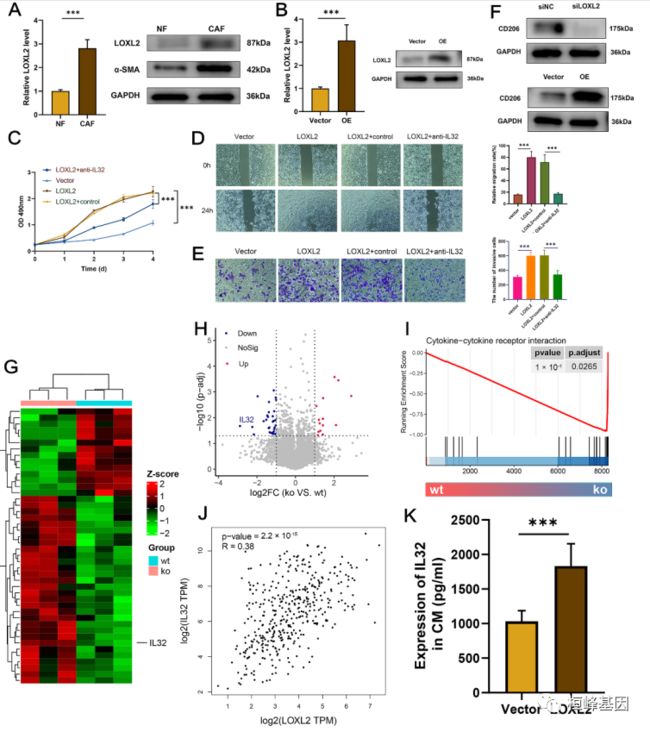
6. CAFs中LOXL2的上调促进了BCa细胞的增殖、迁移和转移
(A) LOXL2和α-SMA在CAFs中mRNA和蛋白水平上调。
(B) qRT-PCR和WB验证过表达的疗效。
(C) CCK-8实验证明LOXL2在CAFs中与抗il32一起或不一起促进增殖。
(D,E)对T24细胞进行与(C)相同的处理,并进行伤口愈合试验(D)和Transwell试验(E),以评估肿瘤细胞的迁移和侵袭潜力。
(F)条件LOXL2影响CD206蛋白水平。
(G,H) IL32在siLOXL2组下调。
(I)siLOXL2富集。
(J) LOXL2和IL32之间的相关性。
(K) ELISA检测经LOXL2处理的CAFs培养上清中IL32蛋白的表达。
这篇文章利用桓峰基因公众号上的教程完全可以实现这些图表的绘制,过程需要自己细致的探究,毕竟不是很简单的分析,能发到6+也需要有一定的生信分析和结果解读的能力,有需要此类分析的老师可以联系桓峰基因!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!
References:
Chen H, Yang W, Xue X, Li Y, Jin Z, Ji Z. Integrated Analysis Revealed an Inflammatory Cancer-Associated Fibroblast-Based Subtypes with Promising Implications in Predicting the Prognosis and Immunotherapeutic Response of Bladder Cancer Patients. Int J Mol Sci. 2022;23(24):15970. Published 2022 Dec 15. doi:10.3390/ijms232415970
Zhang, Z.; Chen, L.; Chen, H.; Zhao, J.; Li, K.; Sun, J.; Zhou, M. Pan-cancer landscape of T-cell exhaustion heterogeneity within the tumor microenvironment revealed a progressive roadmap of hierarchical dysfunction associated with prognosis and therapeutic efficacy. eBioMedicine 2022, 83, 104207.
Wang, Z.; Wang, Y.; Yang, T.; Xing, H.; Wang, Y.; Gao, L.; Guo, X.; Xing, B.; Wang, Y.; Ma, W. Machine learning revealed stemness features and a novel stemness-based classification with appealing implications in discriminating the prognosis, immunotherapy and temozolomide responses of 906 glioblastoma patients. Brief. Bioinform. 2021, 22, bbab032.