mindspore-HRNet的新冠肺炎CT分割
mindspore-HRNet的新冠肺炎CT分割
- 环境
- 一、数据集准备
-
- 1.切片
- 2.生成mask
- 二、训练,测试
-
- 分割可视化效果
- 总结
环境
mindspore-gpu==1.7.0
一、数据集准备
此次实验的数据集为COVID-19 CT scans,链接:https://www.kaggle.com/datasets/andrewmvd/covid19-ct-scans
该数据集包含20例诊断为COVID-19的患者的CT扫描以及专家对肺部和感染的分割
主要由四个部分组成:新冠肺炎CT,只标记了感染区域,标记了肺部区域,标记了感染区域和肺部

通过Ascend model zoom下载HRNet源码
1.切片
下载后的数据集为三维CT数据,nii格式,通过代码可视化看一下:
import matplotlib
from matplotlib import pylab as plt
import nibabel as nib
from nibabel import nifti1
from nibabel.viewers import OrthoSlicer3D
example_filename = 'coronacases_org_001.nii'
img = nib.load(example_filename)
print(img)
print(img.header['db_name']) # 输出头信息
#显示图像
width, height, queue = img.dataobj.shape
OrthoSlicer3D(img.dataobj).show()

由于缺乏CT数据的训练经验,以及暂时不太会实现mindspore读取.nii数据,于是准备对CT数据进行轴向切片,然后转换为jpg格式的原始数据及png格式的mask图像
但其实这样做存在数据信息损耗的问题,可能对分割精度有影响
主要是因为Ct数据和二维图像的灰度值范围及位宽不一样
切片代码如下:
import numpy as np
import os # 遍历文件夹
import nibabel as nib # nii格式一般都会用到这个包
import imageio # 转换成图像
def nii_to_image(niifile):
filenames = os.listdir(filepath) # 读取nii文件夹
print(filenames)
slice_trans = []
for f in filenames[2:6]:
# 开始读取nii文件
img_path = os.path.join(filepath, f)
img = nib.load(img_path) # 读取nii
img_fdata = img.get_fdata()
fname = f.replace('.nii', '') # 去掉nii的后缀名
img_f_path = os.path.join(imgfile, fname)
# 创建nii对应的图像的文件夹
if not os.path.exists(img_f_path):
os.mkdir(img_f_path) # 新建文件夹
# 开始转换为图像
(x, y, z) = img.shape
for i in range(z): # z是图像的序列
silce = img_fdata[:, i, :] # 选择哪个方向的切片都可以
imageio.imwrite(os.path.join(img_f_path, '{}.png'.format(i)), silce)
# 保存图像
if __name__ == '__main__':
filepath = 'infection_mask/'
# filenames2 = os.listdir(filepath1)
# print(filenames2)
# for i in filenames2:
# filepath= os.path.join('ct_scans/', i)
imgfile = "images/"
nii_to_image(filepath)
以同样的方式,对标记CT数据进行切片
让我们看下结果:
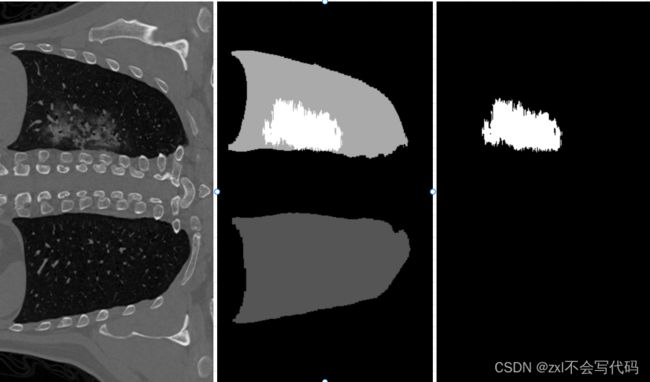
其中,白色的部分为病变区域
此次实验只使用标记了病变区域的标签,也就是第三幅图这种
2.生成mask
标记了感染区域的png图像虽然已经是8位图,但不能直接拿来训练,需要将正常区域像素值改为0,病变区域改为1
自己写了个脚本转换下:
import PIL.Image as Image
import os
filePath = 'train_segmentations/'
f = os.listdir(filePath)
for image in f:
print(image)
img = Image.open('train_segmentations/'+image)
img_array = img.load()
# 遍历每一个像素块,并处理颜色
width, height = img.size # 获取宽度和高度
for x in range(0, width):
for y in range(0, height):
r = img_array[x, y] # 获取一个像素块的rgb
if r==255: # 判断规则
img_array[x, y] = 1
else:
img_array[x, y] = 0
img.save("label/"+image)
转换结果为全黑的图,因为像素值都为0,1,太低了,所以看上去是全黑的

最后,按照官方的脚本制作索引文件train.lst, val.lst
最终的数据集结构如下:

二、训练,测试
执行train.py进行训练:
对验证集数据进行验证,计算mIoU:
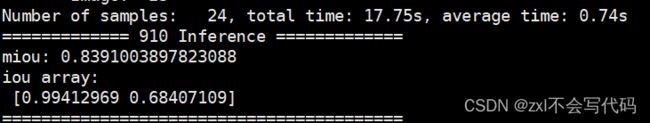
Miou很高,主要是我训练集和测试集很多数据重复,但能看出来分割效果确实还可以
分割可视化效果
总结
基本实现了基于昇思mindspore的新冠肺炎CT数据病灶分割
存在以下两点问题:
1.数据集转为2维图像的信息损耗
2.深度学习依旧是数据驱动的,而病灶图像难以获取,需要进行数据赠广或弱监督学习
