Nat. Biotechnol. | 利用深度学习从基因转录数据中预测药物疗效
作者 | 蒋长志
审稿 | 蒋立坤
今天给大家介绍来自北京大学医学部的谢正伟团队发表在Nature Biotechnology上的文章,文章提出了一种基于深度学习的药效预测系统(DLEPS),该系统将药物SMILES分子输入神经网络来拟合药物作用下基因表达谱变化信息,从而预测疾病药物分子,该模型在测试集上的预测效果(Pearson相关系数)达到0.74;并进行了案例分析,即分别预测肥胖、高尿酸血症和非酒精性脂肪性肝炎三种疾病药物分子,随后通过构建小鼠模型验证了系统预测的候选药物的有效性,表明该系统具有通用性,可以预测多种疾病的药物分子。
1
研究背景
在过去的几十年里,基于靶蛋白的药物研发已经是一种成功的方法,但是这些方法不能处理缺乏明确定义靶蛋白的疾病,那么针对这种疾病的策略是设计一个能够独立于特定靶标的药效预测模型;比如Stokes, J. M等人于2020年发表在Cell期刊上的研究提出使用定制的深度学习模型来发现治疗大肠杆菌感染的新候选抗生素,但是该模型依赖于特定单一疾病状态的表型数据,缺乏推广到其他疾病的能力。因此,设计出一种能够预测多种疾病药物分子的通用系统是有重要意义的。
2
主要贡献
(1)本文提出了一个基于深度学习的疾病药物分子预测模型,该模型是利用药物分子和药物作用下的基因表达谱信息进行训练,不需要知道靶蛋白信息,这给缺乏明确定义靶蛋白的疾病药物分子预测带来新的思路;
(2)本文做了生物实验验证了系统的有效性,分别是使用褐变基因特征寻找抗肥胖分子、使用多个基因特征识别抗高尿酸血症分子以及鉴定抗非酒精性脂肪性肝炎的药物分子。
3
模型
3.1数据预处理
本文使用的数据集是L1000,该数据来源于一个项目库(Integrated Network-Based Cellular Signatures),对其进行清洗处理,按照以下规则进行清洗:
分子必须有超过5个备份;
分子的SMILES要能使用RDKIT(version 2017.9.1)工具解析。
随后,通过处理过程获得了与疾病的标志性基因有关的17051个已经验证的分子,其中随机选取14051个分子作为训练集,1500个分子作为验证集以及1500分子作为测试集。
3.2 方法
模型输入与疾病标志性基因表达谱变化有关的药物分子数据,输出药物作用下的基因表达谱信息,从而预测疾病药物分子。首先,使用GVAE(语法树模型)对药物分子的SMILES进行编码,即将SMILES解析成一个语法树(有76个节点类型),并使用one-hot编码成一个向量;然后将SMILES的向量表示输入到包含三个一维卷积层的神经网络中获得新的特征表示,随后将获得的新特征表示拼接成一维的向量输入到全连接层中输出两个新的特征表示,分别是中心向量和半径向量,对中心向量和半径向量进行采样获得新的向量表示,随后作为隐向量输入到一个含有5层全连接层的神经网络(其中每层都施加Dropout机制,前3层的激活函数采用Relu,第4层的激活函数采用Tanh,最后一层不使用激活函数)来预测基因表达谱的变化,这里使用均方误差作为损失函数,并采用Adam作为优化算法。模型框架如下图所示:
DELEPS模型框架图
4
实验
4.1 预测结果
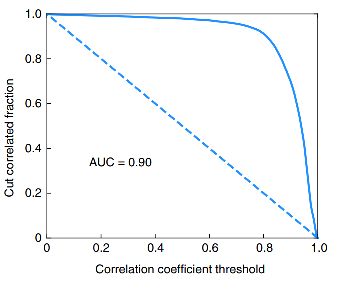
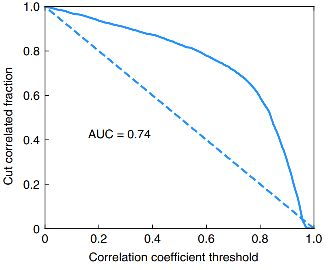
本文采用Pearson相关系数作为评价指标来评估模型的性能,并通过设置不同的Pearson相关系数阈值来计算平均Pearson相关系数作为模型性能指标;模型的训练集和测试集的不同Pearson相关系数阈值绘制的类似于ROC曲线如下图(a)和(b)所示:
4.2 案例分析
本文为了验证DLEPS系统的有效性,进行寻找肥胖、高尿酸血症以及非酒精性脂肪肝炎三种疾病的潜在治疗药物实验,并构建小鼠模型来验证系统的预测结果,结果表明系统具有通用性,可以预测多种疾病药物分子。
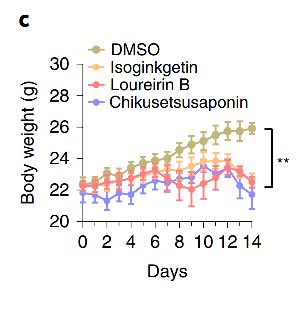
1)对于肥胖疾病,本文将DLEPS预测出的前几种候选药物分子喂入小鼠体内,通过观察小鼠的肥胖状况来筛选出治疗肥胖疾病的潜在药物分子。
注入潜在的药物分子后小鼠体重变化图
2)对于高尿酸血症(hyperuricemia), 本文将DLEPS预测的几种候选药物分子在小鼠体内进行生物实验,从而筛选出潜在的治疗高尿酸血症药物分子。
使用候选药物治疗高尿酸血症小鼠体内的血尿酸水平情况
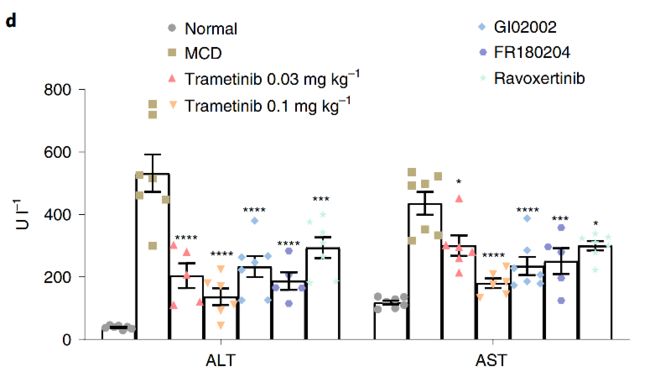
3)对于非酒精性脂肪性肝炎,本文将DLEPS预测的几种候选药物分子在小鼠体内进行生物实验,从而筛选出潜在的抗非酒精性脂肪性肝炎药物分子。
注射不同药物分子14天之后小鼠体内血清中ALT(丙氨酸氨基转移酶)和AST(天冬氨酸氨基转移酶)水平
5
总结
基于靶标-蛋白的药物发现方法面临着许多疾病的通路机制不清楚的困难,为了克服这一困难,本文提出了一个基于深度学习的药效预测系统(DLEPS),该系统利用复杂疾病的基因指纹信息来预测潜在疾病药物分子,使用超过百万个基因指纹和与这些基因指纹信息有关联的17051个结构多样的小分子数据进行训练和测试,其中测试集的Pearson相关系数达到0.74,并对三种疾病进行了案例分析,通过构建小鼠模型来验证DLEPS系统预测的这三种疾病的药物分子,结果表明该系统具有通用性,可以预测多种疾病药物分子,也给理解疾病的通路机制带来新视角。
参考资料
Zhu, J., Wang, J., Wang, X. et al. Prediction of drug efficacy from transcriptional profiles with deep learning. Nat Biotechnol (2021).
https://doi.org/10.1038/s41587-021-00946-z
代码
https://github.com/kekegg/DLEPS
往期精品(点击图片直达文字对应教程)
机器学习
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集