论文解读:预测lncRNA的相互作用生物分子类型:一种集成深度学习方法
Predicting the interaction biomolecule types forlncRNA: an ensemble deep learning approach
目录
1.摘要
2.引言
3.材料和方法
1.数据描述
2.特征提取
3.模型构建
4.评估标准
4.结果
1.LncRNA-生物分子相互作用的统计与分析
2.lncIBTP的初步培训以及评价
3.与特定lncRNA分子预测方法的比较
4.个案研究和跨物种验证
5.讨论
1.摘要
长非编码核糖核酸通过与脱氧核糖核酸、核糖核酸和蛋白质等生物分子的相互作用,在各种生理和病理过程中发挥着重要作用。现有的用于预测lncRNA功能的计算机方法主要依赖于计算lncRNA的相似性或研究lncRNA是否能与特定的生物分子或疾病相互作用。在这项工作中,我们从不同的角度探索了lncRNA的功能:我们提出了一个工具来预测给定lncRNA的相互作用生物分子类型。为了这个目的,我们首先研究了lncRNA-RNA、lncRNA-蛋白质和lncRNA-DNA相互作用的主要分子机制。然后,我们开发了一个集成深度学习模型:lncIBTP (lncRNA相互作用生物分子T型预测)。该模型预测了lncRNA与不同类型生物分子之间的相互作用。在5倍交叉验证中,lncIBTP在接收机工作特性曲线和精度-召回曲线下的精度平均值分别为0.7042、0.7903和0.6421,说明了模型的有效性。此外,基于对收集的已发表数据和预测结果的分析,我们假设与脱氧核糖核酸相互作用的lncRNAs的特征可能不同于仅与核糖核酸相互作用的lnc rnas的特征。
2.引言
长非编码核糖核酸是长度超过200个核苷酸的转录物,不编码蛋白质。近年来,lncRNAs因其在复杂的生理和病理过程中发挥重要作用而引起了研究者的广泛关注[1]。尽管大量的lncRNAs已经被注释,但是只有少数被很好地功能化。以往研究报道lncRNAs参与多种调控功能,如表观遗传修饰、染色质重塑、基因转录、翻译、蛋白质功能等。它们通过与其他生物分子如核糖核酸、蛋白质和脱氧核糖核酸相互作用来执行这些功能。
LncRNA与编码RNA(mRNA)和非编码RNA(ncRNA)相互作用。在lncRNA-mRNA相互作用的情况下,lnc RNA直接或间接调节mRNA的表达。一方面,lncRNA直接靶向mRNA,调控其剪接、编辑、亚细胞分布和稳定性。另一方面,lncRNA和mRNA可以通过形成竞争的内源性RNA (ceRNA)网络,通过共享的miRNA响应元件发生串扰。
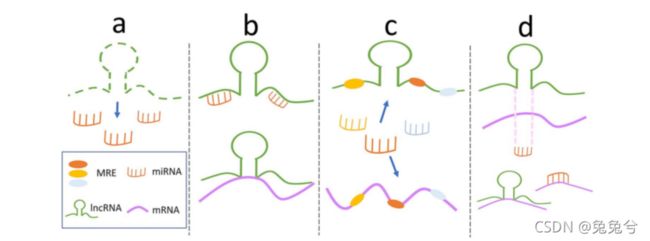
在lncRNA-ncRNA相互作用的情况下,lncRNA充当小ncRNA的前体,如miRNA ,snoRNA 和 piRNA。此外,在lncRNA和miRNA的关联中,miRNA调节lncRNA的表达和稳定性。一些内含子的末端与内含子的位置相对应;这类基因被称为sno-lncRNA 。先前的研究报道,sno-lncRNAs与Fox家族剪接调节因子密切相关,它们可以改变细胞中的剪接模式。此外,lncRNA与其他lncRNA相互作用,在表观遗传调控中发挥关键作用,包括等位基因和基因座特异性控制。研究表明,lncRNA Jpx可能激活lncRNA Xist,并能在激活剂和阻遏剂之间建立动态平衡,从而确保X染色体失活。图1显示了lncRNA和RNA之间常见的相互作用类型
图1.lncRNA–RNA相互作用类型(A) lncRNA是小ncRNA的宿主(B) lncRNA直接与RNA相互作用(C) lncRNA通过ceRNA网络中的miRNA调节mRNA(D) lncRNA通过与miRNA在结合位点上竞争来调节mRNA。
LncRNA还通过与蛋白质相互作用实现多种功能。一般来说,lncRNA在与蛋白质的关系中充当信号、向导、支架和诱饵。首先,lncRNA对刺激作出反应,并充当划分空间、时间和基因表达的信号[21]。其次,lncRNA通过引导蛋白质进入染色质位点来调节基因的表达。第三,lncRNA作为参与结构作用的蛋白质支架。最后,lncRNA充当远离目标位点的蛋白质的诱饵。
还发现了lncRNA与蛋白质相互作用的一些新关系。LncRNA-蛋白质相互作用可以调节LncRNA的稳定性,同时LncRNA可以作为蛋白质的ceRNA(内源性RNA),限制蛋白质与其靶mRNA之间的结合。此外,lncRNA直接或间接调节蛋白质的表达。
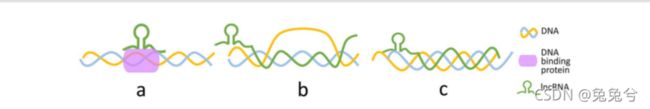
在lncRNA与DNA的相互作用中,lncRNA主要通过三种途径与DNA相互作用:(i)lncRNA与DNA结合蛋白结合(ii)lncRNA与DNA形成R环,(iii)lncRNA与DNA形成三联体。首先,在lncRNA介导的招募过程中,lncRNA可以通过靶向DNA位点来抑制或激活其与DNA结合蛋白的附着[4,29]。其次,R环的形成可能有助于基因表达。研究还表明,染色质压缩和功能中R-环的奇异性部分与lncRNAs在基因组组织和基因转录调控中的作用有关[31]。第三,lncRNA通过三联体结构调节基因的表达。图2简要说明了三种lncRNA–DNA相互作用的机制。
图2.lncRNA–DNA相互作用类型(A) lncRNA通过DNA结合蛋白与DNA相互作用(B) R环(C) DNA:RNA三联体。
基于lncRNA与其他生物分子之间的相互作用,人们开发了各种生物信息学工具来揭示lncRNA的功能。这些方法主要预测给定的lncRNA是否与一个或多个不同类型相互作用的特定生物分子相关。提出了一种深度学习方法来预测lncRNA–DNA结合,等等。因为一些lncRNA–生物分子相互作用与疾病有关,预测这种相互作用也有助于理解疾病和lncRNA–疾病预测。然而,对于新定义的lncRNAs,要完全预测它们的功能,像我们上面提到的那样运行所有类型的lncRNAs相互作用预测是相当不切实际的,如果我们能够通过指示它们相互作用的生物分子类型来缩小预测范围,预测过程可能会更加有效。
在这项工作中,我们的目标是预测一个lncRNA可能与之相互作用的生物分子类型。为此,我们研究了lncRNA与三种主要生物分子,即DNA、RNA和蛋白质之间的相互作用。首先,我们简要分析了我们在不同交互模式下收集的数据,然后我们开发了一种基于序列数据的集成深度学习方法lncIBTP(lncRNA交互生物分子类型预测)。根据lncRNA与不同类型生物分子的相互作用,我们将lncRNA分为四种类型,并从它们的数据中进一步确定。因此,lncRNA有四种类型:仅与RNA相互作用的lncRNA、仅与蛋白质相互作用的lncRNA、与RNA&蛋白相互作用的lncRNA和与DNA相关的lncRNA。与DNA相关的lncRNA类型是能够与DNA相互作用的lncRNA;它还包括四种类型的相互作用:仅与DNA、DNA& RNA、DNA&蛋白质和DNA& RNA&蛋白质相互作用的lncRNA。在5倍交叉验证测试中,删除冗余数据集的准确度(Acc)、宏观平均准确度、宏观平均召回率和宏观平均F分数的平均值为0.704,0.565、0.719和0.610。此外,对小鼠数据进行跨物种验证的Acc为0.517,这有点低。一般来说,考虑到四标签分类的随机猜测Acc为0.25,lncIBTP的表现令人满意。它通过指示可能与lncRNA相互作用的生物分子类型,帮助我们破译lncRNA功能代码,这缩小了lncRNA和特定分子之间进行预测的范围。此外,预测结果和我们收集的数据帮助我们得出以下假设:与DNA相互作用的lncRNAs的特征可能不同于仅与RNA相互作用的lncRNAs。
3.材料和方法
1.数据描述
我们从NPInter v4.0获得人类lncRNA数据,lncRInter数据库。这两个数据库提供了lncRNA和其他生物分子之间经实验验证的功能性相互作用。在每个数据库中,我们根据lncRNA与不同生物分子类型之间的相互作用将lncRNA分为七种类型。七种类型的lncRNA如下:lncRNA仅与RNA相互作用、lncRNA仅与蛋白质相互作用、lncRNA仅与DNA相互作用、lncRNA与蛋白质和RNA相互作用、lncRNA与蛋白质和DNA相互作用、lncRNA与RNA和DNA相互作用以及lncRNA与蛋白质、RNA和DNA相互作用。图3A说明了在两个数据库中被分为七种类型的独特lncRNAs的数量;然而,没有将lncRNA分为RNA型和DNA型。与其他类型相比,仅与DNA、蛋白质& DNA、RNA& DNA&蛋白质、RNA& DNA相关的lncRNA数量非常少。因此,我们将这四种类型归为一种单一类型,并将其注释为DNA相关类型。通过无重复地结合两个数据库的结果,并与GENCODE release 33 lncRNA注释进行对比,我们构建了数据集,用于预测lncRNA与以下类型生物分子的四种相互作用:lncRNA与RNA、蛋白质、RNA&蛋白质和DNA相关。最近,一项研究报告了lncRNA MIR100HG的DNA:RNA三联体的形成[32]。该lncRNA也被添加到我们的数据集中,并被归类为DNA相关类型。我们进一步应用了CD-HIT withr holdof0.8删除每个类的冗余。图3B显示了删除数据集中冗余前后每个类的lncRNA数量。
图3.数据描述(A) 两个数据库中的数据类和数据量,左:NPInter v4.0,右:lncRInter。图中的数字表示每种相互作用类型中lncRNA的数量,重叠表示lncRNA与两种或更多类型的分子相互作用。在NPV4.0数据库,共有五种类型的lncRNA:仅与RNA、仅与蛋白质、RNA&蛋白质、DNA&蛋白质和RNA& DNA&蛋白质相互作用的lncRNA;在lncRInter数据库中,它们是六种类型的lncRNA:仅与RNA、仅与蛋白质、仅与DNA、RNA&蛋白质、DNA&蛋白质和RNA& DNA&蛋白质相互作用的lncRNA.(B) 构造的数据集的数据类和数量。数据分为四类:仅与RNA相互作用的lncRNA、仅与蛋白质相互作用的lncRNA、与RNA&蛋白质相互作用的lncRNA和与DNA相关的lncRNA。该数字表示与GENCODE注释对比后从上述两个数据库收集的lncRNA数量/在四个类中进一步移除阈值为0.8的冗余(C) 样本种类的数据类别和数量。该数字表示与GENCODE注释对比后从NpInter 4.0收集的lncRNA数量。
2.特征提取
k-mer是一种流行的特征表示方法;它考虑了序列中“k”相邻核苷酸的频率。该方法已成功用于解决与RNA相关的分类问题。g-gap和g-bigap特征是k-mer方法的改进版本;它们是通过在2-聚体中的两个单核苷酸和4-聚体中的两个核苷酸对之间引入间隙而形成的。在本研究中,我们使用k-mer、g-gap和g-bigap从序列信息中提取特征。为了确定k和g的值,我们在数据集的每个类别中随机选择20%的数据作为验证数据。从剩下的80%的数据中,我们使用以下两种方法对每一类抽取500个样本:(i) 用bagging策略对弱类进行过采样,RNA和DNA的类别相关和,并且(ii)对主要类别随机取样不足,蛋白质类和RNA&蛋白质。因此,我们用不同的k、g及其组合来训练模型。该模型用一个卷积层和50个epochs进行训练。结果如补充图S1所示。最终的特征矩阵是通过将3-mer、2-bigap和3-bigap方法与总共576个(43 44个)特征相结合而得到的,在几种选择中获得了最佳的整体Acc
图S1。在k-mer、g-gap和g-bigap特征表示方法中选择不同的k和g时的验证结果。结果来自于用1层CNN和50个历元训练的模型。
3.模型构建
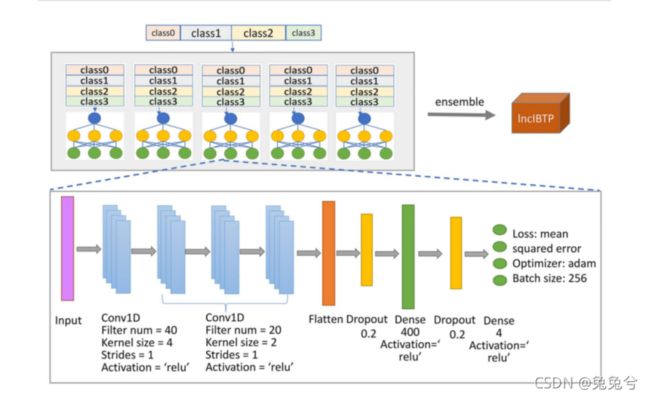
在我们的数据集中,数据极不平衡,仅与蛋白质和RNA&蛋白质相互作用的lncRNA样本数是仅与RNA和DNA相关蛋白相互作用的样本数的12倍左右。为了消除这种预测偏差,我们在训练期间对与套袋策略相关的RNA和DNA类进行过采样,并对蛋白质类和RNA&蛋白质类进行随机欠采样,从而对每个类抽取n个样本。n值取2000,即进一步发现,在多种选择中,可实现最佳的整体Acc(补充图S2A)。利用平衡训练数据,我们在验证数据集上进一步确定了单卷积神经网络(CNN)模型的其他参数,如卷积层数和核大小、激活函数、密集层大小,等。每次我们在保持其他参数不变的情况下更改一个参数的值,然后选择达到最高总体Acc的参数作为该参数的最终值。参数设置和相应性能如补充图S2B–H所示。此外,还采用了集成思想来提高预测性能;采用五个单CNN模型作为集成模型;更多详细信息请参见结果部分的“lncIBTP初始培训”。在上述策略的帮助下,我们最终构建了lncIBTP方法:一种用于预测lncRNA相互作用生物分子类型的集成深度学习方法。通过对原始训练数据集进行过采样或欠采样,重新选择每个模型的训练数据。lncIBTP的预测标签定义为最大概率得分从输入样本中脱落的标签。图4显示了lncIBTP的详细结构和参数。
图S2。网络参数的确定(a) 在每个类别中,通过不同的样本数选择实现的精度。这些模型用1个卷积层(滤波器数量=40,内核大小=2,步长=1)训练,50个历元,20%退出(b) 使用不同数量的卷积层实现的精度(c) 通过不同的辍学率选择获得的准确度(d) 通过选择不同的激活函数实现的精度(e) 在第一个卷积层中,通过选择不同的内核大小来实现精度(f) 通过不同的优化器选择实现的精度(g) 通过选择不同的致密层尺寸获得的精度(h) 通过不同的训练时间选择获得的总体精度。
图4.lncIBTP的体系结构。类别0-3代表分别与RNA、蛋白质、RNA&蛋白质和DNA相关的lncRNAs相互作用。之所以采用激活函数ReLU,是因为与其他函数(函数为f(x)=max)相比,其性能更好。采用D r o p o t l a y e r以防止过度装配。
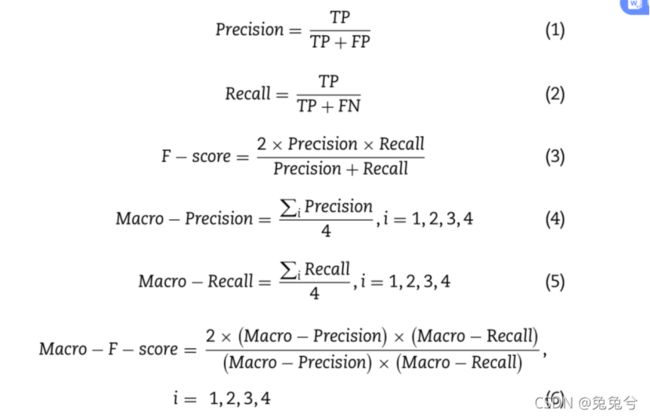
4.评估标准
评估标准包括总体Acc、宏观平均精度、宏观平均召回率和宏观平均F分数,根据四个标签分类中各自的公式计算得出:
其中,TP和FP分别表示一类中的真预测数和假预测数,TN和FN分别表示其余三类中的真预测数和假预测数。
4.结果
1.LncRNA-生物分子相互作用的统计与分析
我们统计分析了与lncRNAs相互作用的生物分子的类型,并根据NPInter v4.0数据库中提供的原始数据分析了lncRNAs的功能。与lncRNA–DNA相互作用相关的数据量远低于lncRNA–RNA和lncRNA–蛋白质相互作用的数据量。在lncRNA–RNA相互作用过程中,lncRNA主要与miRNA相互作用,但也有一些与mRNA相关;然而,他们中只有少数,少于1.5%的lncRNA与其他类型的ncRNA相互作用(图5A)。此外,大多数lncRNA通过与蛋白质和DNA结合与它们相互作用,而在lncRNA与RNA相互作用期间,大多数lncRNA遵循结合和调节途径(图5B)。此外,据报道发现一些lncRNA与多个生物分子相互作用。例如,lncRNA HELLPAR与多种RNA分子相互作用;lncRNA AC007319.1与多种蛋白质相互作用;lncRNA XIST与多个RNA和蛋白质分子相互作用,lncRNA NEAT1与多个RNA和DNA分子相互作用。然而,一些lncRNA仅与单个生物分子相互作用。同时,据报道发现类似WDR33的蛋白质与多个lncRNAs相互作用。根据本研究前面定义的四个类别,我们数据集中的lncRNA-生物分子相互作用的一部分如图5C所示。
图5.lncRNA–生物分子相互作用类型和功能分析(A) 人类lncRNA–RNA相互作用中的RNA类型,其中数据从NPInter v4.0中提取数据库(B) lncRNA-生物分子相互作用中相互作用模式数量的log2表示,其中数据来自NPInter 4.0。(C)我们的数据集中的相互作用的演示,以及四个类别的部分数据;从左到右:lncRNAs与RNA、蛋白质、RNA&蛋白质和DNA相关蛋白相互作用
2.lncIBTP的初步培训以及评价
为了观察是否可以通过降低特征维数来进一步提高性能,我们采用F分数和卡方检验法来进行特征选择。对于每种方法,我们对不同大小的特征子集进行三次验证,并在补充图S3中记录其平均精度。然而,在这四类分类问题中,所选择的特征子集并没有显示出明显的优势,使用整个特征集可以获得最佳性能。因此,我们决定使用整个特征集来训练模型。
为了确定集成模型的数量,我们在每次验证中训练了10个模型,并选择了最佳的n个模型进行集成。我们选择了n=3,5,7,10,并重点研究了两类小数据量相互作用(RNA和DNA相关)的预测性能。我们最终选择了n=5,这导致了整体最佳性能,两个等级的平均曲线下面积(AUC)分别为0.723和0.834(补充表S1)。
图S3。特征选择的性能。上限:采用卡方检验法选择特征的准确度,下限:采用F评分法选择特征的准确度。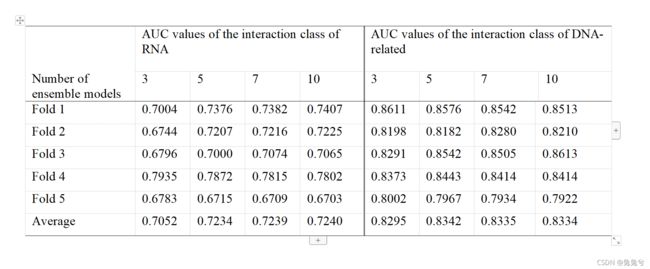
表S1。不同集合模型数下的性能
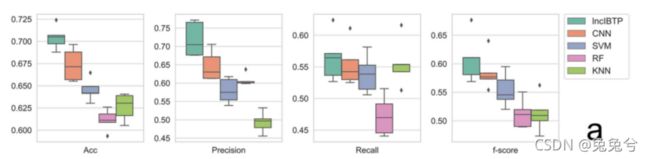
为了评估我们开发的预测因子lncIBTP区分lncRNA和生物分子类型之间相互作用的能力,我们进行了5倍交叉验证。每个班级的样本被随机分为五组。对于每次验证,选择一组数据作为测试数据,而其余四组数据作为训练数据。我们将lncIBTP的结果与其他基线分类算法进行比较,如支持向量机(SVM)、随机森林(RF)和k-最近邻(KNN)在方框和胡须图中,其中可以显示模型倾斜(图6A)。通过在参数网格上交叉验证网格搜索,将基线模型的参数确定为最优参数。每个基线模型的候选参数和最终确定的参数记录在补充表S2中。
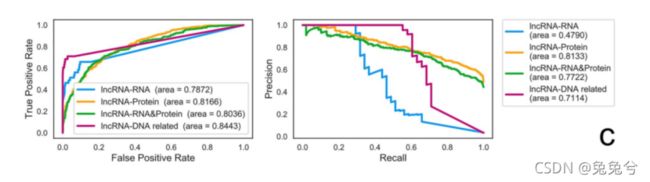
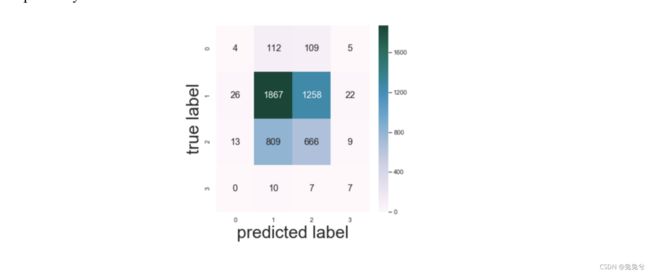
图6.lncIBTP方法的评估(A) lncIBTP和基线方法的5倍交叉验证的盒状图和晶须图(B) 5倍交叉验证的混淆矩阵。从左到右:折叠1-5作为测试数据,x轴:预测标签,y轴:真实标签,类别0-3分别代表与RNA、蛋白质、RNA&蛋白质和DNA相关的lncRNAs相互作用(C) 第一次验证的四个类别的ROC(左)和PRC(右)。
3.与特定lncRNA分子预测方法的比较
lncRNA-分子相互作用预测中的一些工具可以预测lncRNA的候选伙伴;因此,我们可以根据这些预测推断lncRNA与之相互作用的生物分子的类型。我们将lncIBTP的结果与RNAInter的预测结果进行了比较。RNAInter是一个RNA相互作用组数据库。。除了实验证据外,RNA还可以输出计算预测的相互作用分子,用于输入lncRNA与lncRNA–RNA、lncRNA–蛋白质和lncRNA–DNA之间的相互作用,并获得保密分数。我们搜索了数据集中每个lncRNA的所有三种相互作用类型的计算预测结果。如果预测任何分子在相应的相互作用中与lncRNA相互作用的分数大于0.5,则该lncRNA被视为与该类型的生物分子相互作用。补充图S5中绘制了RNAInter对5倍验证数据集预测结果的混淆矩阵。RNAInter预测的平均Acc为0.535,比lncIBTP低约0.169。当我们使用lncIBTP作为前步来定义lncRNA相互作用生物分子的类型时,RNAInter预测的正确预测lncRNAs的相应类型的分子约为92.32%(平均结果为5倍),分数大于0.5。
图S5。来自RNAInter的混淆矩阵预测结果。类别0至3分别代表与RNA、蛋白质、RNA&蛋白质和DNA相关的lncRNA相互作用。
4.个案研究和跨物种验证
我们还构建了一个新的独立测试数据集来进一步评估lncIBTP的能力。我们从NCBI核苷酸数据库下载可用的序列数据。新数据集包括四个新的lncRNA,包括RAIN、SOCAR、LINC01980和LINC02407。据报道,lncRNA RAIN和SOCAR与蛋白质相互作用,LINC02407与miRNA相互作用,LINC01980与miRNA和蛋白质相互作用。我们使用lncIBTP识别它们相互作用的生物分子类型;lncIBTP预测RAIN、LINC01980和LINC02407与RNA&蛋白质相互作用,并预测SOCAR与蛋白质相互作用。lncIBTP的预测结果与报道的相互作用类型基本一致。虽然RAIN 仅与蛋白质相互作用,而LINC02407仅与miRNA相互作用,但我们的预测结果表明它们可能与RNA和蛋白质相互作用。
通过使用由所有人类数据训练的模型来测试小鼠数据来进行跨物种验证。小鼠数据的总Acc为0.517。如混淆矩阵(补充图S6)所示,小鼠数据预测的趋势与人类数据的趋势相似。在这里,跨物种验证的性能不是很好。一个可能的原因是人类和老鼠物种的特征不同。我们可视化了两个物种中四类的特征热图(补充图S7)。两种动物的某些特征表现出明显的差异。例如,人类数据中RNA、蛋白质、RNA&蛋白质和DNA相关相互作用类别的“CGC”(第1行第7列,从0开始排序)的平均频率分别为0.0045、0.0042、0.0051和0.0045,其中最高值在RNA&蛋白质类别中。然而,在小鼠数据中,相应四个类别的“CGC”平均频率分别为0.0057、0.0051、0.0053和0.0082,其中所有值均大于人类,且最高值在DNA相关类别中。为了提高鼠标的性能,可能的解决方案是调整人体模型中的参数。然而,在调整模型之前,预计会有更多的实验数据,因为目前小鼠中与DNA相关的数据量非常小。i.e.24数据集。
图S6。用于小鼠数据跨物种验证的混淆矩阵。类别0至3分别代表与RNA、蛋白质、RNA&蛋白质和DNA相关的lncRNA相互作用。
图S7。人类和鼠标数据集中4个类的特征热图。上图:人类数据,下图:鼠标数据。从左到右的热图分别对应于RNA、蛋白质、RNA&蛋白质和DNA相关的类别。原始特征(3-mer、2-bigap和3-bigap)被重新塑造为32*18的大小。
5.讨论
由于其复杂的分子机制,预测lncRNAs的功能具有挑战性。为了破解lncRNA功能的密码,我们重点研究了三种lncRNA-生物分子相互作用,lncRNA–RNA、lncRNA–蛋白质和lncRNA–DNA。首先,我们分别综述了lncRNA与RNA、蛋白质和DNA相互作用的主要机制。然后,我们提出了一个集成的深度学习模型,即lncIBTP。该模型用于确定我们是否能够预测lncRNA与不同类型的生物分子之间的相互作用,RNA,蛋白质,RNA&蛋白质和DNA相关。结果表明,lncIBTP可有效识别特定lncRNA与不同类型生物分子之间可能的相互作用。在5倍交叉验证中,其Acc、宏观平均召回率、宏观平均精密度和宏观平均F分数分别在0.688到0.724、0.527到0.624、0.676到0.772和0.569到0.677之间。Acc的窄范围分布也表明lncIBTP的健壮性。此外,小鼠数据跨物种验证的总Acc为0.517;Acc相对较低的一个可能原因是人类和小鼠之间的特征不同。此外,基于混淆矩阵、ROC和PRC以及收集的数据,我们得出以下假设:与DNA相互作用的lncRNAs的特征可能不同于仅与RNA相互作用的lncRNAs。
在未来的应用中,lncIBTP可作为特定lncRNA-分子相互作用的预步骤,以提高预测新发现的lncRNA功能的效率。此外,由于一些lncRNA-生物分子相互作用在疾病中起着重要作用,lncIBTP的应用也可能有助于提高lncRNA-疾病预测的性能。
然而,lncIBTP有一个局限性:它对lncRNA与RNA相互作用的预测性能不如其他类别。一个主要原因是lncRNA仅与RNA相互作用的数据量有限。因此,我们建议应探索更多数据,以帮助进一步实施该模型。此外,已有研究报道lncRNA的功能与其二级结构有关。因此,lncRNA的二级结构特征应通过lncIBTP模型进一步研究。