NLP学习(十五)-NLP实战之基于K-Means文本聚类-Python3
何为聚类
简单理解,如果一个数据集合包含N个实例,根据某种准则可以将这N个实例划分为m个类别,每个类别中的实例都是相关的,而不同类别之间是区别的也就是不相关的,这个过程就叫聚类了。
聚类过程
1)特征选择(feature selection):就像其他分类任务一样,特征往往是一切活动的基础,如何选取特征来尽可能的表达需要分类的信息是一个重要问题。表达性强的特征将很影响聚类效果。这点在以后的实验中我会展示。
2)近邻测度(proximity measure):当选定了实例向量的特征表达后,如何判断两个实例向量相似呢?这个问题是非常关键的一个问题,在聚类过程中也有着决定性的意义,因为聚类本质在区分相似与不相似,而近邻测度就是对这种相似性的一种定义。
3)聚类准则(clustering criterion):定义了相似性还不够,结合近邻测度,如何判断相似才是关键。直观理解聚类准则这个概念就是何时聚类,何时不聚类的聚类条件。当我们使用聚类算法进行计算时,如何聚类是算法关心的,而聚与否需要一个标准,聚类准则就是这个标准。(话说标准这东西一拿出来,够吓人了吧_)
4)聚类算法(clustering algorithm):这个东西不用细说了吧,整个学习的重中之重,核心的东西这里不讲,以后会细说,简单开个头——利用近邻测度和聚类准则开始聚类的过程。
5)结果验证(validation of the results):其实对于PR的作者提出这个过程也放到聚类任务流程中,我觉得有点冗余,因为对于验证算法的正确性这事应该放到算法层面吧,可以把4)和5)结合至一层。因为算法正确和有穷的验证本身就是算法的特性嘛。(谁设计了一个算法不得证明啊)
6)(interpretation of the results):中文版的PR上翻译为结果判定,而我感觉字面意思就是结果解释。(聚类最终会将数据集分成若干个类,做事前要有原则,做事后要有解释,这个就是解释了。自圆其说可能是比较好的了_)
聚类准则
聚类准则就是一个分类标准,对于示例中这样一个数据集合,如何聚类呢。当然聚类的可能情况有很多。比如,如果我们按照年级是否为大于1来分类,那么数据集X分为两类:{张三},{李四,张飞,赵云};如果按照班级不同来分,分为两类:{张三,李四},{张飞,赵云};如果按照成绩是否及格来分(假设及格为60分),分两类:{张三,李四,赵云},{张飞}。当然聚类准则的设计往往是复杂的,就看你想怎么划分了。按照对分类思想的几何理解,数据集相当于样本空间,数据实例的特征数(本例共有4个特征[姓名,年级,班级,数学成绩])相当于空间维度,而实例向量对应到空间中的一个点。那么聚类准则就应该是那些神奇的超平面(对应有数学函数表达式,我个人认为这些函数就等同于聚类准则),这些超平面将数据“完美的”分离开了。
聚类特征类型
聚类时用到的特征如何区分呢,有什么类型要求?聚类的特征按照域划分,可以分为连续的特征和离散特征。其中连续特征对应的定义域是数据空间R的连续子空间,而离散特征对应的是离散子集,另外如果离散特征只包含两个特征值,那么这个离散特征又叫二值特征。 根据特征取值的相对意义又可以将特征分为以下四种:标量的(Nominal),顺序的(Ordinal),区间尺度的(Interval-scaled)以及比率尺度的(Ratio-scaled)。其中,标量特征用于编码一类特征的可能状态,比如人的性别,编码为男和女;天气状况编码为阴、晴和雨等。顺序特征同标量特征类似,同样是一系列状态的编码,只是对这些编码稍加约束,即编码顺序是有意义的,比如对一道菜,它的特征有{很难吃,难吃,一般,好吃,美味}几个值来定义状态,但是这些状态是有顺序意义的。这类特征我认为就是标量特征的一个特定子集,或者是一个加约束的标量特征。区间尺度特征表示该特征数值之间的区间有意义而数值的比率无意义,经典例子就是温度,A地的温度(20℃)比B地(15℃)高5度,这里的区间差值是有意义的,但你不能说A地比B地热1/3,这是无意义的。比率特征与此相反,其比率是有意义的,经典例子是重量,C重100g,D重50g,那么C比D重2倍,这是有意义的。(当然说C比D重50g也是可以的,因此可以认为区间尺度是比率尺度的一个真子集)。
在常见应用中,包括我们平日关心的编程实现中,一般只定义nominal特征和numeric特征,其中nominal可以用string来表示,而numeric可以用number来表示。(weka中的attribute的特征类型就是这么定义的)!!weka是个好东西WEKA使用教程.pdf
聚类算法的分类
划分方法
划分方法就是根据用户输入值k把给定对象分成k组(满足2个条件:1. 每个组至少包含一个对象。2. 每个对象必须且只属于一个组),每组都是一个聚类,然后利用循环再定位技术变换聚类里面的对象,直到客观划分标准(常成为相似函数,如距离)最优为止。典型代表:k-means, k-medoids 层次的方法
层次的方法对给定的对象集合进行层次分解。分为2类:凝聚的和分裂的。凝聚的方法也叫自底向上的方法,即一开始将每个对象作为一个单独的簇,然后根据一定标准进行合并,直到所有对象合并为一个簇或达到终止条件为止。分裂的方法也叫自顶向下的方法,即一开始将所有对象放到一个簇中,然后进行分裂,直到所有对象都成为单独的一个簇或达到终止条件为止。典型代表:CURE,BIRCH
基于密度的方法
基于密度的方法即不断增长所获得的聚类直到邻近(对象)密度超过一定的阀值(如一个聚类中的对象数或一个给定半径内必须包含至少的对象数)为止。典型代表:DBSCAN,OPTICS。
基于网格的方法
基于网格的方法即将对象空间划分为有限数目的单元以形成网格结构。所有聚类操作都在这一网格结构上进行。典型代表:STING。
基于模型的方法
基于模型的方法即为每个聚类假设一个模型,然后按照模型去发现符合的对像。这样的方法经常基于这样的假设:数据是根据潜在的概率分布生成的。主要有2类:统计学方法和神经网络方法。典型代表:COBWEB,SOMs
何为k-means
K-means算法是很典型的基于距离的聚类算法,采用距离作为相似性的评价指标,即认为两个对象的距离越近,其相似度就越大。该算法认为簇是由距离靠近的对象组成的,因此把得到紧凑且独立的簇作为最终目标。
k个初始类聚类中心点的选取对聚类结果具有较大的影响,因为在该算法第一步中是随机的选取任意k个对象作为初始聚类的中心,初始地代表一个簇。该算法在每次迭代中对数据集中剩余的每个对象,根据其与各个簇中心的距离将每个对象重新赋给最近的簇。当考察完所有数据对象后,一次迭代运算完成,新的聚类中心被计算出来。如果在一次迭代前后,J的值没有发生变化,说明算法已经收敛。
算法过程
1)从N个文档随机选取K个文档作为质心
2)对剩余的每个文档测量其到每个质心的距离,并把它归到最近的质心的类
3)重新计算已经得到的各个类的质心
4)迭代2~3步直至新的质心与原质心相等或小于指定阀值,算法结束
说明如下:首先从n个数据对象任意选择 k 个对象作为初始聚类中心;而对于所剩下其它对象,则根据它们与这些聚类中心的相似度(距离),分别将它们分配给与其最相似的(聚类中心所代表的)聚类;然 后再计算每个所获新聚类的聚类中心(该聚类中所有对象的均值);不断重复这一过程直到标准测度函数开始收敛为止。一般都采用均方差作为标准测度函数. k个聚类具有以下特点:各聚类本身尽可能的紧凑,而各聚类之间尽可能的分开。
具体如下:
输入:k, data[n];
(1) 选择k个初始中心点,例如c[0]=data[0],…c[k-1]=data[k-1];
(2) 对于data[0]….data[n],分别与c[0]…c[k-1]比较,假定与c[i]差值最少,就标记为i;
(3) 对于所有标记为i点,重新计算c[i]={ 所有标记为i的data[j]之和}/标记为i的个数;
(4) 重复(2)(3),直到所有c[i]值的变化小于给定阈值。
k-means 算法缺点
① 在 K-means 算法中 K 是事先给定的,这个 K 值的选定是非常难以估计的。很多时候,事先并不知道给定的数据集应该分成多少个类别才最合适。这也是 K-means 算法的一个不足。有的算法是通过类的自动合并和分裂,得到较为合理的类型数目 K,例如 ISODATA 算法。关于 K-means 算法中聚类数目K 值的确定在文献中,是根据方差分析理论,应用混合 F 统计量来确定最佳分类数,并应用了模糊划分熵来验证最佳分类数的正确性。在文献中,使用了一种结合全协方差矩阵的 RPCL 算法,并逐步删除那些只包含少量训练数据的类。而文献中使用的是一种称为次胜者受罚的竞争学习规则,来自动决定类的适当数目。它的思想是:对每个输入而言,不仅竞争获胜单元的权值被修正以适应输入值,而且对次胜单元采用惩罚的方法使之远离输入值。
② 在 K-means 算法中,首先需要根据初始聚类中心来确定一个初始划分,然后对初始划分进行优化。这个初始聚类中心的选择对聚类结果有较大的影响,一旦初始值选择的不好,可能无法得到有效的聚类结果,这也成为 K-means算法的一个主要问题。对于该问题的解决,许多算法采用遗传算法(GA),例如文献 中采用遗传算法(GA)进行初始化,以内部聚类准则作为评价指标。
③ 从 K-means 算法框架可以看出,该算法需要不断地进行样本分类调整,不断地计算调整后的新的聚类中心,因此当数据量非常大时,算法的时间开销是非常大的。所以需要对算法的时间复杂度进行分析、改进,提高算法应用范围。在文献中从该算法的时间复杂度进行分析考虑,通过一定的相似性准则来去掉聚类中心的侯选集。而在文献中,使用的 K-means 算法是对样本数据进行聚类,无论是初始点的选择还是一次迭代完成时对数据的调整,都是建立在随机选取的样本数据的基础之上,这样可以提高算法的收敛速度。
何为TD-IDF
TD-IDF(term frequency /inverse document frequency)不止一次看到这个概念了,让我印象最深的是吴军在《数学之美》中对这个概念简约的描述。数学之美.pdf
TF-IDF(term frequency–inverse document frequency)是一种用于资讯检索与资讯探勘的常用加权技术。TF-IDF是一种统计方法,用以评估一字词对于一个文件集或一个语料库中的其中一份文件的重要程度。字词的重要性随着它在文件中出现的次数成正比增加,但同时会随着它在语料库中出现的频率成反比下降。
TF-IDF算法是建立在这样一个假设之上的:对区别文档最有意义的词语应该是那些在文档中出现频率高,而在整个文档集合的其他文档中出现频率少的词语,所以如果特征空间坐标系取TF词频作为测度,就可以体现同类文本的特点。另外考虑到单词区别不同类别的能力,TFIDF法认为一个单词出现的文本频数越小,它区别不同类别文本的能力就越大。因此引入了逆文本频度IDF的概念,以TF和IDF的乘积作为特征空间坐标系的取值测度,并用它完成对权值TF的调整,调整权值的目的在于突出重要单词,抑制次要单词。
k-means 的文本聚类
前面介绍了TD-IDF我们可以通过用TD-IDF衡量每个单词在文件中的重要程度。如果多个文件,它们的文件中的各个单词的重要程度相似,我就可以说这些文件是相似的。如何评价这些文件的相似度呢?一种很自然的想法是用两者的欧几里得距离来作为相异度,欧几里得距离的定义如下:

其意义就是两个元素在欧氏空间中的集合距离,因为其直观易懂且可解释性强,被广泛用于标识两个标量元素的相异度。我们可以将X,Y分别理解为两篇文本文件,xi,y是每个文件单词的TD-IDF值。这样就可以算出两文件的相似度了。这样我们可以将文件聚类的问题转化为一般性的聚类过程,样本空间中的两点的距离可以欧式距离描述。除欧氏距离外,常用作度量标量相异度的还有曼哈顿距离和闵可夫斯基距离,两者定义如下:
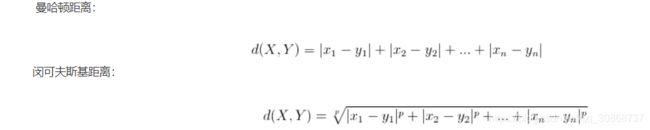
整个文本聚类过程可以先后分为两步:1、计算文本集合各个文档中TD-IDF值,2,根据计算的结果,对文件集合用k-means聚类方法进行迭代聚类。
TD-IDF的计算
假设文档集合T ={n|tn, n>1}。
- 对文档进行分词或Tokennize处理,去掉停用词。
- 计算各个词出现的次数freq(wi),则TF(i) = freq(wi)/sum( freq(w1…n)) ,有时候sum( freq(w1…n))可以用max( freq(w1…n)),做归一处理。
- 统计文件集合中有词wj出现的文件个数doc_freq(wj).则IDF= 1/log(doc_freq(wj);
- 根据上面的计算我们可以算出文件i,单词wj的TD-IDF权值W[i][j]= TD(j)*IDF(j)。其中i为文件集合T中的一个文件,而j是文件集合T中的一个单词。
通过对文件集合T的计算我们可以得到一个二维数组(矩阵)W[i][j].
K-means聚类的迭代过程
- 随机选取k个文件生成k个聚类cluster,k个文件分别对应这k个聚类的聚类中心Mean(cluster) = k ;对应的操作为从W[i][j]中0~i的范围内选k行(每一行代表一个样本),分别生成k个聚类,并使得聚类的中心mean为该行。
- 对W[i][j]的每一行,分别计算它们与k个聚类中心的距离(通过欧氏距离)distance(i,k)。
- 对W[i][j]的每一行,分别计算它们最近的一个聚类中心的n(i) = ki。
- 判断W[i][j]的每一行所代表的样本是否属于聚类,若所有样本最近的n(i)聚类就是它们的目前所属的聚类则结束迭代,否则进行下一步。
- 根据n(i) ,将样本i加入到聚类k中,重新计算计算每个聚类中心(去聚类中各个样本的平均值),调到第2步。
算法代码实例
# -*- coding: utf-8 -*-
import sys, os
import numpy as np
from numpy import *
import jieba
import math
import jieba.analyse
jieba.load_userdict("userdict.txt")
def read_from_file(file_name):
with open(file_name, "r") as fp:
words = fp.read()
return words
def stop_words(stop_word_file):
words = read_from_file(stop_word_file)
result = jieba.cut(words)
new_words = []
for r in result:
new_words.append(r)
return set(new_words)
def gen_sim(A, B):
num = float(np.dot(A, B.T))
denum = np.linalg.norm(A) * np.linalg.norm(B)
if denum == 0:
denum = 1
cosn = num / denum
sim = 0.5 + 0.5 * cosn
return sim
def del_stop_words(words, stop_words_set):
result = jieba.cut(words)
new_words = []
for r in result:
if r not in stop_words_set:
new_words.append(r)
# print r.encode("utf-8"),
# print len(new_words),len(set(new_words))
return new_words
def tfidf(term, doc, word_dict, docset):
tf = float(doc.count(term)) / (len(doc) + 0.001)
idf = math.log(float(len(docset)) / word_dict[term])
return tf * idf
def idf(term, word_dict, docset):
idf = math.log(float(len(docset)) / word_dict[term])
return idf
def word_in_docs(word_set, docs):
word_dict = {}
for word in word_set:
# print word.encode("utf-8")
word_dict[word] = len([doc for doc in docs if word in doc])
# print word_dict[word],
return word_dict
def get_all_vector(file_path, stop_words_set):
names = [os.path.join(file_path, f) for f in os.listdir(file_path)]
posts = [open(name).read() for name in names]
docs = []
word_set = set()
for post in posts:
doc = del_stop_words(post, stop_words_set)
docs.append(doc)
word_set |= set(doc)
# print len(doc),len(word_set)
word_set = list(word_set)
docs_vsm = []
# for word in word_set[:30]:
# print word.encode("utf-8"),
for doc in docs:
temp_vector = []
for word in word_set:
temp_vector.append(doc.count(word) * 1.0)
# print temp_vector[-30:-1]
docs_vsm.append(temp_vector)
docs_matrix = np.array(docs_vsm)
# print docs_matrix.shape
# print len(np.nonzero(docs_matrix[:,3])[0])
column_sum = [float(len(np.nonzero(docs_matrix[:, i])[0])) for i in range(docs_matrix.shape[1])]
column_sum = np.array(column_sum)
column_sum = docs_matrix.shape[0] / column_sum
idf = np.log(column_sum)
idf = np.diag(idf)
# print idf.shape
# row_sum = [ docs_matrix[i].sum() for i in range(docs_matrix.shape[0]) ]
# print idf
# print column_sum
for doc_v in docs_matrix:
if doc_v.sum() == 0:
doc_v = doc_v / 1
else:
doc_v = doc_v / (doc_v.sum())
tfidf = np.dot(docs_matrix, idf)
return names, tfidf
def randCent(dataSet, k):
n = shape(dataSet)[1]
centroids = mat(zeros((k, n))) # create centroid mat
for j in range(n): # create random cluster centers, within bounds of each dimension
minJ = min(dataSet[:, j])
rangeJ = float(max(dataSet[:, j]) - minJ)
centroids[:, j] = mat(minJ + rangeJ * random.rand(k, 1))
return centroids
def kMeans(dataSet, k, distMeas=gen_sim, createCent=randCent):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m, 2))) # create mat to assign data points
# to a centroid, also holds SE of each point
centroids = createCent(dataSet, k)
clusterChanged = True
counter = 0
while counter <= 50:
counter += 1
clusterChanged = False
for i in range(m): # for each data point assign it to the closest centroid
minDist = inf;
minIndex = -1
for j in range(k):
distJI = distMeas(centroids[j, :], dataSet[i, :])
if distJI < minDist:
minDist = distJI;
minIndex = j
if clusterAssment[i, 0] != minIndex:
clusterChanged = True
clusterAssment[i, :] = minIndex, minDist ** 2
# print centroids
for cent in range(k): # recalculate centroids
ptsInClust = dataSet[nonzero(clusterAssment[:, 0].A == cent)[0]] # get all the point in this cluster
centroids[cent, :] = mean(ptsInClust, axis=0) # assign centroid to mean
return centroids, clusterAssment
if __name__ == "__main__":
stop_words = stop_words("./stop_words.txt")
names, tfidf_mat = get_all_vector("./chinese/", stop_words)
myCentroids, clustAssing = kMeans(tfidf_mat, 3, gen_sim, randCent)
for label, name in zip(clustAssing[:, 0], names):
print (label, name)
参考
Kmeans豆瓣书籍文本聚类实战
机器学习-文本聚类实例-kmeans