Multitask Classification of Breast Cancer Pathological Images Using SE-DenseNet
摘要:
本文采用密集连接的卷积网络(DenseNet)结构,吸收SeNet,对一组苏木精和伊红(H&E)染色乳腺组织学显微镜 Camelyon16 进行多任务分类。全幻灯片图像(WSI)通常存储在多分辨率金字塔中,我们的数据集包含Camelyon16 在*5、*20、*40,三倍放大率下的patches。 我们的多任务是通过连接同一网络末端的两个分类器来识别patches的放大率并区分提取的patch是否属于WSI的转移性肿瘤区域。无论是在多任务还是单任务上,我们的网络都表现出了良好的性能,SE-DenseNet-40在CIFAR-10上的准确率更是达到了92.92%。
METHODOLOGY
我们自己设计的四个网络:DenseNet-22, DenseNet-40, SEDenseNet-22, SE-DenseNet-40
转移学习:DenseNet-121、DenseNet-169、DenseNet-201来检测Camelyon16的转移瘤区域。
转移学习的训练方法:
第一种:转移学习通过使用pre-trained densenet - 121 densenet - 169 densenet - 201(所有三个网络没有最高层)分别在我们的数据集上提取特征,然后训练一个新的2类分类小网络(两个FC层1024和2节点)。
第二种:去掉预先训练好的网络的最后一个FC层,用两个新的FC层(1024和2个节点)替代。在两个新的FC层之前冻结所有层,然后在我们的数据集上进行训练。与第一种方法相比,第二种方法的效率很低,因为每个patch都要迭代几次。
三个任务:
第一个:要求将(每次放大后的)数据集的patch分为两类:转移性肿瘤补片和正常补片。应用经典的数据集CIFAR-10训练我们的四个网络来验证网络的性能。
第二个:与第一个一样分为两类,唯一的区别在于,训练集是三个不同的放大下的和的数据集
第三个:首先在每个放大了的训练集上添加一个额外的标签来描述放大率(5 is 0,20 is 1,40 is 2),然后混合组成新的数据集。与前两个任务的网络架构相比,在网络末尾添加了一个three-class分类器,这个分类器将放大倍数分为三类:*5,*20和*40。训练损失是两个分类器损失的总和, 损失的权重设置为超参数。
Model
6 convolutional layers of DenseNet-22
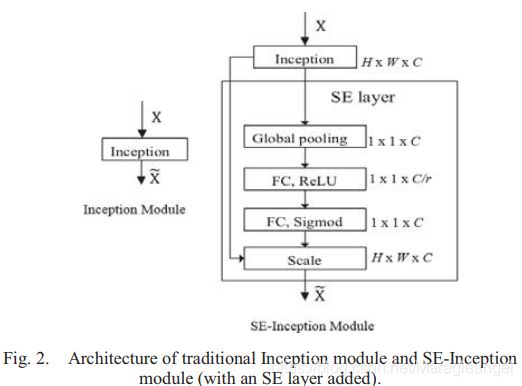
SE-DenseNet:
注:我们在SE-DenseNet-22和SE-DenseNet-40中应用SE-PRE块[3]。
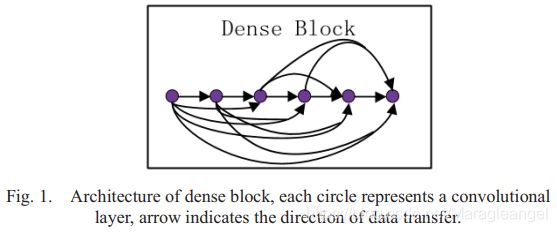
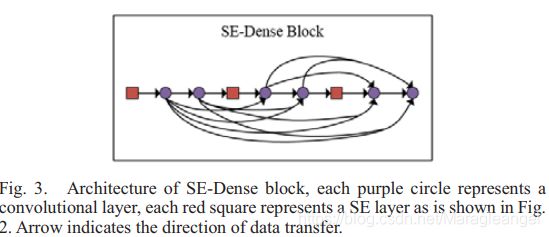
SEDense block见下图:
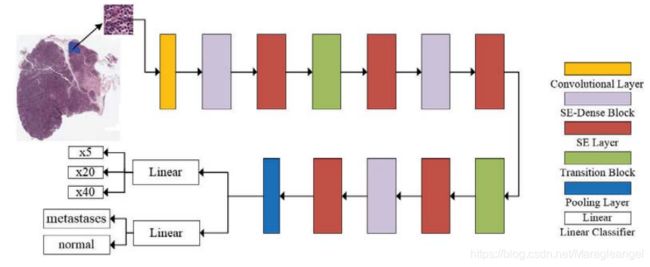
多任务分类框架的SE-DenseNet-22体系结构见下图。
黄色块表示卷积层,紫色块表示图3所示的SE层,,红色块表示图2所示的SE层。绿色块表示转换块,每个转换块由一个卷积层和一个池化层组成。蓝色块表示池化层。Linear是线性分类器,输入是网络中生成的特征,输出是输入patch的标签。与SE-DenseNet相比,DenseNet中不存在SE层。
block==3,growth rate==12(growth rate是指与前一个convolutional layer相比在当前的convolutional layer中加入的filter的数量)因为我们四个network都是为了防止network增长的太宽,提高parameter efficiency 。
每个dense block 或SE-dense block包含相同数量的卷积层,每个block的卷积层数等于(N-4)/3,(N为网络的总层数,分别设置为22和40)。dense block和SE-dense block的唯一区别是在SE-dense block中每两个卷积层之前添加一个SE层。
初始滤波器数量是指在我们的四个网络中将第一个dense block设置为24之前,开始卷积层的滤波器数量。
Dataset
训练数据集包含两个部分
第一个训练数据集包含170个WSIs of lymph node(100个正常的载玻片和70个有转移的载玻片)
第二个训练数据集包含100个WSIs(60个正常的载玻片和40个有转移的载玻片)。
patches: *5 128×128 pixels 24919 patches 80% 20%
*20 256 ×256 pixels 111008 patches 90% 10%
*40 256 ×256 pixels 136744 patches 90% 10%
Training
我们首先在三次放大下分别对我们的四个网络在CIFAR-10和Camelyon16训练,然后在Camelyon16上应用转移学习。
第二个任务,首先应该将128*128个补丁调整为256*256,与256*256个补丁混合在一起,形成新的训练集来训练我们的四个网络。
第三个任务:根据第二个任务生成的训练数据,我们*5= 0,*20=1,*40=2,然后应用新的数据集来训练我们的多任务分类模型。