ChimeraX cryoEM 可视化教程:细菌ATP合酶
ChimeraX cryoEM 可视化教程:细菌ATP合酶
翻译自:https://www.rbvi.ucsf.edu/chimerax/data/stanford-jul2019/tutorial.html
这篇ChimeraX教程将着眼于如何可视化细菌ATP合成酶在3.0和3.2 A分辨率下由CryoEM测定的三种构象的原子模型和maps。我们将创建与2019年2月出版物中类似的数据视图。

原子模型PDB 6n2y、6n2z、6n30。EMDB map 9333,9334,9335。ChimeraX 可以直接从PDB和EMDB获取这些数据文件,也可以下载这些文件以供offline脱机使用。
Topic
- 基础:打开、移动、样式、链…
- 在3种构象之间变形 Morphing。
- 查看结合的ADP、磷酸盐、ATP。
- 检查侧链密度、区域zones。
- 将原子模型拟合到地图中 Fit。
- 查看质子通道。
- 查看单向旋转。
Basics: open, movement, styles, chains…
- 命令“open 6n2y”,或工具栏的“Open”按钮,或“File/Open”菜单从本地驱动加载文件。
- Log chain和ligand table:单击letter选择原子,单击名称显示sequence。

显示序列或用命令:
seq chain /a
seq chain #1/a
(黄色是螺旋部分,蓝色是beta折叠区,白框是结构中缺失部分)

-
移动:Mac触控板、2指旋转、3指平移、4指缩放(或捏)。
鼠标,左键旋转,右键平移,滚轮缩放。 -
旋转中心:在曲面上关闭,或在缩小时位于模型中心。
-
详细信息…log link,显示publication、species、experimental method和resolution。
其他-不确定在开始和之后显示多少图像。
- 工具栏图标:保存图像。隐藏/显示原子/卡通、样式、背景颜色、照明模式、摩尔mol显示、着色、hbonds。
- 使用鼠标选择,或选择菜单、up arrow promotion、所选内容的工具栏操作。
- 预设。柱面Cylinder显示很酷,再加上显示ligands和保存图像。让他们尝试不同的样式。
- 帮助菜单Help menu、用户指南user’s guide。
Morphing between 3 conformations
open 6n2z;open 6n2z
Model Panel: show/hide按钮以显示或隐藏每个模型。
morp #1,2 前两个结构之间变形。试试滑块上的播放按钮。可能需要使用simple lighting来提高速度。
关闭morph,单击模型面板中的model4,然后按close按钮。
在三种构象之间morph,并结束在开始的那个:“morph #1,2,3 wrap true same true”
“wrap true”选项返回到起始模型。
“same true”选项表示不使用序列比对算法,而是假定相同的序列以加快计算速度。
morph在每个结构之间有20 steps,若要增加此设置,请参阅使用命令“help morph”的文档。
按滑块右侧的红色圆圈录制movie。
Close morph
Viewing bound ADP, phosphate, ATP
打开CryoEM map,“open 9333 from emdb”,或者用工具栏Open按钮或File/Open从本地上打开文件。
Toolbar, molecule display, stick style.
Volume step 2 to step 1。
手动调整直方图上的阈值。可能需要simple lighting以实现更快的updates。
使用直方图上的color按钮使surface透明。
居中ADP并使用clip planes命令:“view :ADP”
网格样式Mesh style:用histogram上的菜单选择样式。
显示所有内容:view all
磷酸盐在哪,用命令“sel :ADP:PO4”显示它们位于不同的结合位点。不确定原因。
仅在lower contour level查看“tentatively assigned”磷酸盐phosphate周围的密度用命令:“view :PO4”。
view all
Inspecting side-chain density, zones
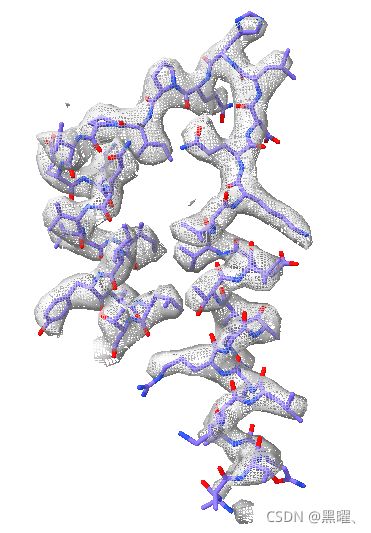
eLife的Figure 1 supplement 4展示了侧链的cryoEM密度示例。
显示链A残基404-441附近的密度。
命令“surface zone #4 near #1/A:404-441”
隐藏所有原子:使用工具栏图标或命令“hide #1”
显示原子:“show #1/A:404-441”
分子工具栏,color “heteroatom”。
要制作finer mesh用命令:“volume #4 subdivide true”和“volume #4 subdivide true subdivisionlevels 2”
volume #4 level 1.5
图形工具栏白色背景。
“vol #4 subdivide false”避免之后surface计算慢
Fitting atomic model into map
建模一开始通常是将现有模型fit到map上。
将3.9A X射线结构4xd7(复合体的F1一半)fit map。
命令:“open 4xd7”
按住ctrl键并单击以选择4xd7原子。
使用工具栏Right Mouse的平移translate和旋转rotate将模型放入map中。
使用map里的menu Tools / Volume Data / Fit 来优化4xd7与9333的拟合。
map中的fine detail导致无法找到good global fit,因此用smoothed map去fit。
用命令“volume gaussian #4 sdev 3”
Fit到此map有更大的收敛半径。
仍需要获得正确的6-fold对称对齐才能匹配轴亚基。
有时可以color map来匹配附近的原子:“color zone #4 near #5 dist 8”
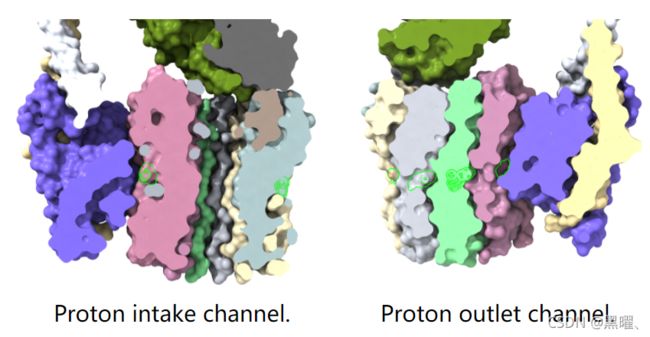
Look at proton channels
看看驱动motor的质子路径,如eLife论文中的图4所示。
跨膜鼓在谷氨酸E56上携带质子:“select /c*:56”。作为球体sphere显示。
Drum在膜中,质子从下侧沿“a”亚基之间的通道向上移动,到达鼓上,绕转一圈,释放到另一个通道,从膜的另一侧退出。
分子工具栏,显示surface。
在左侧是长螺旋杆定向视图Orient view。
在工具栏中启用clip mouse模式。使用simple lighting可加快clip plane的运动速度。
Clip可从下方到E56查看通道。
按住Alt键可clip远侧以显示通道上方释放质子。
positioning clip plane后,为较暗的通道腔打开full lighting。
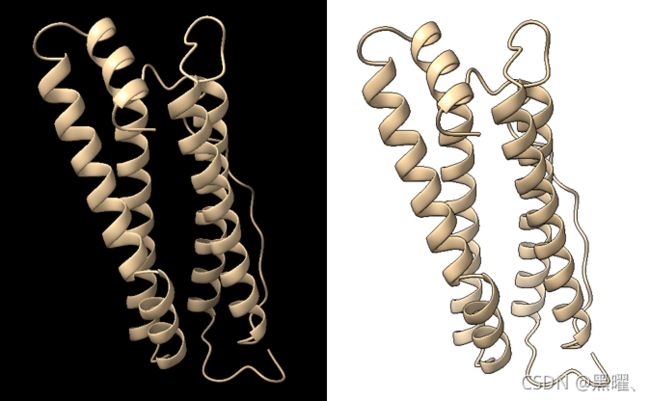
Look at unidirectional rotation
eLife论文的图3提出了一种机制,说明了为什么motor朝一个方向旋转能产生ATP而反向阻塞。
关闭剪辑命令:“clip off”
定向结构轴垂直于屏幕并且F1六聚体在前面。
使motor 轴epsilon亚基(链H)更明显:“color /H red surface”
clip mouse模式,剪切。
epsilon亚基(红色)与六聚体的beta亚基(灰色)碰撞,阻止顺时针旋转。
一些命令:
标记:
label #1/A:157
预设:
preset inter #不显示轮廓,默认背景(黑)
preset pub #显示轮廓,白背景
上色:color spec colorname [target string] [transparency percent]
color /d & helix sky blue cartoons #给D链的cartoon形式的螺旋蓝色
color bychain #按链上色
color byatom/byelement #按每个原子/元素上色
color byhet #按非碳原子上色
color bypolymer #按多聚体上色(给每个单聚体上色)
color bymodel #按模型上色
rainbow #每个螺旋颜色不同
rainbow chains #按链上色
rainbow residues #按残基上色
rainbow polymers #按单聚体上色
rainbow structures #按模型结构上色