机器学习--聚类分析
聚类分析
聚类是一种无监督的分类方法。我们可以对变量聚类或者样本聚类,从而达到将相似性大的样本或变量分到一类,组内区分度较小,组间区分度大的目的。
聚类的方法,也会根据聚类的目的分为若干种,一种是基于变量的聚类,比如层次聚类,另一种是基于样本的聚类,比如k-means,还有基于密度的聚类(dbscan)
这里主要分享最近学习和因为一些需求新写的代码部分----k-means和层次聚类。
- 层次聚类
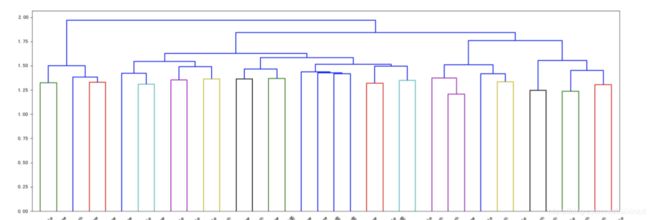
可以将聚类的结果用谱系图形象的展示出来,通过谱系图,可以清晰的看到分类的层级(以及遇到的几个需求是,根据不同的方差解释度,将谱系图的聚类输出)
层次聚类 在r中使用hclust进行,最近在写python 这里分享python和sas的代码实现。
####层次聚类
###聚类
import scipy.cluster.hierarchy as shc
from scipy.cluster.hierarchy import dendrogram, linkage
from sklearn.cluster import AgglomerativeClustering
import matplotlib.pyplot as plt
pd_data=corr.toPandas()
variable_list=corr.columns
variable_list.remove('id')
variable_list
pd_datatt=pd_data[variable_list]
#####linkage函数进行聚类 可以选择不同的参数
linked = linkage(pd_datatt, 'ward')
labelList = variable_list
plt.figure(figsize=(50, 30))
plt.xticks(size = 1000)
dendrogram(linked,
orientation='top',
labels=labelList,
distance_sort='descending',
leaf_font_size=10.,
show_leaf_counts=True)
plt.rcParams['font.sans-serif']=['SimHei']
# plt.show()
plt.savefig('cluster_var.png',dpi=80)
####这里遇到的一个需求是,要直接输出谱系图的结果,相当于指定要分几类后,按照谱系图输出类别
###这个需求可以直接用n_clusters做到
cluster = AgglomerativeClustering(n_clusters=10, affinity='euclidean', linkage='ward')
label_clus = pd.DataFrame({'label':variable_list,'clust':cluster.fit_predict(pd_data.drop('id',axis=1))})
但是,随后又遇到了一个新的需求,需要通过在谱系图上设置一个划分距离的百分比(相当于上图中,比如要在1.75处进行划分,就相当于要设置一个比例 1.75/2)
####直接在pyspark里写了
distance_table = pd.DataFrame(linked)
distance_table.columns = ['cluster1','cluster2','distance','cnt']
input_percent=0.8
distance_table= spark.createDataFrame(distance_table)\
.withColumn('maxdist',max('distance').over(Window.orderBy(lit(1)))).orderBy('distance')\
.withColumn('dist_per',col('distance')/col('maxdist'))
distance_value=distance_table\
.where(col('dist_per')
解决了上一个问题之后,又遇到了一个新的问题,在sas中得到的层次聚类结果和python略有不同(应该是某些方法不一样),然后就需要在sas中也做一步,按照解释方差的比例进行划分,但没有找到sas的函数,观察sas得到的结果,其实很像决策树找节点。
原理比较简单,所要做的,是从树图最底下的标签开始,不断寻找上级父节点,以及对应的已解释方差比例,然后得到一张全表后,筛选大于等于临界值部分。这个时候,最小的解释方差比例对应的父节点,其实已经把标签分成若干类了(这个很像之前分享过的决策树寻找父节点,之前写过r版本,以下分享python版本)
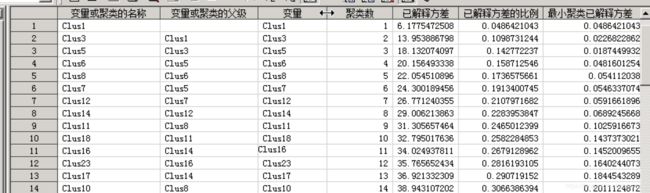
###导入上图的源数据
data_input = pd.read_excel('tree_result_raw.xlsx', dtype = 'str')
data_input['label']='result1'
label_data=data_input[data_input['label']=='result1']
parent=label_data[label_data['_NAME_'].str.contains("Clus")]
child=label_data[label_data['_NAME_'].str.contains("Clus")==False]
rownumber=child.count()['label']
output = pd.DataFrame()
###寻找自节点对应的父节点 合并到数据集中
def getparent(child_input,father_input):
global data
data = pd.DataFrame()
result1=parent[parent['_NAME_']==father_input][['_PARENT_','_PROPOR_','label']]
result1['child']=child_input
result1['current']=father_input
global parent_loop
parent_loop=result1.iloc[0,0]
data=pd.concat( [data,result1], axis=0)
return data
###开始循环 直到找到的父节点是clus1 即最顶层
for i in range(0,rownumber):
child_loop=child.iloc[i,0]
parent_loop=child.iloc[i,1]
while parent_loop !='Clus1':
getparent(child_input=child_loop,father_input=parent_loop)
output=pd.concat( [output,data], axis=0)
print(i)
###把所有自节点合并到找到的父集中
child0=child[['_PARENT_','_PROPOR_','label','_NAME_']]
child0['current']=''
child0.columns=['_PARENT_','_PROPOR_','label','child','current']
child0
output0=pd.concat([child0,output],axis=0)
###根据需要的临界值 输出此时对应的组别
var_cut=0
def group_cut(var_cut):
output_cut= spark.createDataFrame(output0)\
.withColumn('_PROPOR_',col('_PROPOR_').cast('float'))\
.where(col('_PROPOR_')>var_cut)\
.withColumn('min',min('_PROPOR_').over(Window.partitionBy('label','child').orderBy('label')))\
.where(col('_PROPOR_')==col('min')).orderBy('_PARENT_')
global group_need
group_need=output_cut.groupBy('label').agg(countDistinct('_PARENT_')).collect()[0][1]
return group_need,var_cut,output_cut
###循环遍历
var_cut=0
group_need=0
while group_need<10:
group_cut(var_cut)
print(group_need,var_cut)
var_cut=var_cut+0.02
P.S. 顺便分享一下sas的层次聚类的代码:
proc contents data=rawdata2 out=cont2 noprint;run;
proc sql noprint;
select kcompress("'"||name||"'n ") into : name1 separated by ' '
from cont2
where name not in ('id');
quit;
proc varclus data=rawdata2 outstat=clus centroid short PLOTS(MAXPOINTS=400)
outtree=clust_tree;
var &name1.;
run;
- k-means
k-means主要用在已知分类数后(比如通过层次聚类,已经确定组数),直接对样本点进行划分,python代码如下:
###聚类
#根据correlation 用kmeans 对变量聚类
from pyspark.ml.feature import VectorAssembler,StringIndexer,VectorIndexer,IndexToString
from pyspark.ml.clustering import BisectingKMeans
inputcol=corr.columns
inputcol.remove('id')
inputcol
# # #矢量化
vecAssembler=VectorAssembler(inputCols=inputcol,outputCol="features")
df_Kmeans=vecAssembler.transform(corr).select('id','features')
df_Kmeans.write.mode('overwrite').parquet(path)
df_Kmeans=spark.read.parquet(path)
# # # #导入已有模型
kmeans=BisectingKMeans().setK(15).setSeed(78667).setFeaturesCol("features").setMinDivisibleClusterSize(5)
model=kmeans.fit(df_Kmeans)
model.write().overwrite().save(path)
model=pyspark.ml.clustering.BisectingKMeansModel.load(path)
transformed=model.transform(df_Kmeans).select('id','prediction')
transformed.write.mode('overwrite').parquet(path)