[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review
[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review
- 0. Abstract
- 1. Introduction
- 2. Fusion strategies: an overview
- 3. Early fusion
-
- 3.1 Direct modeling
- 3.2 Latent representation with multimodal AEs
-
- 3.2.1 Autoencoders
- 3.2.2 Variational AEs 变分AEs
- 3.3 Discussion of early fusion strategies
- 4. Intermediate fusion
-
- 4.1 Homogeneous network design 同构网络设计
-
- 4.1.1 Marginal homogeneous fusion 边缘同质融合
- 4.1.2 Joint homogeneous fusion 联合同质融合
- 4.2 Heterogeneous network design 异构网络设计
-
- 4.2.1 Marginal heterogeneous fusion
- 4.2.2 Joint heterogeneous fusion
- 4.3 Discussion on intermediate fusion
- 5. Late fusion
-
- 5.1 Discussion of late fusion
- 6. Discussion and conclusion
这个系列会解读一些列医学多模态融合的文章,了解近在医学领域这两年(2020之后)最新的多模态融合方法.
paper4: Multimodal deep learning for biomedical data fusion: a review
国内打不开链接,需要原文的链接加v发:liyihao76
0. Abstract
生物医学数据正变得越来越多模式,从而捕捉生物过程之间潜在的复杂关系。基于深度学习 (DL) 的数据融合策略是对这些非线性关系进行建模的一种流行方法。因此,我们回顾了此类方法的最新(state-of-the-art)技术水平,并提出了详细的分类法(taxonomy),以促进更明智地选择生物医学应用的融合策略,以及对新方法的研究。通过这样做,我们发现深度融合策略通常优于单峰(unimodal)和浅层(shallow)方法。此外,所提出的融合策略的子类别显示出不同的优点和缺点。对当前方法的回顾表明,特别是对于中间融合策略,联合表示(joint representation)学习是首选方法,因为它有效地模拟了不同层次生物组织的复杂相互作用。最后,我们注意到,基于先前生物知识或搜索策略的渐进式融合是一条有前途的未来研究道路。同样,利用转移学习可能会克服多模态数据集的样本量限制。随着这些数据集越来越多,多模态DL方法提供了训练整体模型的机会,这些模型可以学习健康和疾病背后的复杂调节动态。
- Keywords: fusion strategies, data integration, deep neural networks, multimodal machine learning, representation learning, multi-omics(多组学)
1. Introduction
单个细胞和完整的生物体是典型的复杂系统,因为它们由许多不同的部分组成,这些部分相互作用并产生紧急行为 [ 1 ]。在尝试对复杂疾病进行预测时,了解这些相互作用尤为重要。数据模态是使用特定传感器 [ 2 ] 测量此类现象的结果,因此它本身提供的信息有限。使用多模态数据,可以获得有关各个部分及其紧急行为的信息。由于高通量技术的快速发展,我们现在可以前所未有地访问大规模多模态生物医学数据,从而有机会利用这些更丰富的信息。
数据融合是来自不同模式的数据的组合,这些模式提供对常见现象的不同观点以解决推理问题。与单峰方法相比,这有望以更少的错误解决此类问题 [ 3 ]。更具体地说,数据融合的优势可以分为互补、冗余和协作特征 [ 4、5 ] ,尽管这些特征并不相互排斥。
数据融合在生物医学领域的优势可以通过对癌症患者的多模态研究来说明。来自肿瘤的基因组数据能够识别癌症驱动基因,而来自活组织检查的全幻灯片图像 (WSI) 提供了肿瘤形态和微环境的视图。这些模式是“互补的”,因为它们提供了有关现象的不同部分的信息,否则无法观察到。转录组学和蛋白质组学数据的融合是互补的,因为所有的 mRNA 都没有翻译成蛋白质,并且是“冗余的”,因为蛋白质的丰度证实了特定 mRNA 到蛋白质的翻译。当数据嘈杂或有许多缺失值时,这种冗余尤为重要。来自同一肿瘤的 miRNA 和 mRNA 测序的数据可以被认为是“合作的”,因为组合信息增加了复杂性。两种模式的融合为例如致癌基因的蛋白质的差异丰度提供了可能的解释。这可能在预测患者对某种治疗的反应方面发挥重要作用。
融合策略的目的是有效地利用不同模态的互补、冗余和协作特征。为了充分利用这些对感兴趣现象的看法,必须部署机器学习 (ML) 方法,这些方法能够融合具有不同统计特性、非生物变异来源、高维性的结构化和非结构化数据 [ 6 ]以及不同模式的缺失值 [ 2 ]。
近年来,多模态 ML 方法在各个领域得到越来越多的研究和应用 [ 6 , 11 ]。图 1说明了生物医学领域的这种趋势。多模态深度学习 (DL) 尤其具有优于浅层数据融合方法的优势。全连接神经网络 (FCNN) 是深度神经网络 (DNN) 的传统形式,可以看作是有向无环图,它映射输入X到label Y是通过几个隐藏层的非线性计算操作[ 12 ]。表 1总结了常见的 DL 架构。此类算法的目标是学习输入数据的高级表示,通过找到潜在的分离因素之间的简单依赖关系来改进最终分类器的预测。较早的层学习数据的简单抽象,而较深的层将这些组合成更抽象的表示形式,这些表示形式为学习任务提供信息 [ 13 ]。至关重要的是,多模态 DL 能够模拟非线性模态内和模态间关系。这导致其在各种领域的应用[ 2]. 然而,生物医学应用面临多模态融合的特定挑战,例如与组合维度相比样本量较小、整个模态缺失以及模态之间的维度不平衡。
![[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review_第1张图片](http://img.e-com-net.com/image/info8/3e7b6df3e4674a6aab65f65b4f22f6e5.jpg)
| Architecture | Description |
|---|---|
| Fully connected neural networks | FCNNs是最传统的深度神经网络(DNNs)。在一个层中,每个神经元都与后续层的所有神经元相连[12]。 |
| Convolutional neural networks | CNN能够对空间结构进行建模,如图像或DNA序列。每个神经元都与后续层的所有神经元相连。在卷积层中,卷积核在输入数据上滑动以模拟局部信息[12]。 |
| Recurrent neural networks | RNNs通过维护一个编码先前时间步骤信息的状态向量,对顺序数据进行良好的建模。这个状态由网络的隐藏单元表示,并在每个时间步骤中更新[12]。 |
| Graph neural networks | GNNs是由实体和它们的连接组成的图形模型,代表了例如组织的分子或细胞核。GNNs的层可以采取不同的形式,如卷积和递归[14]。 |
| Autoencoders | AE通过首先压缩输入数据,然后重建原始输入数据来学习低维的编码。层可以是不同的类型,如全连接或卷积[15]。 |
尽管已经审查了用于生物医学应用程序的 DL 架构 [ 16 ],但针对异构数据的不同的基于 DL 的融合策略尚未得到审查。这在本综述中得到解决,我们在其中描述了生物医学领域中基于 DL 的融合策略的最新技术水平。此外,我们提出了一个分类法,它不仅概述了早期、中期和晚期融合的标准分类,而且还描述了对希望应用或增强当前方法的研究人员和从业者有用的子类别。此外,本综述的目的是为不同融合策略在哪些条件下最有可能表现良好提供指导。
为此,首先概述了主要的融合策略,并提出了更详细的分类法。接下来,详细描述早期、中期和晚期融合类别及其子类别,并广泛举例说明其在生物医学问题中的应用。最后,我们讨论了所描述的策略在生物医学领域的挑战和机遇,并为未来的研究提出了建议。
2. Fusion strategies: an overview
DNN 学习输入数据的层次表示的能力使它们特别适合应用于多模态学习问题。如何以一种能够有效组合异质模态的方式找到边缘(marginal)和联合表示(joint representations)的挑战是多模态融合的核心 [ 11 ]。因此,我们在提出详细的分类法时采用了表征学习的观点(见表 2)。
| Fusion strategy | Taxonomy Subcategory 1 分类法子类1 | Taxonomy Subcategory 2 | Papers |
|---|---|---|---|
| Early fusion | Approach | Architecture | |
| Direct modeling | Fully connected | [17–19] | |
| Convolutional | [20–23] | ||
| Recurrent | [20, 24] | ||
| Autoencoder | Regular | [25–34] | |
| Denoising | [33, 35–37] | ||
| Stacked | [37–40] | ||
| Variational 变分的 | [33, 40–42] | ||
| Intermediate fusion | Branch | Representation | |
| Homogeneous design同质化设计 | Marginal | [43–49] | |
| Joint | [21, 28, 38, 41, 50–63] | ||
| Heterogeneous designs 异构设计 | Marginal | [64–68] | |
| Joint | [69–81] | ||
| Late fusion | Aggregation聚合 | Model contribution | |
| Averaging | Equal | [82–84] | |
| Weighted | [82–84] | ||
| Meta-learning | Weighted | [83, 88] |
“边际表示(Marginal representation)”被定义为单峰(unimodal)输入数据转换的结果,理想情况下是以发现潜在有用因素的方式。“联合表示(joint representation)”由代表基于多种模态(multiple modalities)的潜在因素的特征组成,因此编码信息可能是互补的、冗余的或合作的。Baltrušaitis等人[ 11 ] 还描述了“协调表示”,其中多模式数据未投影到公共空间中。学习到的边缘表示受到其他模态表示的约束,例如相似性约束。
在很大程度上,融合策略可以根据融合层的输入状态分为早期、中期和晚期融合 [ 2 ](图 2中的蓝色层)。
![[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review_第2张图片](http://img.e-com-net.com/image/info8/d6a1ba0beedd4727b123b526f25590e6.jpg)
在“早期融合”中,原始输入数据被连接起来,生成的向量被视为单峰输入(unimodal input),这意味着 DL 架构不会区分来自哪个模态特征(参见图 2a )。 直接学习多模态输入的联合表示,没有明确学习边际表示。我们进一步区分了基于输入数据“直接建模”的早期融合,通过 DNN 等同于它们的单峰对应物,以及首先学习低维联合表示的“自动编码器”(AE) 方法,后者又用于进一步建模与监督或无监督方法。
早期融合的优势在于其简单性,因为无需做出关于如何提取边缘表示的设计选择。尽管它很简单,但早期融合策略可以从低级特征中学习跨模态关系。然而,这种方法可能无法识别模态之间的关系,因为它们只在更高的抽象层次上变得明显,因为没有明确学习边缘表示。此外,早期融合策略对模态的不同采样率很敏感 [ 2 ]。
在“中间融合”中,学习并融合了特征向量形式的边缘表示,而不是原始多模态数据(参见图 2b)。这种边缘表示可以通过相同类型的神经网络(完全连接、卷积神经网络等)学习,因此我们将其称为“同构”设计网络。或者,边缘表示是通过不同类型的网络学习的,因此称为“异构”设计。正如命名所暗示的那样,前者在模态同质(homogeneous)时更常见,而后者更好地处理多模态数据的异质性(heterogeneity)。
我们进一步区分 "边际 "中间融合和 "联合 "中间融合,前者是将边际表征串联起来并直接输入到分类器中,后者是学习更抽象的联合特征。边缘中间融合有时也被称为特征后期融合或后期融合。我们把这些方法归类为中间融合,因为融合层的输入是特征,而后期融合被定义为子模型的决策融合。然而,需要注意的是,文献中使用了不同的术语。在联合中间融合中,可以发现进一步的多模态分解因素,从而提高最终分类器的性能。在这种情况下,渐进式融合成为一种有趣的可能性,其中高度相关的模态被提前融合,而其他模态则在架构的后期被融合[2]。
中间融合策略的优势在于它们可以灵活地找到融合边缘表示的正确深度和顺序。这可以说更密切地反映了模式之间的真实关系。因此,可能会发现更有用的联合和边际潜在因素。DL 架构特别适用于中间融合,因为它们很容易通过将边缘表示连接到共享层来融合边缘表示,并将层次表示与自然世界对应起来。
在“后期融合”中,不是合并原始数据或学习到的特征,而是将单独的单峰子模型的决策合并为最终决策 [ 2 , 11 ](参见图 2c)。这允许学习良好的边缘表示,因为每个模型都可以适应特定的模态。此外,子模型的误差可能不相关,因此具有互补效应 [ 2]. 但是,最终模型无法学习对数据或特征级别的多模态影响。我们根据子模型决策的聚合方式进一步区分后期融合策略。这些预测可以以相等或加权的方式“平均”。或者,在 ML 模型接收预测概率作为输入并学习做出最终预测的情况下执行“元学习”。
3. Early fusion
3.1 Direct modeling
在某种程度上,DL的成功可以归功于从大数据集中学习得很好,即使特征的数量很高[89]。然而,生物医学领域的数据集往往具有较小的样本量,特别是与它们的维度相比。尽管如此,早期融合的一种方法是将不同模式的输入特征串联起来,形式上是 x c o n c a t = x 1 ∣ ∣ x 2 ∣ ∣ . . . ∣ ∣ x m x_{concat}=x_1||x_2||...||x_m xconcat=x1∣∣x2∣∣...∣∣xm,其中x_i是一种模式的输入向量(见图3a)。由此产生的连接向量 x c o n c a t x_{concat} xconcat被输入到DL架构的第一层。该神经网络不区分来自不同模态的特征。在这种方法中,跨模态和模态内的相关性是在一个低水平的抽象中同时学习的。
如果特征的排序与学习任务无关,矢量 x c o n c a t x_{concat} xconcat可以用全连接输入层来建模,如文献[17, 18]和文献[19]中的约束。如果输入特征的排序包含结构信息,如基因组数据或临床数据的时间序列的情况下,递归层[20, 24]或卷积层[20, 21, 23]可以应用于串联的向量。在这种情况下,顺序信息也可以作为每个样本的矩阵来堆叠,而不是一个串联的一维矢量。例如,矩阵中的每一列可以代表基因组中的一个位置,行代表模式(见图3a)。在卷积层的情况下,内核可以在矩阵上滑动以提取相关特征。在递归层的情况下,每一列可以被看作是一次性的步骤。
3.2 Latent representation with multimodal AEs
3.2.1 Autoencoders
另一种常用的从xconcat中学习的方法 的另一种常用方法是找到一个较低维度的联合潜在表示,其中包含重建原始输入的必要信息。AE是一种架构,能够通过编码器函数f(x)和解码器函数g(z)以无监督的方式从输入x中学习这种嵌入z(见图3b)[15]。这很有用,因为输入x的一些潜在因素 也解释了条件概率p(y|x) [13]。AE的目的是最小化重建损失函数。
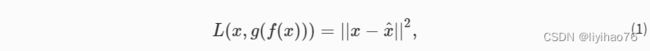
其中x^ 是重构的输入。通过最小化重建损失,AE的目标是接近原始输入特征。如果f(x)和g(z)是线性函数,那么z位于主成分子空间中,使得AE类似于主成分分析。然而,如果编码器和解码器是非线性的,并且数据中存在非线性,它们可以将输入特征映射到一个比原则分量更有信息量的低维空间中的流形。为了提取这个低维流形,有必要通过设置构成z的神经元数量低于x的二维性来约束架构,也被称为AE的不完全性[12]。重要的是,嵌入空间中的单一潜在因素可能在一个以上的模式中变得可见,因此证明了使用多模式AE的合理性,以 x c o n c a t x_{concat} xconcat 作为输入。
尽管 AE 并非早期融合策略所独有,但它们经常在生物医学文献中用于学习联合表示(joint representations)。学习后,联合表示z可用于进一步建模。例如,在癌症患者生存子类型分析中,通常在通过 AE 进行联合表示学习之后是使用单变量 Cox 比例风险 [ 90 ] 建模进行特征选择的步骤。然后使用选定的潜在特征通过无监督方法为每个患者推断与其风险子类型相对应的标签。最终在这些标签上训练了一个监督模型,以便稍后预测看不见的患者的数据。特别是在使用多组学模式的癌症患者生存分型中,这种步骤顺序变得很流行 [25–31, 34].
对于同样的临床任务,研究人员已经调整了类似的工作流程,但应用去噪AE(DAE)[91]来代替[35, 36]。通过向输入x添加噪声 而不是重建损失中的x(等式1),DAE必须学习重建,同时去除噪声以近似未被破坏的向量x。这使得AE具有过度完整性,并且具有大量参数的编码器和解码器。对于AE来说,超完备性可能是可取的,因为它具有对噪声的鲁棒性等特性。
同样,不同形式的 AE 已被用于早期融合生物医学数据。伊斯兰教等[ 38 ] 和 Rakshit等人[ 39 ] 使用堆叠 AE (SAE) 来融合多组学数据,以对乳腺癌的分子亚型进行分类。在 SAE 中,几个 AE 被堆叠并顺序训练以重建前面编码器的输出。然后可以针对分类任务对该架构进行微调。在 [ 38 ] 的情况下,所提出的方法执行类似于中间融合方法。米奥托等人[ 37] 将堆叠 DAE 应用于多模态电子健康记录 (EHR) 数据,有效地代表低维空间中的患者,从而实现多种临床预测建模,例如疾病的发作。
正如 Jaroszewicz等人所展示的,早期 AE 融合也可用于初始化另一个神经网络的第一层。[ 32 ] 关于染色质峰的精细定位。使用有用的数据联合潜在表示进行初始化可以显着增强训练过程。可以进一步调整联合表示层,从而能够学习更多与任务相关的联合表示。
3.2.2 Variational AEs 变分AEs
如前所述,假设高维数据x位于一个低维流形上。这个假设可以表示为一个有向概率模型,其中数据点x是由低维变量z的随机过程产生的(见图3c)。假设z是由高斯分布 p θ ∗ ( z ) p_{θ^∗}(z) pθ∗(z)产生的,其中 θ ∗ θ^∗ θ∗是真正的生成参数。因此, p θ ∗ ( z ) p θ ∗ ( x ∣ z ) p_{θ^∗}(z)p_{θ^∗}(x|z) pθ∗(z)pθ∗(x∣z)是看到数据x的可能性。尽管z是感兴趣的现象的更直接的表示,但在嵌入空间中直接表示数据是很有用的。然而,z和 θ ∗ θ^∗ θ∗是不是直接可观察的,在大多数情况下估计真实的后验 p θ ∗ ( z ∣ x ) p_{θ^∗}(z|x) pθ∗(z∣x)是难以做到的。
变量AE(VAE)[92]能够通过学习所谓的识别模型 q ϕ ( z ∣ x ) q_ϕ(z|x) qϕ(z∣x)来近似真实的后验,反过来可以从中学习 p θ ( x ∣ z ) p_θ(x|z) pθ(x∣z)。在这个例子中,解码器从 N ( μ ( i ) , σ ( i ) I ) N(μ(i),σ(i)I) N(μ(i),σ(i)I)中取样 z ( i ) z(i) z(i),其中I是身份矩阵,并学习 p θ ( x ( i ) ∣ z ( i ) ) p_θ(x^{(i)}|z^{(i)}) pθ(x(i)∣z(i))来重建输入 x ( i ) x^{(i)} x(i)。这种方法的一个预期优势是,由于它的生成性,潜在空间z更加平滑,从而对未见过的数据有更好的概括性。此外,对潜在空间分布的先验信念的引入使得对输入数据的建模更加灵活。这些优点在从多模态数据中学习联合潜势表征时也很有用,因为在多模态情况下可以假设所描述的相同过程。
Simidjievski等人[ 41 ] 系统地研究了用于融合乳腺癌数据的 VAE。在比较不同的 VAE 融合策略时,早期融合 VAE 的性能与更复杂的 VAE 架构相当。此外,作者发现正则化方法及其权重的选择对模型的性能有很大影响。罗南等人[ 40 ] 通过应用基于多组学数据的堆叠 VAE,对结直肠癌进行了生存亚型分类,并将细胞系与亚组相匹配。阿尔巴拉迪等人[ 42] 用卷积层替换 VAE 的完全连接层,以学习输入到分类器的嵌入以进行泛癌转移预测。因此,他们表明可以利用多组学数据中的局部模式。
3.3 Discussion of early fusion strategies
大多数早期融合模型与它们的单峰版本没有太大区别。它们实施起来相对简单,因为无需对个体模态进行建模,这也许可以解释它们在生物医学文献中的受欢迎程度。这里回顾的早期非线性融合方法的应用表明,这些方法在预测任务上可以优于浅层方法(例如 [ 35、36 ] )。这表明 DL 方法是传统方法的可行替代方法,即使样本量相对较小,因为在上面审查的应用程序中只有 96 名患者 [ 28 ]。此外,早期融合策略往往优于单峰方法(例如 [ 39]). 然而,不同的模态可以在不同程度上添加信息(例如 [ 36 , 44 ])。
尽管它们有突出的用途,但早期融合策略也有缺点。通过直接对联合表示进行建模,很难找到每种模态的有用边缘表示。模态的相关特征可能只会在更高的抽象层次上变得明显。在联合表示中发现此类特征可能更难实现。此外,模态之间可以有不同的关系。因此,逐渐融合模态而不是全部融合在一层中可能是有益的 [ 2 ]。最后,早期融合往往仅在模态相当同质时应用,例如不同的“组学(omics)”模态。如果模态显示出截然不同的分布,例如图像和分子模态,则早期融合策略不太可能表现良好。
在早期的融合策略中,基于AE的融合在生物医学应用中被频繁使用(见表2)。这些方法的降维能力可以解释它们主要用于高维多组学数据。这些方法的一个局限性是不针对任务的学习。虽然学习 x c o n c a t x_{concat} xconcat的基本因素对预测反应y很有用[13],但AE方法是为了重建输入数据而学习,不一定是为了提取目标的相关因素。因此,学习到的联合潜在表征不能保证对应用的最终目的是最佳的,如果存在标签,进一步的特定目标学习可能是有益的。
佛朗哥等人[ 33 ] 将几种 AE 类型与多组学数据进行了癌症生存亚型早期融合的比较。尽管常规 AE 和 VAE 架构似乎优于其他 AE,但不同数据集上性能之间的巨大差异表明架构选择的重要性。尽管存在上述缺点,一些评论的论文表明,早期融合 AE 可以与中间策略 [38、41] 相提并论,尽管生物医学领域 [ 28 ] 和 [ 93 ] 之外的其他结构化研究表明中间融合优于早期融合策略。
4. Intermediate fusion
尽管早期融合不知道特征源自什么模态,但中间融合策略利用了这种先验知识。学习每种模态的边缘表示以发现模态内的相关性,然后再使用它们来学习联合表示或直接进行预测(参见图 4a)。在下文中,我们将讨论同质和异质中间融合及其子类别。
4.1 Homogeneous network design 同构网络设计
4.1.1 Marginal homogeneous fusion 边缘同质融合
通过连接这些边缘表示,可以将具有相同类型层的分支学习到的边缘特征直接用作决策函数的输入。虽然这种方法能够有效地捕获模态内的相关性,但跨模态关系的建模效率较低,从而降低了数据融合的好处。但是,模型的复杂性降低了,从而降低了过度拟合的风险。因此,如果模式在很大程度上独立地影响结果,则选择仅学习边际表征可能是有益的。这强调了多模式数据的互补性和冗余性,而不是合作方面。
为了预测癌症患者的存活率,Huang等人[ 43 ] 通过两个局部完全连接的分支融合 mRNA 和 miRNA 特征基因矩阵。然后将边缘表示和额外的临床和人口统计数据输入到 Cox 比例风险回归模型中。没有进行联合学习,因为作者明确假设不同的模式独立影响危险函数。
为了将模态与顺序数据融合,每个分支中的循环层都提供了良好的性能,因为可以有效地建模时间依赖性并且输入序列可以是可变长度的,这在生物医学数据中通常是这种情况。循环层能够输出编码输入序列的边缘表示。李等人[ 44 ] 将门控循环单元 (GRU) 网络应用于阿尔茨海默病 (AD) 患者的多模式数据。由 GRU 层组成的每个分支首先分别在分类任务上进行训练。在第二步中,每个分支的边际表示被连接起来,逻辑回归用于做出最终决定。
除了作为早期融合策略受到欢迎外,AE还可以在中间融合中找到应用。独立的AE可以单模态应用,产生一组编码S={z1, z2,…, zm},其中m是模态的数量,zi是由相应AE编码的第i个模态的潜在表示。S中的编码可以串联成向量 z c o n c a t = z 1 ∥ z 2 ∥ . . . ∥ z m z_{concat}=z1∥z2∥...∥zm zconcat=z1∥z2∥...∥zm(见图4b),并作为进一步建模的输入,如聚类后用分类器进行癌症分型[45-47],或直接作为多类分类或生存分析的分类器的输入[48]。原则上,zconcat可以输入到DNN,DNN学习联合表示,使其成为联合融合方法。
4.1.2 Joint homogeneous fusion 联合同质融合
在连接边缘表示之后,可以通过单峰分支之后的多个层来学习联合表示。这种联合表示随后可用于做出决策,并可对跨模态交互进行建模(参见图 4a)。
Sharifi-Noghabi等人[ 50 ] 将单独的完全连接的分支应用于多组学数据,然后是用于药物反应预测的多层分类网络。因此,该分类器学习了输入模态的联合表示。林等人[ 51 ]采用这种方法预测乳腺癌亚型。
为了保留空间信息,如果可以预期模态内的这种依赖性,则可以在每个分支中应用卷积层。与单峰模型类似,可以在每个分支中使用附加层(例如最大池化层)来降低维度并避免过度拟合。至关重要的是,每个分支的特征图可以连接到单独的密集层,然后连接起来。从这个向量中,可以在后续层中学习联合表示。此类架构可应用于多种模式,例如药物的化学结构和基因组数据 [ 21 ] 或多组学模式 [ 38 ]。
另外,单模态分支可以由深度相信网络(DBNs)组成。在多模态DBN中,每一对相邻的层都是受限的博尔兹曼机(RBM),它们被训练为以无监督的方式对两层的联合分布p(xl,xl+1)建模。与SAE类似,在逐层训练过程中,嵌入或隐藏的表示成为后续RBM的可见输入。因此,DBN可以被认为是一个堆叠的RBM。输入数据的分层表示被学习,可用于数据的聚类。另外,这些表征可以作为DNN的一个有用的、计算效率高的初始化,用更昂贵的监督算法(如反向传播)对其进行微调,以学习p(y|x)[94]。
DBN 已广泛用于融合生物医学模式,用于药物再利用 [ 52 ]、癌症患者聚类 [ 53 ] 和预测疾病基因对 [ 54 ]。苏克等人[ 55 ] 应用多模态深度玻尔兹曼机 (DBM) [ 95 ] 从磁共振图像 (MRI) 和正电子发射断层扫描 (PET) 扫描预测 AD。与 DBN 类似,DBM 由堆叠的 RBM 组成,但除了自下而上的学习步骤外,它们还添加了自上而下的反馈,从而能够学习更好的表示。
为了利用模态特定和跨模态相关性,可以在单个 AE 中学习边缘和联合表示(参见图 4c)。最初,AE 由连接到不同模态的分支组成。进一步进入编码器,通过将每个分支连接到z的所有神经元,在这些分支中学习的边缘表示被融合到嵌入层中,如 [ 28 ] 中所做的那样。或者,它们可以融合在一个隐藏层中,从而在最终编码之前实现潜在的进一步学习 [ 57-61 ]。然后嵌入z可用于不同的预测任务。
这种联合表示也可以通过 VAE 学习。Simidjievski等人[ 41 ] 提出并比较了使用 VAE 融合乳腺癌多组学和临床数据的联合 AE 融合的不同版本。希拉等人。[ 56 ] 还发现联合多模态 VAE 对于融合多组学数据很有用,并支持 [ 41 ] 的发现,即最大平均差异作为正则化项优于 Kullback-Leibler 散度。
与 VAE 相关,Lee 和 van der Schaar [ 63 ] 通过应用信息瓶颈原理融合了多组学数据。尽管 VAE 可以有效地找到输入的潜在表示,但它们可能不是预测任务的最佳选择。变分信息瓶颈方法 [ 96 ] 找到一种联合表示,该表示保留来自输入x的与预测目标y相关的信息,同时最大限度地压缩x 。目标函数鼓励算法找到有用的边缘和联合表示。同样,张等人。[ 62] 提出了一种端到端的 VAE 架构,该架构学习用于泛癌分类的 DNA 甲基化和基因表达数据的特定任务联合表示。该架构始终优于 VAE 和支持向量机的组合。
4.2 Heterogeneous network design 异构网络设计
4.2.1 Marginal heterogeneous fusion
能够对具有不同分支的模态进行建模的主要优点是能够将异构数据转换为更好地表示更高级别特征的向量。因此,这些新的特征向量可以在数据类型、不同模态之间的维度和规模不平衡方面“公平竞争”,从而实现比较。与同质中间融合策略一样,这种边缘表示可以简单地连接起来并输入到分类器中。
徐等人[ 68 ] 将计算机断层扫描 (CT) 扫描的实验室测试、临床数据和边缘表示相结合,以预测 COVID-19 感染。张等人[ 64 ] 提出了一种基于 CNN 和 RNN 的融合模型,该模型将时间信号、顺序临床记录以及静态人口统计和入院数据作为不同分支的输入。它将前两种模式嵌入到潜在特征空间中,并将它们与编码的静态信息连接起来。因此,创建了输入到分类器的患者表示。特征选择也可以用于连接的边缘表示,选择对目标变量影响最大的潜在特征,如 [ 65 ] 中所做的那样] 预测透明细胞肾细胞癌患者的预后。郝等人[ 67 ] 缺乏额外的隐藏层与临床数据的低维度,与高维基因组数据融合。然而,作者假设如果有更多的临床特征可用,可能需要额外的关节隐藏层。
通常,在边缘异质融合中,通常对于模态的一个子集,找到边缘表示,然后将其与其他模态的原始数据连接起来。在这些情况下,非编码模态是低维的,不会遭受维数灾难。因此,它们可能不需要通过分离的潜在因素来表示。
4.2.2 Joint heterogeneous fusion
通常,可以合理地假设不同的模态不会独立影响目标变量,而是存在提供信息的跨模态相互作用。在联合异构中间融合中,这种关系是通过从边缘表示中学习特征的交互来建模的。这些交互可以通过首先连接边缘表示并将该向量馈送到特定任务输出层之前的完全连接层来学习。例如,MRI 和临床数据可以融合用于 AD 预测 [ 72 ],或者 MRI、临床和基因组模式可以融合用于 AD 分期检测 [ 69 ]. 此外,可以融合多种成像模式和临床数据的潜在表示,以评估肝细胞癌肝移植的风险 [ 71 ]。
基于这种联合异构中间融合的通用方法,其他研究人员增加了架构改进以应对特定挑战。在实践中,并非为每个患者收集所有模态通常是一个问题。如果缺少整个模式,插补可能会变得具有挑战性,并且仅对完整样本进行训练会限制训练集的大小。通等人[ 70] 提出了一个多任务网络,它可以通过具有单峰输入分支和特定于任务的输出分支来有效地从具有缺失模态的多峰数据中学习。每个任务都反映了一种模态或模态组合的可用性。因此,在训练期间仅更新特定于任务的分支和相应的单峰分支的权重。如 [ 63 ]所示,在同质中间融合中也可以实现对缺失模态的鲁棒性。
为了应对使 DL 架构在多模式环境中更具可解释性的挑战,人们提出了不同的方法。陈等人。[ 79 ] 通过将 Grad-CAM [ 97 ] 应用于 WSI 和集成梯度 [ 98 ] 使用卷积、图卷积 [ 14 ] 和完全连接的分支的细胞图和基因组模态,实现了模态特定的可解释性。在另一份出版物中,Chen等人[ 80 ] 将基于注意力和梯度的可解释性应用于 WSI 和分子模式。此外,对预测性能的贡献归因于不同的模式。康等人[ 73] 使用多组学数据的注意机制 [ 99 ] 来解释基因表达的预测。一般来说,这些基于梯度和注意力的方法表明,异构中间融合不会阻碍允许合理生物学解释的模型。
中间融合策略的优势在于,可以通过强制边缘表示具有相似的大小来减轻模态之间的维度不平衡。然而,如果不平衡非常大,过多地降低较大模态的维度可能会导致信息的大量丢失。严等[ 76 ] 融合了高维 WSI 和 29 个临床变量。为了获得更高的预测性能,临床变量被复制了 20 倍。然而,Mobadersanya等人。[ 77]表明,如果使用先验知识选择低维模态的输入特征,不平衡不一定会导致性能不佳。作者融合了从 WSIs 中学到的组织学特征和只有两个基因组特征,即异柠檬酸脱氢酶突变状态和 1p/19q 共缺失,以预测神经胶质瘤患者的存活率,并显示出统计上显着的性能提高。同样,尽管采用“边际”异质融合策略,Lu等人[ 66] 表明,将患者的生物学性别作为协变量与从 WSI 中学到的特征联系起来,可以提高预测未知原发癌症原发部位的性能。这表明通过仔细选择较小模态的变量可以有效地减轻不平衡。
跨模态交互的无监督学习有助于克服小样本量的限制。Cheerla 和 Gevaert [ 78 ] 提出了一种结合基因组、临床和 WSI 的无监督融合架构,用于癌症预后预测。损失函数的制定使得同一患者的不同模态的边际表征相似,而不同患者的边缘表征不同。因此,可以以无监督的方式学习每种模态的协调表示 [ 11 ],从而导致模态之间的模式编码。这种损失与 Cox 损失函数相结合,可以实现目标特定的特征学习。随后,从协调表示中学习了联合表示。
除了从边缘表示的级联向量中学习联合表示外,每个分支的特征向量也可以按元素聚合。这更明确地模拟了特征交互。例如,特征表示向量可以堆叠为矩阵中的列,并且可以取行方向的最大值、总和或乘积,从而得到与每个分支的边缘表示长度相同的联合向量。淡水河谷席尔瓦等人。[ 74 ] 比较了这些方法,尽管他们没有发现预测长期癌症存活率的性能有很大差异。陈等人。[ 79] 通过对 WSI、细胞相互作用和基因组数据进行建模来预测患者的存活率和几种患者分类。通过采用 Kronecker 产品融合边际表示。生成的三维张量明确编码了特征向量的单峰、双峰和三峰相互作用。张量被进一步输入到一个完全连接的网络,该网络连接到特定于目标的决策函数。作者还成功地将这种融合策略扩展到泛癌生存预测 [ 80 ]。除了逐元素聚合之外,还可以应用基于注意力的融合方法,以便根据重要性对不同的潜在特征进行加权[ 74、79、81 ]。
4.3 Discussion on intermediate fusion
DL 方法特别适合中间融合。分层边缘和联合表示可以在适当的抽象级别进行融合。因此,可以捕获融合策略中模式之间的潜在生物学关系。此外,从边缘表示中学习联合表示似乎是首选方法,正如更频繁地应用联合融合策略所表明的那样(见表 2)。这与模式独立影响目标的概念相矛盾,并支持多模式数据中互补和合作信息的想法。
中间融合方法还为 DL 在生物医学领域的其他普遍挑战提供了解决方案。例如,通过具有单独的分支,可以根据每种模态选择增强可解释性的方法。此外,如上所述,处理特征不平衡、缺失模态和协调表示学习是中间融合方法的优势。生物医学应用特别感兴趣的是中间融合的能力,它通过将不同的网络和网络类型应用于每种模态来缩小模态之间的异质性差距,从而实现成像、分子和临床模态的有效融合。这使 DL 方法更接近临床诊断和预后。
尽管中间融合策略似乎比其他方法具有许多理论上的优势,但很少研究或报告测试这些策略是否在给定问题上实现。如上所述,至少在某些任务上,早期融合可以执行类似于中间融合[ 38、41 ]。然而, [ 28 ] 表明中间 AE 明显优于其早期融合对应物。应用频率似乎在早期和中期融合之间保持平衡,尽管选择可能不仅受到策略性能的影响,而且还受到易用性的影响。
5. Late fusion
在后期融合中,为每种模式训练单独的模型。这些子模型可以被优化,以便它们学习p(y|xi)。其中xi 是来自第i个模态的数据。因为每个模态的输入提供了不同的信息,而且子模型的构造也不同,所以每个模型所犯的错误并不是完全相关的[2]。汇总预测概率的不同策略,从而利用每个模态的互补信息,对于异质模态的融合特别有希望。
聚合来自单独子模型的决策的最简单方法是取各个输出的平均值。对于分类任务,这可能是对每个类别的 softmax 函数的概率进行平均。这种方法假设每个子模型的贡献相同,因为没有对输出进行加权。邓等[ 82 ] 通过训练子模型融合不同类型的药物特征,然后通过平均 65 个类别的概率来汇总它们的预测。黄等人[ 83] 发现,在利用 CT 扫描和 EHR 数据预测肺栓塞检测方面,以正则化 DNN 作为子模型的基于平均的晚期融合优于早期、中期和其他晚期融合策略。索托等人[ 84 ]表明,具有平均结果的后期融合可以优于其他后期和中间融合策略。
为了避免假设所有子模型都具有相同的相关信息来预测目标,可以采用其他聚合方法。质疑这一假设是相关的,因为许多方法表明不同模式对预测性能的贡献不相等(例如参见 [ 36、42 ] )。王等人[ 85 ]通过其不确定性对每个子模型的预测概率进行加权。因此,更容易出错的模型对最终决策的贡献较小。这种方法可以减少最终预测中的不确定性。刘等人[ 86 ] 和 Sun等人[ 87] 通过它们的子模型学习预测的权重作为验证集上的超参数。
或者,元学习方法可以学习不同子模型的预测之间的复杂关系。在这种方法中,子模型的输出被输入到另一个分类器,该分类器学习预测之间的相互作用,以便做出更好的最终预测。尽管仍然无法学习不同模态特征之间的相关性,但可以有效地对跨模态(非线性)线性交互进行建模。黄等人[ 83 ] 应用 FCNN 来融合子模型预测,而 Reda等人[ 88 ]使用连接到分类器的稀疏 SAE 进行最终预测。
5.1 Discussion of late fusion
与早期融合相比,晚期融合可以对异构模态进行建模,甚至可以结合 DL 和浅层 ML 方法,如 Reda等人所做的那样。[ 88 ]。与中间融合类似,输入维度数量的不平衡不会影响最终预测,因此高维模态会“淹没”低维模态。后期融合策略显然具有无法学习不同模态特征之间交互的缺点。当模式相关性较低时,这些策略可能是有利的,因此这个缺点不会生效。
6. Discussion and conclusion
总之,回顾当前关于基于 DL 的融合策略的文献表明,多模态方法通常优于单模态方法。人们还普遍观察到,多模态 DL 方法明显优于浅层 ML 方法。虽然文献很可能倾向于报告积极的结果,但很明显,通过基于 DL 的融合获得的预期收益经常发生。
我们已经概述了早期、中期和晚期融合及其子类别在哪些条件下可能分别发挥最佳作用。主要地,选择取决于要分析的模式和研究人员做出或多或少的架构选择的意愿。然而,不同策略的性能仍然可能非常特定于问题和数据。需要更多的理论知识来进一步说明不同策略在什么条件下表现出色。因此,建议通过实验研究和比较不同的融合策略,并评估各自的优势。
多模态 DL 方法面临与 DL 在生物医学领域普遍面临的挑战相同,包括数据量、质量、可解释性和时间性,如 Miotto等人概述的那样[ 100 ]。但是,必须通过融合策略来解决多模态特定的挑战,例如整个模态的缺失。已经提出了不同的方法,例如多任务学习 [ 70 ]、生成模型 [ 63 ] 和多模态 dropout [ 78、101 ]. 为了变得更具临床相关性,方法需要对不同模式的缺失模式具有鲁棒性,并将对策纳入学习中。此外,随着越来越多的异构数据可用,融合策略需要适应这些模式组合。如上所述,生物过程可以在不同层面上观察到,多模式数据提供了训练整体模型的机会,这些模型可以学习健康和疾病背后的复杂监管动态。异构中间融合和后期融合特别适合这一挑战。
尽管这些挑战正在得到解决,但我们还是想概述一些尚未探索的领域。Ramachandram 和 Taylor [ 2 ] 概述了基于深度学习的融合的优势在于能够根据模式的相似性逐渐融合模式。我们还没有看到在当前的生物医学文献中对此进行了充分的探索。此外,逐步融合可以由先前的生物学知识指导,例如 mRNA、miRNA 和蛋白质之间的已知关系。我们已经看到先验生物学知识为架构决策提供信息的应用,例如根据染色体位置 [ 62 ] 的单独分支、训练损失中的正则化项 [ 49 ] 或将路径编码到架构中 [ 19,67 ]。然而,据我们所知,通知模式的逐渐融合尚未得到全面调查。
生物医学数据融合进一步探索的是如何自动找到最佳融合策略。由于设计融合架构所涉及的选择,找到融合不同模态的最佳方式变得非常重要。从这里审查的方法之间的比较可以看出,这种选择可以是高度特定于问题的。为 DL 架构寻找最佳融合策略是一个活跃的研究领域 [ 2 ],并且有望显着提高性能。徐等人[ 102] 已经应用搜索算法来找到最佳融合策略,以及用于融合 EHR 数据的特定于模态的神经架构搜索。除了这个提议的方法之外,我们发现这种策略没有在生物医学领域得到广泛研究或应用,并且可能会导致有趣的未来研究。
过度拟合训练数据,因此泛化性差,是多模态模型的主要挑战 [ 103 ]。特别是对于多模态生物医学数据集,样本量通常很小,因为生成它们的成本很高,而且生物材料的获取通常是有限的。通常输入变量的数量非常大,特别是如果包含多组学数据。另一方面,架构可以有很多参数,因为必须对多种模式进行建模。这很容易导致学习训练数据中的无信息模式。
迁移学习 (TL) 是将知识从一项任务迁移到相关任务,通常采用预训练网络权重的形式。使用 TL,可以显着减少所需的样本量 [ 104 ]。因此,应该进一步探索多模态生物医学数据集的 TL。尽管我们看到一些 TL 集成在融合策略中(例如 [ 50 ]),但我们相信利用 TL 的多模态架构的大量公共单峰数据集是一条有前途的未来道路。
随着越来越多的临床和实验数据可用,多模式数据融合在生物医学领域的重要性变得越来越明显。DL 融合策略是研究人员和从业者从他们的数据中构建性能最佳模型的有前途的选择。我们希望这篇综述能激发对这些方法的进一步应用和研究。
Key Points:
- 复杂的生物系统可以通过模态内和模态间的非线性函数进行有效建模。
- 多模态 DL 提供了有效且灵活的架构来融合不同抽象级别的同源和异源生物医学数据。
- 深度融合策略经常使用,并且经常优于浅层和单峰方法。
- 由于 TL 和渐进融合等领域尚未得到充分研究,因此多模态 DL 在生物医学领域的潜力仍未得到充分利用。
![[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review_第3张图片](http://img.e-com-net.com/image/info8/ea02718b35df402ca9f468c2df38cafa.jpg)
![[医学多模态融合系列 -4] Multimodal deep learning for biomedical data fusion: a review_第4张图片](http://img.e-com-net.com/image/info8/b8f6a5126d634ea18eaff21c84fca645.jpg)