使用sklearn.ensemble.RandomForestRegressor和GridSearchCV进行成人死亡率预测
原文链接:https://blog.csdn.net/weixin_44491423/article/details/127011461
本文借鉴博主hhhcbw实现方法完成随机森林回归预测成人死亡率,使用训练数据测试模型的最优得分R2=0.8161,在测试集上得分R2=0.5825
成年人死亡率指的是每一千人中15岁至60岁死亡的概率(数学期望)。这里我们给出了世界卫生组织(WHO)下属的全球卫生观察站(GHO)数据存储库跟踪的所有国家健康状况以及许多其他相关因素。要求利用训练数据建立回归模型,并预测成年人死亡率(Adult Mortality)
目录
- 实验要求
- 训练数据
- 数据预处理
-
- 可视化
- 特征间相关性
-
- 相关性矩阵热力图
- 数据间依赖关系
- 预处理
- 随机森林模型拟合
-
- `RandomForestRegressor` 模型
- `GridSearchCV` 网格搜索最优参数组
- 模型性能评估
实验要求
- 训练数据包含 2336 条记录和 22 个字段,对训练数据进行一定的可视化数据分析
- 利用训练数据,选择合适的信息作为特征建立回归模型,并预测测试数据成年人死亡率
- 测试数据(test_data.csv)总共包含 592 条记录,21 个字段,和训练数据相比,除了不包含 Adult Mortality 字段外,其他完全相同。
训练数据
训练数据(train_data.csv)包含 2336 条记录和 22 个字段,每个字段含义说明如下:
- Country:国家
- Year:年份
- Status:发达国家或发展中国家
- Life expectancy:预期寿命
- Infant deaths:每千人口中的婴儿死亡人数
- Alcohol:人均酒精消费量(以升纯酒精为单位)
- percentage expenditure:卫生支出占人均国内生产总值的百分比
- Hepatitis B:一岁儿童乙型肝炎免疫疫苗接种率
- Measles:麻疹每1000人报告的病例数
- BMI:所有人群平均BMI指数
- under-five deaths:每千人口中五岁以下死亡人数
- Polio:1岁儿童脊髓灰质炎免疫覆盖率(%)
- Total expenditure:政府卫生支出占政府总支出的百分比
- Diphtheria:1岁儿童白喉、破伤风类毒素和百日咳免疫接种率(%)
- HIV/AIDS:每千名活产婴儿死于艾滋病毒/艾滋病(0-4岁)
- GDP:人均国内生产总值(美元)
- Population:人口
- thinness 1-19 years:10至19岁儿童和青少年的消瘦流行率
- thinness 5-9 years:5至9岁儿童中的消瘦流行率
- Income composition of resources:财力收入构成方面的人类发展指数(从0到1)
- Schooling:受教育年限
- Adult Mortality:成人死亡率(每1000人中15至60岁死亡的概率)
其中Adult Mortality字段为要预测的标签(label)
数据预处理
可视化
# 数据读取和可视化分析
model_filename = './model.pkl'
imputer_filename = './imputer.pkl'
scaler_filename = './scaler.pkl'
train_data = pd.read_csv('./data/train_data.csv')
trian_data
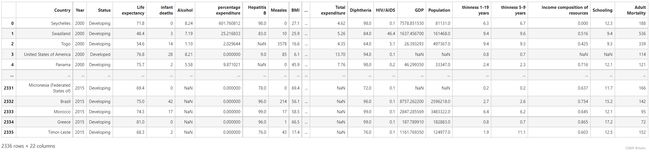
可以看到训练数据中存在一些字段值的缺失NaN,数据中的缺失值是一个非常棘手的问题,很多文献都致力于解决这个问题。那么,数据缺失究竟带来了什么问题?假设有100个样本和20个特征,这些数据都是机器收集回来的。若机器上的某个传感器损坏导致一个特征无效时该怎么办?它们是否还可用?答案是肯定的。因为有时候数据相当昂贵,扔掉和重新获取都是不可取的,所以必须采用一些方法来解决这个问题。下面给出了一些可选的做法:
- 使用可用特征的均值来填补缺失值;
- 使用特殊值来填补缺失值,如-1;
- 忽略有缺失值的样本;
- 使用相似样本的均值添补缺失值;
- 使用另外的机器学习算法预测缺失值。
本文经过测试比较最终使用该特征均值来补全缺失值。
特征间相关性
下面计算各个特征之间的皮尔森相关系数,皮尔森相关系数可以理解为特征与特征之间的线性相关程度,取值[-1,1],正数就是正相关,负数就是负相关。且绝对值越大,即越接近1,相关程度越高。
# 计算各个特征之间的皮尔森相关系数
train_data.corr()
相关性矩阵热力图
# 将相关性矩阵绘制成热力图
corr = train_data.corr()
corr.style.background_gradient(cmap='coolwarm')

根据原博文中的分析:从热力图可以看出 infant deaths 与 under-five deaths 有很强的正相关,即 每千人口中的婴儿死亡人数 与 每千人口中五岁以下死亡人数 有很强的正相关;除此之外,也可以看到 thinness 1-19 years 和 thinness 5-9 years 有很强的正相关,也是一样的道理。
在实际的预处理中,本文并没有对这种相似特征进行去除,而是利用随机森林的特性得到最优随机森林模型允许单个决策树使用特征的最大数量(max_features)参数值。
数据间依赖关系
# 利用seaborn检查可视化数据之间的依赖关系
corr.style.background_gradient(cmap='coolwarm')
预处理
- 补全缺失值:采用均值填充缺失值,
SimpleImputer(strategy='mean', missing_values=np.nan) - 归一化:采用最小最大归一化,
MinMaxScaler()。通常情况下,在建模之前,都需要对数据进行标准化处理,以消除量纲的影响。如果对未标准化的数据直接进行建模,可能会导致模型对数值大的变量学习过多,而对数值小的变量训练不够充分,往往模型效果会不好。常用的数据标准化方法有最大最小归一化、均值方差标准化、小数定标法、定量特征二值化等。最大最小归一化,顾名思义,就是利用数据列中的最大值和最小值进行标准化处理,标准化后的数值处于[0,1]之间,计算方式为数据与该列的最小值作差,再除以极差。具体公式为:
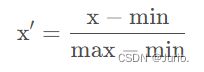
def preprocess_data(data, imputer=None, scaler=None):
column_name = ['Year', 'Life expectancy ', 'infant deaths', 'Alcohol',
'percentage expenditure', 'Hepatitis B', 'Measles ', ' BMI ', 'under-five deaths ',
'Polio', 'Total expenditure', 'Diphtheria ', ' HIV/AIDS', 'GDP', 'Population',
' thinness 1-19 years', ' thinness 5-9 years', 'Income composition of resources',
'Schooling']
data = data.drop(["Country", "Status"], axis=1)
if imputer==None:
imputer = SimpleImputer(strategy='mean', missing_values=np.nan)
imputer = imputer.fit(data[column_name])
data[column_name] = imputer.transform(data[column_name])
if scaler==None:
scaler = MinMaxScaler()
scaler = scaler.fit(data)
data_norm = pd.DataFrame(scaler.transform(data), columns=data.columns)
# data_norm = data_norm.drop(['Year'], axis = 1)
return data_norm, imputer, scaler
随机森林模型拟合
RandomForestRegressor 模型
首先我们简单学习一下sklearn的随机森林回归模型:
官方英文文档地址:https://scikit-learn.org/dev/modules/generated/sklearn.ensemble.RandomForestRegressor.html#sklearn.ensemble.RandomForestRegressor
class sklearn.ensemble.RandomForestRegressor(n_estimators=100, *, criterion='squared_error',
max_depth=None, min_samples_split=2, min_samples_leaf=1, min_weight_fraction_leaf=0.0,
max_features=1.0, max_leaf_nodes=None, min_impurity_decrease=0.0, bootstrap=True, oob_score=False,
n_jobs=None, random_state=None, verbose=0, warm_start=False, ccp_alpha=0.0, max_samples=None
参数说明如下:
n_estimators:整数,可选择(default=100)。 森林里(决策)树的数目。criterion:{“squared_error”, “absolute_error”, “friedman_mse”, “poisson”},(default=“squared_error”)。回归树衡量分枝质量的指标。“squared_error”,它等于作为特征选择标准的方差减少,并使用每个终端节点的均值来最小化 L2 损失;“friedman_mse”,它使用均方误差和弗里德曼的潜在改进分数分割;“absolute_error”表示平均绝对误差,它使用每个终端节点的中值最小化 L1 损失;“poisson”使用泊松偏差的减少来找到分割。使用“absolute_error”进行训练比使用“squared_error”要慢得多。max_depth:整数,(default=None)。树的最大深度。如果为 None,则扩展节点直到所有都是纯叶子结点或直到所有叶子包含少于 min_samples_split 样本。min_samples_split:整数或浮点数,(default=2)。根据属性划分节点时,每个划分最少的样本数。min_samples_leaf:整数或浮点数,(default=1)。叶子节点最少的样本数。min_weight_fraction_leaf:浮点数,(default=0.0)。需要在叶节点处的权重总和(所有输入样本的)的最小加权分数。当未提供 sample_weight 时,样本具有相同的权重。max_features:{“sqrt”, “log2”, None}, 整数或浮点数, (default=1.0)。寻找最佳分割时要考虑的特征数量。- if “int”, max_features即最大特征数
- if “float”, n_features*max_features即最大特征数
- if “auto”, then max_features=sqrt(n_features)
- If “sqrt”, then max_features=sqrt(n_features).
- If “log2”, thenmax_features=log2(n_features).
- If None, then max_features=n_features.
max_leaf_nodes:整数,(default=None)。叶子树的最大样本数。min_impurity_decrease:浮点数,(default=0.0)。如果该分裂导致杂质减少大于或等于该值,则该节点将被分裂。bootstrap:布尔值,(default=True)。构建树时是否有放回的采样。如果为 False,则使用整个数据集来构建每棵树。oob_score:布尔值,(default=False)。是否使用袋外样本来估计泛化分数。仅当 bootstrap=True 时可用。n_jobs:整数,(default=None)。并行job个数。这个在ensemble算法中非常重要,尤其是bagging(而非boosting,因为boosting的每次迭代之间有影响,所以很难进行并行化),因为可以并行从而提高性能。1=不并行;n:n个并行;-1:CPU有多少core,就启动多少job。random_state:整数,(default=None)。控制构建树时使用的样本引导的随机性(如果bootstrap=True)和在每个节点处寻找最佳分割时要考虑的特征采样。verbose:整数,(default=0) 。是否显示任务进程。warm_start:布尔值,(default=False)。当设置为 时True,重用上一次调用的解决方案来拟合并添加更多的估计器到集成中,否则,只适合一个全新的森林。ccp_alpha:非负浮点数,(default=0.0)。用于最小成本复杂度修剪的复杂度参数。将选择具有最大成本复杂度且小于 的子树 ccp_alpha。默认情况下,不进行剪枝。max_samples:整数或浮点数,(default=None)。如果 bootstrap 为 True,则从 X 抽取的样本数以训练每个基本估计器。
(参数真的好多…)
同时,RandomForestRegressor提供了以下属性和方法供我们使用,具体参考官方文档(懒得翻译手打了)。

GridSearchCV 网格搜索最优参数组
GridSearchCV就不赘述了,详见官方文档:https://scikit-learn.org/stable/modules/generated/sklearn.model_selection.GridSearchCV.html
直接上代码:
def gridsearch():
# 需要网格搜索的参数
n_estimators = [i for i in range(100, 551, 20)]
max_depth = [i for i in range(2, 10)]
min_samples_split = [i for i in range(2, 10)]
min_samples_leaf = [i for i in range(1, 10)]
max_features = [i for i in range(10, 20)]
max_samples = [i / 100 for i in range(90, 100)]
parameters = {'n_estimators': n_estimators,
'max_depth': max_depth,
'min_samples_split': min_samples_split,
'min_samples_leaf': min_samples_leaf,
'max_features': max_features,
'max_samples': max_samples}
# 随机森林回归
regressor = RandomForestRegressor(bootstrap=True, oob_score=True, random_state=1)
best_model = GridSearchCV(regressor, parameters, refit=True, cv=6, verbose=1, n_jobs=-1)
x_data_norm, imputer, scaler = preprocess_data(x_train)
train_x = x_data_norm.values
best_model.fit(train_x, y_train)
joblib.dump(best_model, model_filename)
joblib.dump(imputer, imputer_filename)
joblib.dump(scaler, scaler_filename)
return best_model
# GridSearch寻找最优模型参数组
t1 = time.time()
best_model = gridsearch()
t2 = time.time()
print(f'GridSearch cost:{t2-t1} s')
print(f'Best params:{best_model.best_params_}')
print(f'Best score:{best_model.best_score_}')
得到最优参数组和最优模型后,保存最优模型文件到本地。
模型性能评估
- 定义测试函数:
def predict(test_data, filename):
loaded_model = joblib.load(model_filename)
imputer = joblib.load(imputer_filename)
scaler = joblib.load(scaler_filename)
test_data_norm, _, _ = preprocess_data(test_data, imputer, scaler)
test_x = test_data_norm.values
predictions = loaded_model.predict(test_x)
return predictions
- 首先看一下模型在训练集上的表现:
# 模型性能评估
label = train_data.loc[:, 'Adult Mortality']
data = train_data.iloc[:, :-1]
# label = y_test
# data = x_test
y_pred = predict(data, './model.pkl')
r2 = r2_score(label, y_pred)
mse = mean_squared_error(label, y_pred)
print("MSE is {}".format(mse))
print("R2 score is {}".format(r2))
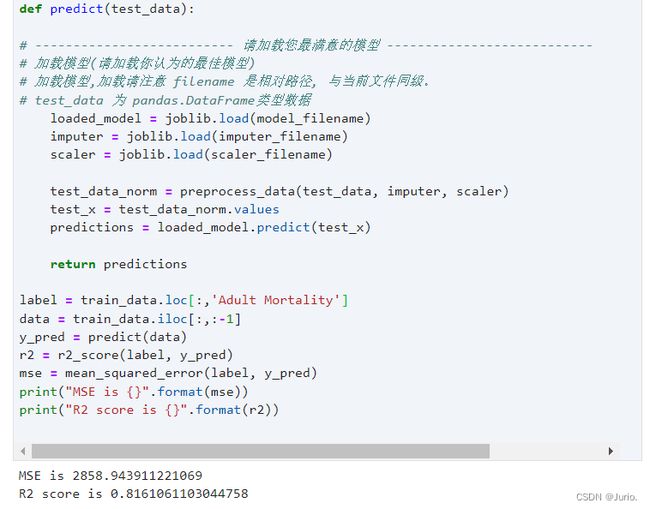
3. 最后使用未知的测试集进行测试:

该测试结果得分应该是R2_score * 100的结果,可以看到存在一定过拟合,后续可以通过优化数据预处理方法、更换更合适的回归模型预测进行改进。
PS: 如果觉得本篇本章对您有所帮助,欢迎关注、评论、赞!同时有任何问题欢迎批评指正,谢谢!
训练数据及代码均在GitHub开源,欢迎Follow、Star(引用给个star,球球了):https://github.com/Jurio0304/RandomForestRegressor-AdultMortality-Prediction

