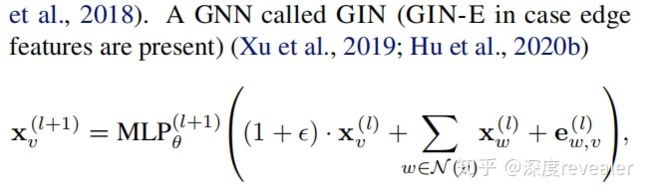带你读论文:分子图表示学习
论文:《Hierarchical Inter-Message Passing for Learning on Molecular Graphs》
背景介绍:
分子图表示学习:一般来说,我们所使用的一些方法是对一个图中的结点或者边进行表示学习,学习出他的embedding,而分子图表示学习是指学习出一整个分子图的embedding。
分子图表示学习的意义:
1.对于分子特征或者功能学习有重要意义
2.对于我们当前的一些任务,使用SMILES等方式来表示分子结构或者序列,丢失信息较多,有时SMILES等特征发现对结果提升并不高
方法:
常规图表示学习流程:
如上图所示,常规图表示流程一般是先有特征或者随机初始化的实体变量特征,然后首先进行聚合操作(aggregate),也就是将邻居节点的信息聚合到本节点,之后就是对自己的信息和聚合的信息进行操作,来update自己的特征。然后,每个节点就有了自己的特征,就可以做一些分类或者link prediction的任务了,而分子表达学习这里会将所有的节点的特征进行累加或者其他的求和操作,来得到整个分子图的embedding,毕竟对于一个分子图来说,一个分子就是一个graph,而不是其他方法中代表某一个生物实体。
而本文主要解决的是什么问题呢?
常规图表示学习遇到的问题:环状结构不好进行检测与处理
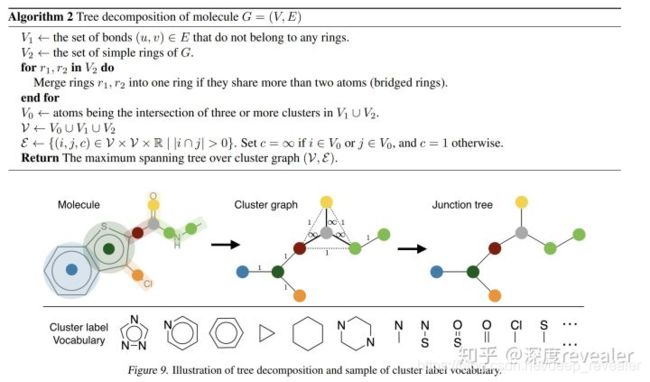
解决逻辑思路:这种带有环状结构的分子图,能否将环状结构转换成其他的结构呢?换言之,有没有一种方法能够将有环图转化为无环图并且能够保证分子的大致结构呢?有,图的树分解
图的树分解的最初工作可以追溯到1998年【1】,他的主要目的就是将分子图转换为一个树形图,而这个树形图中的结点就不再是单一结点,而被称为 cluster(簇)
【1】Rarey, M. and Dixon, J. S. Feature trees: A new molecular similarity measure based on tree matching. Journalof Computer-aided Molecular Design, 12(5):471–490,1998.
方法:
首先是本文基于的对图进行树分解的方法,可以理解为将一个graph转成一个tree的过程
这里主要是一段介绍tree decomposition的方法,其实核心思路就是将多个节点看作是一个整体,成为簇,然后labeling,这样,环状结构就变成了一个单独的节点,这样最后的graph中就不含环状图了。
本文方法:本文融合了原分子图以及原分子图进行树分解之后的树型图两种图的表示方法,本文有两套模型,分别从原分子图以及树型图中进行图表达学习,又学习了原分子图,又排除了环状结构的干扰,然后最后将他们进行拼接,其中作者还提出了一种粗到细,细到粗的模型间的传递方法。模型大体如下:
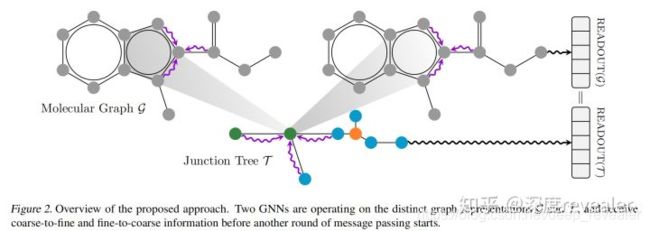
这中间,有一段互相传递的过程,因为直接从树图中进行消息传递和更新,会丢失许多结构信息和一些其他信息,所以还需要在一定程度上保留原图的信息,所以要原图和转成的树图一起来进行消息传递和更新,这中间,他们有一个交流的过程:
然后是将两者最终的结果拼接起来:
实验结果分析:
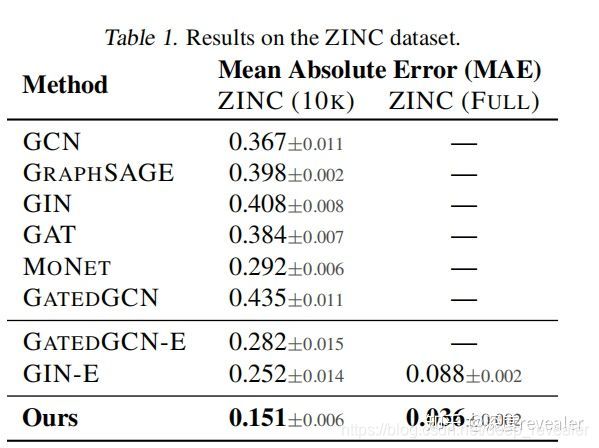
虽然这是一个简单的可以通过直接计算得到的数值,但是这个实验依然能够证明方法的有效性。
任务是预测某些分子特性(以二进制标记显示),例如,某个分子是否抑制HIV病毒复制,可以理解为多分类任务
实验3:OGB Datasets实验:
并且这里还有一个结论:
他们的模型结合少量的层(2层或3层)可达到最佳效果,使其运行时和内存需求与其他baseline(使用5层)相当。 这可以通过以下事实来解释:附加的从粗到细的信息流增加了GNN的接收场大小,因此省略了堆叠多个层的需要。
启发:
1.主要是作为后续的研究方向进行的调研
2.当某种问题不好解决的时候尝试考虑另外一种view,用以解决方法局限问题
3.分子表达学习的话有标准数据集,可能以此为研究方向的话数据方面可能相对来说收集难度稍微小一些。
4.类似attention的机制还可以继续进行开发,分子表达学习应该暂时还进展不深,相关文献不多,但是无论是单独研究,还是通过这种新颖的方式加入到我们现有的工作中,这些都感觉是很不错的工作。
欢迎关注我的CSDN博客:https://blog.csdn.net/deep_revealer
欢迎关注我的微信公众号:深度revealer
欢迎关注我的github:https://github.com/Zhankun-Xiong