scRNA-seq | 吐血整理的单细胞入门教程(从原理到代码实操)(一)

1写在前面
炙手可热的单细胞测序(scRNA-seq)大家肯定都或多或少听过一些,从今天开始陆续分享我的学习笔记,有不对的地方大家多指正~
在开始前我们先思考几个问题,如下:
Q1: 什么是
scRNA-seq,它与bulk RNA-seq相比如何?Q2:
scRNA-seq有哪些典型应用?Q3:
scRNA-seq如何准备样品?
2什么是scRNA-seq?
2.1 什么是bulk RNA-seq
要搞懂什么是scRNA-seq,我们先了解一下bulk RNA-seq。
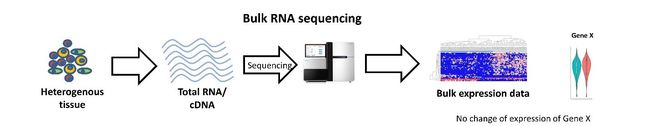
RNA-seq用于由细胞混合物组成的样本中,称为bulk RNA-seq,常用于研究control/diseased、wild-type/mutant之间的转录组差异。
然而,使用bulk RNA-seq,我们只能估计每个基因在细胞群中的平均表达水平☹️,没有考虑该样本中单个细胞基因表达的异质性。
举个栗子,早期发育研究或大脑等复杂组织。
2.2 scRNA-seq横空出世
为了解决异质性的问题,2009年首次报道了单细胞水平的RNA-seq,即scRNA-seq,
与bulk RNA-seq不同的是,使用scRNA-seq可以评估每个基因在不同细胞群中的表达水平分布。
成功解决了转录组中细胞特异性变化的问题。可以发现新的或稀有的细胞类型,识别control/diseased组织之间的差异细胞组成或了解发育过程中的细胞分化。

2.3 scRNA-seq图谱
目前有很多的scRAN-seq图谱,全面解析了不同物种中的细胞类型。举几个栗子,如下:
Human Cell Atlas (H. sapiens)Tabula Muris (M. musculus)Fly Cell Atlas (D. melanogaster)Cell Atlas of Worm (C. elegans)Arabidopsis Root Atlas (A. thaliana)
随着技术的进步,scRNA-seq方法学层出不穷,自首次报道后的技术发展,我们可以看到随着技术的进步,scRNA-seq可以检测到更多的细胞。
但需要说明一下,不同的技术各有其优缺点,还是老观点,新技术不一定是最好,选适合你的就行了。

3scRNA-seq的样品制备
3.1 经典protocol
通常来说,经典的scRNA-seq的protocol可以分为以下几个步骤:
- 从组织和细胞培养中制备 细胞悬浮。
- 细胞筛选(基于 表面marker、转基因技术、染色等)。( 可选步骤)
- 捕获 单个细胞(如
wells,oil droplets)。 - 从 每个细胞中提取 RNA。
- 将 RNA反转录为 cDNA。
- 扩增 cDNA( 体外转录或 PCR)。
- 准备测序文库,
adapters。 - 测序。
- 处理
raw data以获得细胞基因的count矩阵。 - 进行下游分析。

3.2 制备样品困难怎么办?
在难以分离细胞的组织中或在冷冻组织样本中,可能较难实现单细胞的制备,我们可以选择制备单个细胞核样本。
Note! 但需要注意一下,核RNA通常含有较高比例的未加工unprocessed RNA,,会测序到大量含有introns的转录本。
具体的解决方案,我们后面再介绍吧。
3.3 常用protocol的对比
这里我们比较一下目前常用的protocol,这里不包括Smart-seq3和Smart-seq3xpress,后面我们单独介绍这两种方法。

4细胞捕获
目前应用最广泛的三种方法为microtitre-plate, microfluidic-array 和 microfluidic-droplet。
各有其优缺点,这里只做简单介绍,感兴趣的小伙伴可以去Google一下具体差异。
1️⃣ Microtitre-plate
- 优点是建库之前可以对细胞进行观察,识别并丢弃 受损的细胞等。
- 缺点是 低通量。
2️⃣ Microfluidic-array
- 优点是通量比 Microtitre-plate通量高,省试剂。
- 缺点是不适合 稀有细胞的处理。
- 这里提醒大家要注意一下
arrays的nanowells大小问题。
3️⃣ Microfluidic-droplet
- 优点是高通量,低成本。
- 缺点当然也是低成本带来的啦,一般
coverage都比较低,往往检测深度就不够了,transcripts比较少。

Tips! 如果用FACS分离细胞的话,可以染一下live/dead, 避免一些活力低的细胞影响结果。

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰

本文由 mdnice 多平台发布