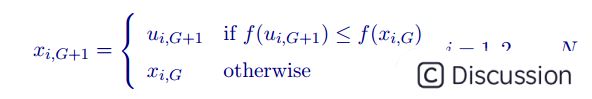【优化求解】差分进化算法(Differential Evolution)matlab源码
差分进化算法DE属于进化算法,这里算法还包括依次遗传算法、进化策略、进化规划。
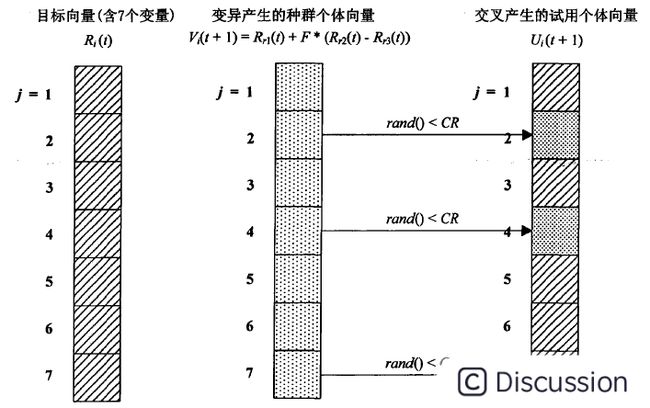
差分进化算法包括三个基本的操作:变异操作、交叉(重组)操作和选择操作。
一、算法建模:
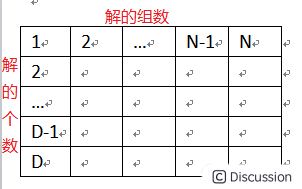
1、假设我们希望得到函数f(x)的最优解,这个函数有D个解。\ 2、为函数f(x)设置一个解的组数N,N至少为4。\ 3、这样我们就得到了N组并且每组解的个数为D的集合,它可以使用N个D维参数向量来表示。\ ![]() \ 因为它类似于遗传算法进化一样,是一代一代的进行进化,最终得到最优个体。所以上面G表示的就是代数。
\ 因为它类似于遗传算法进化一样,是一代一代的进行进化,最终得到最优个体。所以上面G表示的就是代数。
二、初始化
为每个参数定义上界和下界\  \ 在上面的范围内随机的为每个参数取值。这样就得到了一个N组初始解。
\ 在上面的范围内随机的为每个参数取值。这样就得到了一个N组初始解。
上面有N组解,对于一组给定的解X(i,G)随机的从这N组解中选择三组解X(r1,G),X(r2,G),X(r3,G),r1,r2,r3分别代表组的索引,G表示代数,从第一代开始。
其中,F是变异因子,位于[0,2]之间。这样我们就可以得到一组新的解。
四、交叉
下面我们就会对得到的这组新解进行交叉操作了。
CR是交叉概率,处于[0, 1]之间。
五、选择
从上面可以得到一组进化之后的解,为了决定这组解是否成为G+1代中的解,需要将这组新解跟原来那组解的适应度值进行比较,如果优于原来那组解则将它们替换掉,否则保留原来解。适应度值得计算使用的就是适应度函数f(x)。这个函数需要我们之前进行确定。
整个过程的流程图如下:
``` function demo1 %DEMO1 Demo for usage of DIFFERENTIALEVOLUTION.
% Set title optimInfo.title = 'Demo 1 (Rosenbrock''s saddle)';
% Specify objective function objFctHandle = @rosenbrocksaddle;
% Define parameter names, ranges and quantization:
% 1. column: parameter names % 2. column: parameter ranges % 3. column: parameter quantizations % 4. column: initial values (optional)
paramDefCell = { 'parameter1', [-3 3], 0.01 'parameter2', [-3 3], 0.01 };
% Set initial parameter values in struct objFctParams objFctParams.parameter1 = -2; objFctParams.parameter2 = 2.5;
% Set single additional function parameter objFctSettings = 100;
% Get default DE parameters DEParams = getdefaultparams;
% Set number of population members (often 10*D is suggested) DEParams.NP = 20;
% Do not use slave processes here. If you want to, set feedSlaveProc to 1 and % run startmulticoreslave.m in at least one additional Matlab session. DEParams.feedSlaveProc = 0;
% Set times DEParams.maxiter = 20; DEParams.maxtime = 30; % in seconds DEParams.maxclock = [];
% Set display options DEParams.infoIterations = 1; DEParams.infoPeriod = 10; % in seconds
% Do not send E-mails emailParams = [];
% Set random state in order to always use the same population members here setrandomseed(1);
% Start differential evolution [bestmem, bestval, bestFctParams, nrOfIterations, resultFileName] = differentialevolution(... DEParams, paramDefCell, objFctHandle, objFctSettings, objFctParams, emailParams, optimInfo); %#ok
disp(' '); disp('Best parameter set returned by function differentialevolution:'); disp(bestFctParams);
% Continue optimization by loading result file if DEParams.saveHistory
disp(' '); disp(textwrap2(sprintf(... 'Now continuing optimization by loading result file %s.', resultFileName))); disp(' ');
DEParams.maxiter = 100; DEParams.maxtime = 60; % in seconds
[bestmem, bestval, bestFctParams] = differentialevolution(... DEParams, paramDefCell, objFctHandle, objFctSettings, objFctParams, emailParams, optimInfo, ... resultFileName); %#ok
disp(' '); disp('Best parameter set returned by function differentialevolution:'); disp(bestFctParams); end ```