[GWAS]plink数据格式的转化和缺失质控
本篇笔记整理于邓飞大佬的GWAS学习教程,他在B站上发布了一系列教学课程,推荐大家去看看
plink数据格式的转化
plink的bfile格式,二进制不方便查看,我们将其转化为文本map和ped的格式。
plink --bfile [bfile格式的二进制文件] --recode --out test
最后的–out代表输出为test文件.
结果:
PLINK v1.90b7 64-bit (16 Jan 2023) www.cog-genomics.org/plink/1.9/
(C) 2005-2023 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to plink.log.
Options in effect:
--bfile HapMap_3_r3_1
--recode
14204 MB RAM detected; reserving 7102 MB for main workspace.
1457897 variants loaded from .bim file.
165 people (80 males, 85 females) loaded from .fam.
112 phenotype values loaded from .fam.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 112 founders and 53 nonfounders present.
Calculating allele frequencies... done.
Warning: 225 het. haploid genotypes present (see plink.hh ); many commands
treat these as missing.
Total genotyping rate is 0.997378.
1457897 variants and 165 people pass filters and QC.
Among remaining phenotypes, 56 are cases and 56 are controls. (53 phenotypes
are missing.)
--recode ped to plink.ped + plink.map ... done.
我们来查看生成的结果
ls test.*
wc -l test.map test.ped
![[GWAS]plink数据格式的转化和缺失质控_第2张图片](http://img.e-com-net.com/image/info8/fc3760b09f6f4cd3875d4660c5e09d24.jpg)
我们可以看到map文件中有1457897行,代表有1457897个SNP数据
ped文件中有165行,代表有165个基因型个体
我们分别预览一下两个文件
less -S test.ped #点击q退出
map文件和.ped文件
格式说明链接:link
map格式的文件, 主要是图谱文件信息, 主要包括染色体名称, 所在的染色体和所在染色体的坐标.
map文件包括:
- 染色体编号(1-22, X, Y or 0 if unplaced), 未知为0
- SNP名称(字符或数字), 如果不重要, 可以从1编号, 注意要和bed文件SNP列一一对应
- 染色体的摩尔位置(可选项, 可以用0)
- SNP物理坐标
Example
1 snp1 0 1
1 snp2 0 2
1 snp3 0 3
- 这里有3个SNP, 分别名为snp1, snp3, snp3 (第二列)
- 这三个SNP在第一个染色体上 (第一列)
- 第三列为0
- 第四列为SNP所在染色体的坐标
.ped文件
格式说明链接:link
bed格式的文件, 主要包括SNP的信息, 包括个体ID, 系谱信息, 表型和SNP的分型信息.
.ped 文件主要有 6 列,后面都是基因型:
- 第一列: Family ID # 如果没有, 可以用个体ID代替
- 第二列: Individual ID # 个体ID编号
- 第三列: Paternal ID # 父本编号
- 第四列: Maternal ID # 母本编号
- 第五列: Sex (1=male; 2=female; other=unknown) # 性别, 如果未知, 用0表示
- 第六列: Phenotype (0=unknown; 1=unaffected; 2=affected) # 表型数据, 如果未知, 用0表示
- 第七列以后: 为SNP分型数据, 可以是AT CG或11 12, 或者A T C G或1 1 2 2
Example
1 1 0 0 1 0 G G 2 2 C C
1 2 0 0 2 0 A A 0 0 A C
1 3 1 2 1 2 0 0 1 2 A C
2 1 0 0 1 0 A A 2 2 0 0
2 2 0 0 2 2 A A 2 2 0 0
2 3 1 2 1 2 A A 2 2 A A
- 数据包括两个家系 (第一列)
- 每个家系有三个个体 (第二列)
- 第三列父本编号
- 第四列母本编号
- 第五列性别
- 第六列表型值
- 第七列, 第八列为一个基因型
- 第九列, 第十列为第二个基因型
- 第十一列, 第十二列为第三个基因型
缺失质控
缺失指控,一般用两个参数
- –geno
- –mind
–geno筛选SNP; --mind筛选个体
GWAS分析时,拿到基因型数据,拿到表型数据,要首先做到以下几点
- 查看自己的表型数据是否有问题
- 查看自己的基因型数据是否有问题
然后再进行建模,得到显著性SNP以及可视化结果,清洗数据的时间占80%的时间,可见清洗数据很重要.
那么问题来了,为什么要对缺失的数据进行筛选呢?
无论是测序还是芯片,得到的基因型数据要进行质控,而对于缺失数据进行筛选,可以去掉低质量的数据.
如果一个个体,共有50万个SNP数据,发现20%的SNP数据(10万)都缺失,所以我们可以把它去除掉.
同样的道理,如果某个SNP,在500个样本中,缺失率为20%(即该SNP在199个个体中都没有分型结果),我们也可以认为该SNP质量交叉,将它去除掉.
当然,这里的20%是过滤的标注,可以改变质控标准.
我们下面的质控标准是2%
首先,我们查看一下个体缺失的位点数目,每个SNP缺失的个体数
命令:
plink --file test --missing
结果:
$ plink --file test --missing
PLINK v1.90b7 64-bit (16 Jan 2023) www.cog-genomics.org/plink/1.9/
(C) 2005-2023 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to plink.log.
Options in effect:
--file test
--missing
14204 MB RAM detected; reserving 7102 MB for main workspace.
.ped scan complete (for binary autoconversion).
Performing single-pass .bed write (1457897 variants, 165 people).
--file: plink-temporary.bed + plink-temporary.bim + plink-temporary.fam
written.
1457897 variants loaded from .bim file.
165 people (80 males, 85 females) loaded from .fam.
112 phenotype values loaded from .fam.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 112 founders and 53 nonfounders present.
Calculating allele frequencies... done.
Warning: 225 het. haploid genotypes present (see plink.hh ); many commands
treat these as missing.
Total genotyping rate is 0.997378.
--missing: Sample missing data report written to plink.imiss, and variant-based
missing data report written to plink.lmiss.
–missing
–missing: Sample missing data report written to plink.imiss, and variant-based missing data report written to plink.lmiss.
结果生成了两个文件,分别是一个个体ID上SNP缺失的信息,另一个是每个SNP在个体ID中缺失的信息
- 个体缺失位点的统计在plink.imiss中
- 单个SNP缺失的个体数在plink.lmiss中
个体缺失位点统计预览:
- 第一列为家系ID
- 第二列为个体ID
- 第三列为是否表型缺失
- 第四列缺失的SNP个数
- 第五列总SNP个数
- 第六列缺失率
SNP缺失的个体数文件预览
- 第一列为染色体
- 第二列为SNP名称
- 第三列为缺失个数
- 第四列为总个数
- 第五列为缺失率
R语言做直方图
# 先要设置工作路径
indmiss = read.table(file="plink.imiss", header=TRUE)
snpmiss = read.table(file="plink.lmiss", header=TRUE)
# read data into R
summary(indmiss)
summary(snpmiss)
# 显示图片
hist(indmiss[,6],main="Histogram individual missingness") #selects column 6, names header of file
hist(snpmiss[,5],main="Histogram SNP missingness")
# 保存图片
pdf("histimiss.pdf") #indicates pdf format and gives title to file
hist(indmiss[,6],main="Histogram individual missingness") #selects column 6, names header of file
dev.off()
pdf("histlmiss.pdf")
hist(snpmiss[,5],main="Histogram SNP missingness")
dev.off() # shuts down the current device
先展示summary的结果![[GWAS]plink数据格式的转化和缺失质控_第9张图片](http://img.e-com-net.com/image/info8/c368a75a53c9471cb64ad154ac3f5ef6.jpg)
我们可以查看到indmiss和snpmiss的数据信息,对应的是:
- Min最小数
- 1st Qu.1/4位数
- Median中位数
- Mean均值
- 3st Qu.3/4位数
- Max 最大数
![[GWAS]plink数据格式的转化和缺失质控_第10张图片](http://img.e-com-net.com/image/info8/f9ec3859582b4bb5b31985ae29578b55.png)
输出为pdf
个体缺失位点的频率分别在20,40,60,80对应的概率
- 如果一个SNP在个体中2%都是缺失的,那么就删掉该SNP,参数为: --geno 0.02
- 如果一个个体,有2%的SNP都是缺失的,那么就删掉该个体,参数为: – mind 0.02
首先,我们对SNP缺失率进行筛选,先过滤掉SNP缺失率高于2%的SNP
plink --bfile [你的genoType文件] --geno 0.02 --make-bed --out [输出的文件]
![[GWAS]plink数据格式的转化和缺失质控_第13张图片](http://img.e-com-net.com/image/info8/823128bcbf054162a0f95e64dd4a5784.jpg)
看不懂嘛?那就翻译一下,凑活看吧![[GWAS]plink数据格式的转化和缺失质控_第14张图片](http://img.e-com-net.com/image/info8/a13082f1daf940fdaf657024e84d4cfb.jpg)
接着,我们转化为map和ped的形式
plink --bfile [上个输出文件作为这里的输入文件] --recode --out test
![[GWAS]plink数据格式的转化和缺失质控_第15张图片](http://img.e-com-net.com/image/info8/ee958ec4a92d40ed9e71ac18262af9be.jpg)
查看一下过滤之后的行数,之前的是:![[GWAS]plink数据格式的转化和缺失质控_第16张图片](http://img.e-com-net.com/image/info8/d33203c06828484a9aa3838996423db4.png)
现在的是:![[GWAS]plink数据格式的转化和缺失质控_第17张图片](http://img.e-com-net.com/image/info8/e64ff50e2fc844caa30a98c32e8bfca0.png)
我们可以看到这里过滤掉了2万多个位点
从当时的log日志里面其实也可以看出这一点![[GWAS]plink数据格式的转化和缺失质控_第18张图片](http://img.e-com-net.com/image/info8/3e227cc523144e66baeb4a89ea97a1a6.jpg)
可以看到–geno,过滤掉了27454个位点
对样本缺失率进行筛选,过滤掉缺失率高于2%的个体(在之前过滤掉的基础上进行筛选)
plink --bfile HapMap_3_r3_2 --mind 0.02 --make-bed --out HapMap_3_r3_3
![[GWAS]plink数据格式的转化和缺失质控_第19张图片](http://img.e-com-net.com/image/info8/7699e2b3582f4133a5b9e6d21029b077.jpg)
我们注意到日志中的这句话:
0 people removed due to missing genotype data(–mind)
代表没有过滤掉个体,所以剩余个体:165;剩余SNP:1430443
同时我们对个体和SNP的缺失率进行筛选,要注意,在正式分析的时候,一般先进行SNP的质控,然后再进行样本的质控.
意思就是:在运行代码的时候,先运行"–geno",再运行’–mind’,不能两步合成一步.![[GWAS]plink数据格式的转化和缺失质控_第20张图片](http://img.e-com-net.com/image/info8/5f7e0498da8c4cc996399d96d66ba0d3.jpg)
这个问题在邓飞老师的公众号中有提到.![[GWAS]plink数据格式的转化和缺失质控_第21张图片](http://img.e-com-net.com/image/info8/139c87cc45344a5591eec53514817af5.jpg)
无论是SNP的缺失率,还是样本的缺失率,都是针对检出率进行的质控。如果一个群体中有些亚群对某些片段没有分型(片段缺失),这种情况下,对于样本进行质控或者样本和SNP同时质控,会将样本删除,而这些样本不是由于DNA质量差或者实验室的原因导致的缺失,而是由于这些样本本身的片段缺失导致的缺失,这种情况下,先对样本或者样本和SNP同时质控,会导致较大的错误。
为了避免这种情况,可以先对SNP的缺失率进行质控,这样由于某些亚群片段缺失导致的缺失,就会在SNP质控时将其删除,就不会影响后续的样本缺失质控的结果。
上面案例中,有一个样本,如果先进行SNP缺失质控再进行样本质控就不会被删除。而先进行样本质控或者同时质控,就会被删除。
看不懂的话可以这样理解:
SNP太多了,如果先进行样本的缺失SNP筛选,那么因为SNP的数量庞大,造成的SNP缺失的样本数量也会很大,导致被删掉的样本就会很多
相反,如果事先将一些缺失率很高的SNP去掉,就使得一些样本解决了"最后一公里的问题",使它们得以"存活下来".
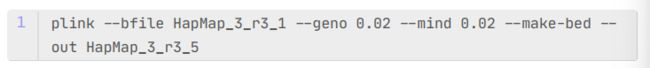
总结:如果单独过滤和合并过滤的差异较大的话,建议使用单独过滤的方法,即先过滤SNP,再过滤样本
两步合在一起的操作:
![[GWAS]plink数据格式的转化和缺失质控_第22张图片](http://img.e-com-net.com/image/info8/0adca68ddb0e468792a4ac404c7fc088.jpg)
和上面我们得到的结果就不太一样了
可以看出,合并质控和单独质控是有区别的,在这种情况之下,建议先进行SNP质控,再进行样本质控
(重要的事情说三遍!)
![[GWAS]plink数据格式的转化和缺失质控_第1张图片](http://img.e-com-net.com/image/info8/a1b88ce1c0d94493b9dfa538b915ec93.jpg)
![[GWAS]plink数据格式的转化和缺失质控_第3张图片](http://img.e-com-net.com/image/info8/8e27a360b8c24836a14b284b1f2a2980.jpg)
![[GWAS]plink数据格式的转化和缺失质控_第4张图片](http://img.e-com-net.com/image/info8/9138b0ee3bcc46a698794e897a74690a.png)
![[GWAS]plink数据格式的转化和缺失质控_第5张图片](http://img.e-com-net.com/image/info8/29a06ce8f43e4e1c9445926bb6d4e48e.jpg)
![[GWAS]plink数据格式的转化和缺失质控_第6张图片](http://img.e-com-net.com/image/info8/ffe5ccbb435249729b242e45cd9fc977.png)
![[GWAS]plink数据格式的转化和缺失质控_第7张图片](http://img.e-com-net.com/image/info8/c9216594eb2a460b8956e396db6f591f.png)
![[GWAS]plink数据格式的转化和缺失质控_第8张图片](http://img.e-com-net.com/image/info8/e125f6190fa048f28a3fc208b4b34f3e.jpg)
![[GWAS]plink数据格式的转化和缺失质控_第11张图片](http://img.e-com-net.com/image/info8/13ac52089246445cbc76e7a2698995d0.jpg)
![[GWAS]plink数据格式的转化和缺失质控_第12张图片](http://img.e-com-net.com/image/info8/d6ec4cb079c7434c8169c77d65742b93.jpg)