医学图像分割常用指标及代码(pytorch)
现在,医学图像分割有很多现成的工具包可以快速测量一些指标,比如python中的medpy库。但是,我们还是要学习一下滴!该文章列出了一些常用的指标,并解释了它的原理。
目录
- 一、医学图像分割常用指标及代码
-
- 1. Classic measures
-
- 1、What is T 1 、 T 0 、 P 1 、 P 0 T_1、T_0、P_1、P_0 T1、T0、P1、P0? [^1]
- 2、What is TP、TN、FP、TN?
- 3、Sensitivity (Recall, true positive rate)
- 4、Specificity(true negative rate)
- 5、Precision
- 2. Similarity
-
- 1、Dice Coefficient
- 2、Jaccard Coefficient
- 3. Distance
-
- 1、Hausdorff Distance (HD)
- 2、Average Surface Distance (ASD)
- 二、快速实现指标的工具和代码
- 参考资料
一、医学图像分割常用指标及代码
1. Classic measures
1、What is T 1 、 T 0 、 P 1 、 P 0 T_1、T_0、P_1、P_0 T1、T0、P1、P0? 1
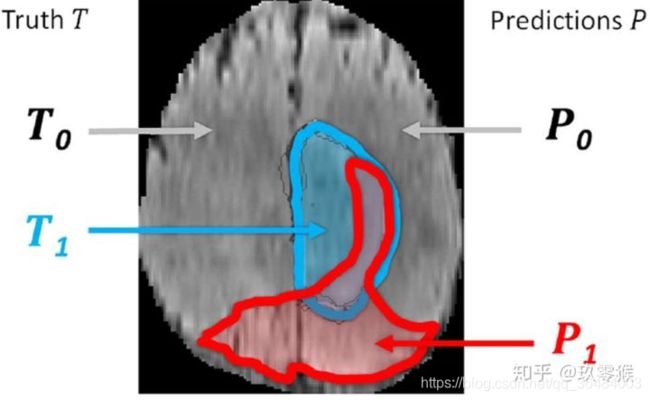
T 1 T_1 T1蓝色部分表示真实脑肿瘤区域(GroundTruth)
T 0 T_0 T0蓝色的其它部分为正常脑区域
P 1 P_1 P1红色部分表示预测的脑肿瘤区域
P 0 P_0 P0 红色的其它部分为预测的正常脑区域
2、What is TP、TN、FP、TN?
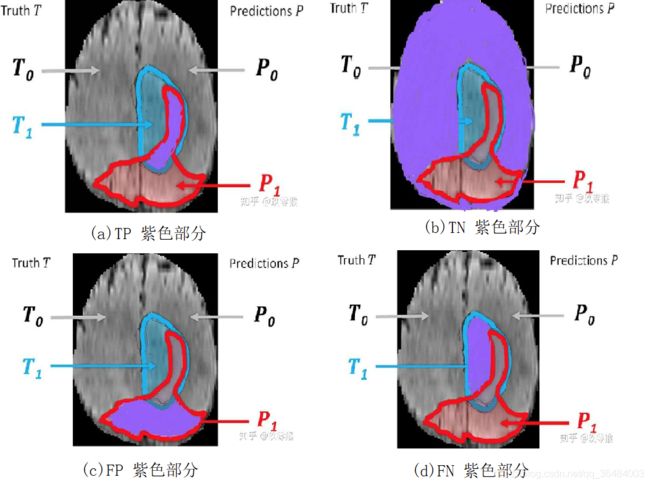
(a) TP:True Positive,被判定为正样本,事实上也是正样本 ,即蓝色与红色的交集
(b) TN:True Negative,被判定为负样本,事实上也是负样本,即红色与蓝色以外区域
(b) FP:False Positive,被判定为正样本,但事实上是负样本,即红色中除了蓝色部分
(d) FN:False Negative,被判定为负样本,但事实上是正样本,即蓝色中除了红色部分
3、Sensitivity (Recall, true positive rate)
真阳性率(true positive rate,TPR)也称为敏感度和召回率,它衡量的是真实背景中的阳性体素部分,即衡量的是分割实验中能分割感兴趣区域的能力。
R e c a l l = S e n s i t i v i t y = T P R = R g t ⋂ R p r e d R g t = T P T P + F N Recall = Sensitivity = TPR = \frac{R_{gt} \bigcap R_{pred}}{R_{gt}}= \frac{TP}{TP + FN} Recall=Sensitivity=TPR=RgtRgt⋂Rpred=TP+FNTP
R g t R_{gt} Rgt 是ground true 的分割结果, R p r e d R_{pred} Rpred是预测的分割结果。
pytorch代码:
def recall(predict, target): #Sensitivity, Recall, true positive rate都一样
if torch.is_tensor(predict):
predict = torch.sigmoid(predict).data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
predict = numpy.atleast_1d(predict.astype(numpy.bool))
target = numpy.atleast_1d(target.astype(numpy.bool))
tp = numpy.count_nonzero(predict & target)
fn = numpy.count_nonzero(~predict & target)
try:
recall = tp / float(tp + fn)
except ZeroDivisionError:
recall = 0.0
return recall
4、Specificity(true negative rate)
真阴性率(true negative rate,TNR)也称为特异性,它衡量的是地面真值分割中的负体素(background)部分,即其衡量的是分割实验中能正确判断不是感兴趣区域像素点的能力。
S p e c i f i c i t y = T N R = T N T N + F P Specificity = TNR = \frac{TN}{TN + FP} Specificity=TNR=TN+FPTN
pytorch代码:
def specificity(predict, target): #Specificity,true negative rate一样
if torch.is_tensor(predict):
predict = torch.sigmoid(predict).data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
predict = numpy.atleast_1d(predict.astype(numpy.bool))
target = numpy.atleast_1d(target.astype(numpy.bool))
tn = numpy.count_nonzero(~predict & ~target)
fp = numpy.count_nonzero(predict & ~target)
try:
specificity = tn / float(tn + fp)
except ZeroDivisionError:
specificity = 0.0
return specificity
5、Precision
精确度,也叫阳性预测值(PPV)。
P r e c i s i o n = P P V = R g t ⋂ R p r e d R p r e d = T P T P + F P Precision = PPV = \frac{R_{gt} \bigcap R_{pred}}{R_{pred}}= \frac{TP}{TP + FP} Precision=PPV=RpredRgt⋂Rpred=TP+FPTP
pytorch代码:
def precision(predict, target):
if torch.is_tensor(predict):
predict = torch.sigmoid(predict).data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
predict = numpy.atleast_1d(predict.astype(numpy.bool))
target = numpy.atleast_1d(target.astype(numpy.bool))
tp = numpy.count_nonzero(predict & target)
fp = numpy.count_nonzero(predict & ~target)
try:
precision = tp / float(tp + fp)
except ZeroDivisionError:
precision = 0.0
return precision
2. Similarity
1、Dice Coefficient
DICE(值域为[0,1]):也称为重叠指数,表示两个物体相交的面积占总面积的比值。
D I C E = 2 ∣ P 1 ⋂ T 1 ∣ ∣ P 1 ∣ + ∣ T 2 ∣ = 2 ∣ R p r e d ⋂ R g t ∣ ∣ R p r e d ∣ + ∣ R g t ∣ = 2 T P 2 T P + F P + F N DICE = \frac{2 \vert P_1 \bigcap T_1 \vert}{\vert P_1 \vert + \vert T_2 \vert}= \frac{2 \vert R_{pred}\bigcap R_{gt} \vert}{\vert R_{pred} \vert + \vert R_{gt} \vert} = \frac{2TP}{2TP + FP + FN} DICE=∣P1∣+∣T2∣2∣P1⋂T1∣=∣Rpred∣+∣Rgt∣2∣Rpred⋂Rgt∣=2TP+FP+FN2TP
pytorch 代码:
import torch
import numpy
from medpy import metric
predict = numpy.array([1, 2, 3, 4], dtype=float) #predict是预测结果
target = numpy.array([1, 0, 1, 2], dtype=float) #target是ground true
def dice(predict, target):
if torch.is_tensor(predict):
predict = torch.sigmoid(predict).data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
predict = numpy.atleast_1d(predict.astype(numpy.bool)) #转一维数组
target = numpy.atleast_1d(target.astype(numpy.bool))
intersection = numpy.count_nonzero(predict & target) #计算非零个数
size_i1 = numpy.count_nonzero(predict)
size_i2 = numpy.count_nonzero(target)
try:
dice = 2. * intersection / float(size_i1 + size_i2)
except ZeroDivisionError:
dice = 0.0
return dice
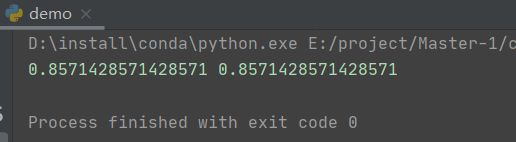
dice1 = dice(predict, target)
dice2 = metric.binary.dc(predict, target)
print(dice1, dice2)
2、Jaccard Coefficient
J A C = ∣ R g t ⋂ R p r e d ∣ ∣ R g t ⋃ R p r e d ∣ = T P T P + F P + F N JAC = \frac{\vert R_{gt} \bigcap R_{pred} \vert}{\vert R_{gt} \bigcup R_{pred} \vert} = \frac{TP}{TP + FP + FN} JAC=∣Rgt⋃Rpred∣∣Rgt⋂Rpred∣=TP+FP+FNTP
因此,DICE和JAC之间的关系是:
D I C E = 2 J A C 1 + J A C DICE = \frac {2JAC}{1 + JAC} DICE=1+JAC2JAC
pytorch代码:
from medpy import metric
import torch
import numpy
predict = numpy.array([[1, 2, 3, 4], [5, 6, 7, 8]], dtype=float)
target = numpy.array([[1, 6, 1, 0], [1, 2, 0, 0]], dtype=float)
def jac(predict, target):
if torch.is_tensor(predict):
predict = torch.sigmoid(predict).data.cpu().numpy()
if torch.is_tensor(target):
target = target.data.cpu().numpy()
predict = numpy.atleast_1d(predict.astype(numpy.bool))
target = numpy.atleast_1d(target.astype(numpy.bool))
intersection = numpy.count_nonzero(predict & target)
union = numpy.count_nonzero(predict | target)
jac = float(intersection) / float(union)
return jac
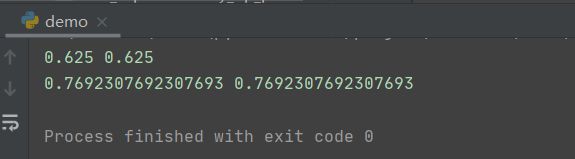
jac1 = jac(predict, target)
jac2 = metric.binary.jc(predict, target)
print(jac1, jac2)
#验证一下DICE和JAC的关系
dice1 = dice(predict, target)
dice2 = 2 * jac1 / (1 + jac1)
print(dice1, dice2)
3. Distance
这部分的代码过于复杂,就不一一实现了。哭唧唧,太难了~
1、Hausdorff Distance (HD)
Hausdorff_95就是是最后的值乘以95%,目的是为了消除离群值的一个非常小的子集的影响。
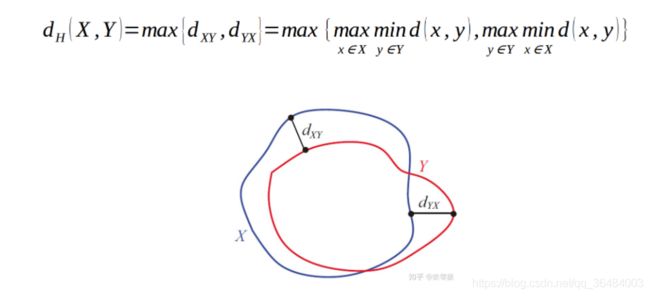
2、Average Surface Distance (ASD)
二、快速实现指标的工具和代码
医学图像处理的python库medpy:
官方文档链接
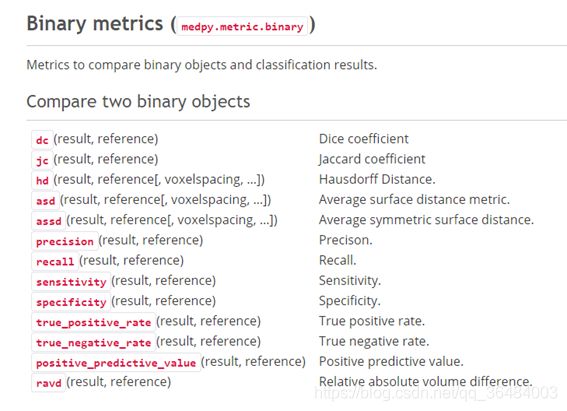
以上各个指标的pytorch代码都是参考medpy包中的代码,那我们如何快速实现这些指标呢?
答案如下(直接调包好爽啊哈哈哈哈):
from medpy import metric
def calculate_metric_percase(pred, gt):
dice = metric.binary.dc(pred, gt)
jc = metric.binary.jc(pred, gt)
hd = metric.binary.hd95(pred, gt)
asd = metric.binary.asd(pred, gt)
return dice, jc, hd, asd
参考资料
分割常用评价指标Dice、Hausdorff_95、IOU、PPV等
2.机器学习&图像分割——模型评价总结
3.CSDN中的LaTeX数学公式的基本操作
4.Metrics for evaluating 3D medical image segmentation: analysis, selection, and tool. ↩︎