paddle实践---基于Unet的右心室内膜分割
基于UNet的右心室分割
任务说明
利用UNet网络对RSCV数据集进行分割预测。
RSCV数据集简介:解压该数据集后,我们可以得到5个文件:一个训练集,两个测试集及其各自心内膜心外膜的坐标。
训练集中包含16个病人的目录,每个病人目录下有原始图像集合P0Xdicom,里面都是dcm文件;心内膜心外膜的坐标目录P0Xcontours-manual;以及人工标注的图像列表P0Xlist.txt
开发工具
Python版本:Python3.7
框架版本:Paddlepaddle2.2.2
相关库:pandas、os、tqdm、logging、numpy、PIL中的Image、
cv2、pydicom、matplotlib.pyplot、scipy.misc
设计内容
软件的设计框架逻辑图为:
数据处理
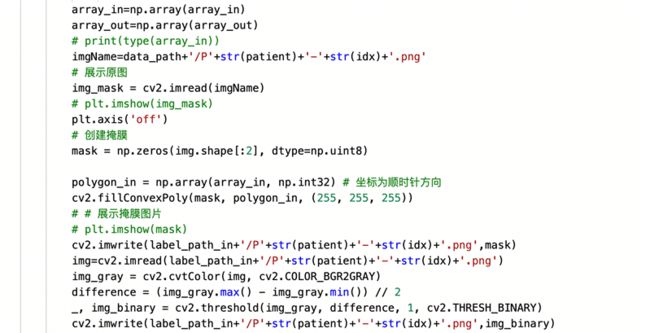
该部分我们首先将dcm文件转化成png文件,再把心内膜与心外膜的坐标列表txt文件转化成坐标数组,利用cv2的fillConvexPoly函数将轮廓转成掩膜图片并保存成png格式。
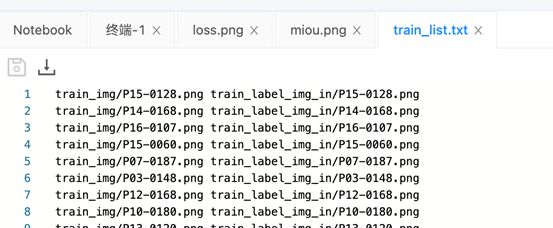
同时,因为PaddleSeg采用通用的文件列表方式组织训练集、验证集和测试集,所以我们在训练、评估、可视化过程前必须准备好相应的文件列表。文件列表的格式为原始图片路径 标注图片路径
构建网络
我们另外定义了一个BCEloss的函数,并调用Diceloss。我们将会通过训练和测试选择其一作为我们最后的损失函数。
- BCEloss:用于二分类的交叉熵损失函数
- Diceloss:集合相似度函数。
Dice系数,一种集合相似度度量函数,通常用于计算两个样本点的相似度(值范围为[0, 1])。计算公式为:![]()
|X⋂Y| - X 和 Y 之间的交集;|X| 和 |Y| 分别表示 X 和 Y 的元素个数. 其中,分子中的系数 2,是因为分母存在重复计算 X 和 Y 之间的共同元素的原因。
如果Dice系数越大,表明集合越相似,Loss越小;反之亦然。
训练部分
我们基于paddleSeg里面的train.py文件,重写了一遍train函数,使之更符合我们的训练要求。为了选择最好的模型,我们在调整了部分超参数。
损失函数:BCEloss和Diceloss
优化器:Adam和SGD
(其余固定的超参数可以在代码中观察到)
经过我们的测试,模型训练两千轮的效果基本达到最好,往后效果无太大变化。
在该环节中,为了能够观察到损失函数的变化趋势,我们另外将三种损失函数画在同一张图中,详见第7部分。
测试部分
在评估模型过程中,由于paddle自带的predict函数没有算miou的,所以我们重新写了一个计算miou函数。同样为了匹配predict函数的输入,我们需要将输入图像转化成图像列表。
为了更好更直观地看到模型的性能优劣,我们计算miou并画出miou变化曲线图。
我们发现,miou曲线非常跌宕,在部分图像中,miou值特别低。为了解释该现象,我们另外还绘制了箱型图,观察离群值。观察后我们发现,除了极端部分(及右心室太小了),大部分情况下都是正常预测的。
算法原理
Unet
Unet包括两部分:第一部分,特征提取;第二部分上采样部分。由于网络结构像U型,所以叫Unet网络。
特征提取部分,每经过一个池化层就一个尺度,包括原图尺度一共有5个尺度。
上采样部分,每上采样一次,就和特征提取部分对应的通道数相同尺度融合,但是融合之前要将其裁剪到合适的尺寸再拼接。可以看到,输入是572x572的,但是输出变成了388x388,这说明经过网络以后,输出的结果和原图不是完全对应的。
蓝色箭头代表3x3的卷积操作,红色箭头代表2x2的最大池化操作,需要注意的是,如果pooling之前featuremap的大小是奇数,有可能会损失一些信息。所以要选取合适的输入大小,因为2*2的max-pooling算子适用于偶数像素点的图像长宽。绿色箭头代表2x2的反卷积操作,操作会将featuremap的大小乘2。灰色箭头表示复制和剪切操作,可以发现,在同一层左边的最后一层要比右边的第一层要大一些,这就导致了,想要利用浅层的feature,就要进行一些剪切。输出的最后一层,使用了1x1的卷积层做了分类。最后输出了两层:前景和背景。
Unet++
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-2n4woWRO-1653408228309)(https://s2.loli.net/2022/05/25/tEzlYAudCkaTJXx.png)]
我们需要图像的浅层特征和深层特征,但是如果每次都是用UNet来训练得到这些数据的话,所耗费的时间过久。而UNet++解决了不同数据量、不同场景应用对网络深度的要求,直接省去对UNet网络深度修改的时间。
观察Unet++的网络架构,很明显看出来它是一个可以被剪枝的网络架构,这样子它就可以达到任务所需要的任意深度。
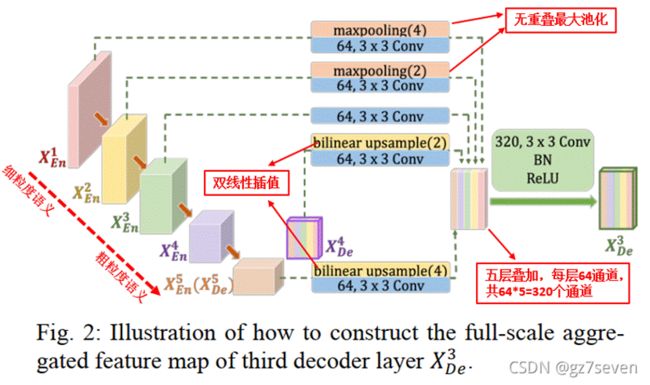
Unet3+
UNet3+有一个全尺寸连接模块,通过这个模块可构造出最后的特征图。它的全尺寸连接主要是来自三个部分:
\1. 较小尺度编码器,此部分保留了细节信息,即融合细粒度语义。编码器分别下采样4倍和2倍,以统一特征图的大小。
\2. 相同尺度编码器,此部分直接进行3*3卷积,64通道操作。
\3. 较大尺度解码器,此部分融合了粗粒度语义信息。分别进行2倍与4倍的上采样,同样是为了保证特征图大小的统一。
UNet3+相比与前两个网络,还有个全尺度监督。UNet++也有个全尺度监督,但是它是只对第一层进行深连接,即对全分辨率特征图进行深监督。而UNet3+是每个解码器对应一个侧输出,通过ground truth进行监督。为了实现深度监控,每个解码器的最后一层被送入一个普通的3 × 3卷积层,然后是一个双线性上采样和一个sigmoid函数。
加上双线性上采样可以将第2、3、4、5层扩展成全分辨率特征图,保证与第一层相同,这也是全尺寸深监督的关键操作。另外,它还可以最大限度保证上采样过程中边缘信息的完整性(医学图像边缘的不确定性决定要尽量保障边缘信息不丢失)。
另外,为了防止非器官图像的过度分割、提高模型的分割精度,UNet3+中添加一个额外的分类任务来预测输入图像是否有器官,从而实现更精准的分割。主要通过对不同尺度的解码器模块的结果进行一系列的操作来实现,包括Dropout,1x1的卷积,max pooling和 sigmoid函数,就可以得到一个分类结果(0表示没有器官,1表示有器官)。然后,将分类结果和分割结果相乘,这样就可以抑制对非器官图像的过度分割,从而提高模型的分割精度,实现精准分割。
程序实现的核心部分我觉得有两个:对原数据的处理及重写train函数。
原数据的处理主要就是如何将坐标转化成掩膜图片,然后就是匹配PeddleSeg的输入。
Train函数:
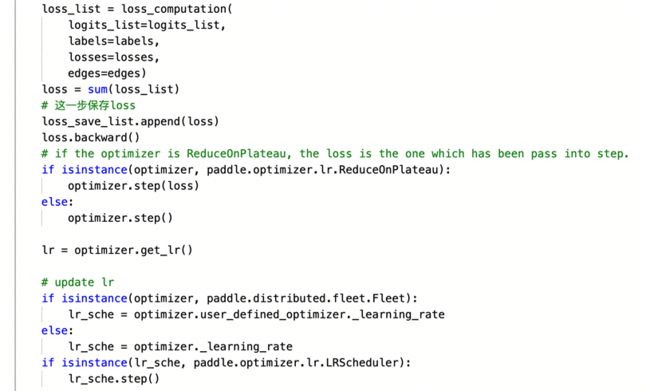
计算损失函数并且更新学习率。
每次训练完模型都要经过验证集,验证集的作用是筛出在验证集上miou最高,及效果最好的模型参数,并加以保存。
调试过程
在调试过程中我们主要做的工作是针对不同的超参数做修改。
得到以下的图表:(均以测试集的miou作为比较的指标)
- lr:2e-3,以UNet为基准模型,iters=1000
| BCEloss | Diceloss | |
|---|---|---|
| Adam | 0.673170 | 0.623592 |
| SGD | 0.013464(但是loss图像下降的很好看)(跑2000轮之后,到达0.67655, 2500/3000轮到0.71) | 0.634773(但是loss跌宕特别强,没有很明显的下降趋势) |
- lr=2e-3,iters=1000
| BCEloss | Diceloss | |||
|---|---|---|---|---|
| UNet | UNet++ | UNet | UNet++ | |
| Adam | 0.654321 | 0.657797 | 0.627849 | 0.648341 |
| SGD | 0.019578 | 0.092349 | 0.627037 | 0.663573 |
- l Loss function=BCEloss, optimizer=Adam,iters=1000
| lr\网络模型 | UNet | UNet++ |
|---|---|---|
| 2e-3 | 0.654321 | 0.657797 |
| 2e-4 | 0.695880 | 0.698098 |
结果分析
- UNet,UNet++,UNet3+在相同超参数下训练两千轮的loss变化图和miou变化图:
lr=2e-3,iters=2000,optimizer=Adam,loss function=BCEloss
| 网络模型 | UNet | UNet++ | UNet3+ |
|---|---|---|---|
| 测试集上的miou | 0.706586 | 0.724783 | 0.7067295 |
观察一下结果,发现大部分结果是正常的,除了少数离群值
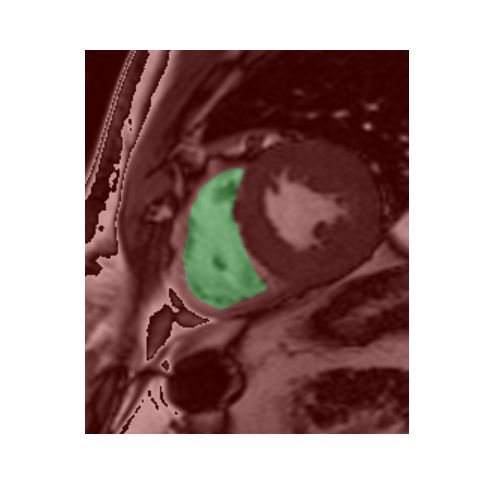
找一下识别效果不太好的图片,就是这些心内膜已经很小的图片,此时比较难识别,经常识别就是空的
而像这种面积很大的很好识别
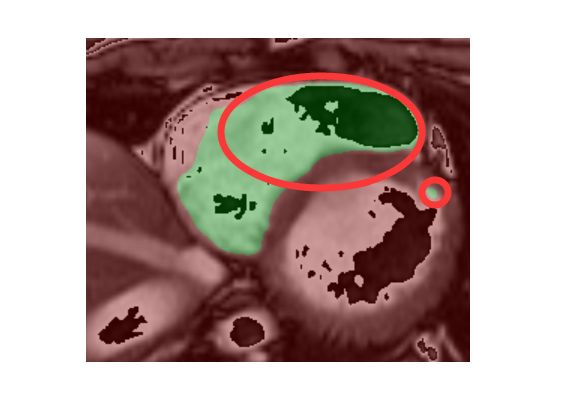
另外还有一个问题,有时识别会出现两块这种情况,这样或许能通过增大感受野去解决
源码已经公开于paddle平台
欢迎fork欢迎讨论
源码地址
最后再略微吐槽下,paddle的在线环境实在不太好用
感谢我的两位队友的支持!