RNA 21. SCI 文章中单基因富集分析
点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
88篇原创内容
公众号
今天就来剖析一下单基因到底怎么做?这个事情也算困扰好多老师的一个难题,总有有老师来问我,我就有这么一个基因,正在研究,但是我觉得实验的时间长,效率低,能否通过生信的方式找到该基因的一些特性,之后补充一些实验的验证,发一篇SCI文章,OK,今天就来唠叨一下单个基因发文章的生信思路!
有需要上述单基因生信分析内容的老师可以加我微信:
另外还有肿瘤克隆进化线上培训课程,有研究这个方向的老师,扫码预约记得来听啊!
前言
最近好多文章都是以单个基因为中心,干湿结合发一篇分值蛮高的 SCI 论文,这个同样也是目前的一个趋势,单个基因在 pan-cancer中的分布情况,比如功能,临床预测预后等,都是一个方向,精细分析一个基因将是个性化诊疗的一个方向。我们看到发在frotiers上的一篇文章,就是单基因生信分析的这个思路。
基本思路
单基因富集分析并不是说拿单个基因来进行富集分析,单个基因怎么能进行富集分析呢?一个基因根本没法进行统计检验。
其实,这里说的单基因并不是拿单个基因来富集,而是基于单个基因来进行富集分析,这个“基于”,就是以单个基因为基础,向外扩展,抓取与其相关的基因,然后用这些相关的基因来进行功能富集
所以,要理解这个单基因富集分析的意思,这样一说就已经很明了了。针对单个基因我们可以做什么?
主要有两种做法:
1. 定性分组
我们可以根据给定基因的表达值对样本进行分组,然后识别在两组样本之间差异表达的基因,最后用这些差异表达基因来进行功能富集。
2. 定量相关
通过计算其他基因与目标基因表达之间的相关性,将具有显著相关的基因作为一个集合,也可以进行富集分析。
3. 定性分组与定量相关结合
简单说就是通过给定基因的表达对样本分组,然后做差异分析,同时做该基因与其他基因之间的关系,对差异基因进一步筛选,获得最后的基因集进行后续的功能上的分析等。
实操演练
1. 软件安装
我们选安装几个软件包,如limma,做基因表达差异分析,TCGAbiolinks自带函数TCGAanalyze_DEA 可以满足分析,软件安装及加载, 如下:
if (!require(TCGAbiolinks)) BiocManager::install("TCGAbiolinks")
if (!require(limma)) BiocManager::install("limma")
if (!require(clusterProfiler)) BiocManager::install("clusterProfiler")
if (!require(org.Hs.eg.db)) BiocManager::install("org.Hs.eg.db")
if (!require(enrichplot)) BiocManager::install("enrichplot")
if (!require(ggplot2)) install.packages("ggplot2")
if (!require(future.apply)) install.packages("future.apply")
if (!require(dbplyr)) install.packages("dbplyr")
if (!require(tidygraph)) install.packages("tidygraph")
if (!require(tibble)) install.packages("tibble")
library(future.apply)
library(TCGAbiolinks)
library(limma)
library(ggplot2)
library(clusterProfiler)
library(org.Hs.eg.db)
library(enrichplot)
library(dbplyr)
library(tidygraph)
library(tibble)
2. 数据读取
TCGA 数据库下载数据,结直肠癌包括结肠癌和直肠癌两部分的合并,因此我们现在两个数据集 TCGA-COAD 和 TCGA-READ 的基因 count 数据,在进行转换为 TPM/RPKM,进行后续的分析。
# 读数据
dataFilt <- read.table("TCGA-CRC.exp.txt", header = T, check.names = F, sep = "\t",
row.names = 1)
dataFilt[1:3, 1:3]
## TCGA-NH-A8F7-06 TCGA-3L-AA1B-01 TCGA-4N-A93T-01
## TSPAN6 276.557009 140.610717 130.749413
## TNMD 1.320335 1.251314 3.444375
## DPM1 349.186948 149.857030 180.484569
3. 定性分组
我们以 PD-L2 基因为例,我们发现PD-L1的基因在结直肠癌中的 symbol 为 PDCD1LG2,以该基因表达值的中位值来对样本进行分组,并做差异分析,如下:
### 定性分组
gene <- "PDCD1LG2"
gene.exp <- dataFilt[gene, ]
gene.exp <- as.numeric(gene.exp)
label <- ifelse(gene.exp < median(gene.exp), "Low", "High")
group.low <- dataFilt[, label == "Low"]
group.high <- dataFilt[, label == "High"]
DEGs <- TCGAanalyze_DEA(mat1 = group.low, mat2 = group.high, metadata = FALSE, pipeline = "limma",
Cond1type = "Low", Cond2type = "High", fdr.cut = 0.001, logFC.cut = 2, )
dim(DEGs)
## [1] 6239 6
head(DEGs)
## logFC AveExpr t P.Value adj.P.Val B
## CD86 9.052203 7.907559 21.44244 1.039311e-78 3.578243e-74 168.4738
## PDCD1LG2 4.519871 3.333475 20.84369 2.358380e-75 4.059834e-71 160.8064
## FPR3 25.432977 21.702873 20.77414 5.768679e-75 6.620328e-71 159.9189
## CD53 55.411014 51.385913 20.57422 7.516402e-74 6.469555e-70 157.3715
## C3AR1 18.728915 16.726681 20.52239 1.460987e-73 1.006007e-69 156.7120
## TMEM273 4.491173 4.943034 20.44457 3.959893e-73 2.272253e-69 155.7226
4. 定量相关
我们对其他基因与 PDCD1LG2 基因进行相关性检验,由于基因较多,我们使用并行的方式来计算。
####### 定量相关
library(future.apply)
batch_cor <- function(exp, gene) {
y = as.numeric(exp[gene, ])
gene_list = rownames(exp)
gene_list = gene_list[rownames(exp) != gene]
do.call(rbind, future_lapply(gene_list, function(x) {
ct <- cor.test(as.numeric(exp[x, ]), y, type = "spearman")
data.frame(key = gene, gene = x, cor = ct$estimate, p.value = ct$p.value)
}))
}
plan(multiprocess)
system.time(res.cor <- batch_cor(dataFilt, gene))
## 用户 系统 流逝
## 2.39 2.32 60.87
head(res.cor)
## key gene cor p.value
## cor PDCD1LG2 TSPAN6 -0.23551713 2.977089e-10
## cor1 PDCD1LG2 TNMD -0.05425580 1.521704e-01
## cor2 PDCD1LG2 DPM1 -0.16877363 7.354801e-06
## cor3 PDCD1LG2 SCYL3 0.12294976 1.134606e-03
## cor4 PDCD1LG2 C1orf112 0.01347008 7.223997e-01
## cor5 PDCD1LG2 FGR 0.72313837 5.855290e-114
5. 定性分组与定量相关结合
由于差异基因的个数过多,我们可以通过定量相关性,选择R2>0.6的高度相关的基因做后续的分析,如下:
resCor <- na.omit(res.cor[(res.cor$cor > 0.6 & res.cor$p.value < 0.001), ])
dim(resCor)
## [1] 332 4
head(resCor)
## key gene cor p.value
## cor5 PDCD1LG2 FGR 0.7231384 5.855290e-114
## cor239 PDCD1LG2 TRAF3IP3 0.6076993 1.082598e-71
## cor266 PDCD1LG2 CD4 0.6945782 1.249530e-101
## cor268 PDCD1LG2 BTK 0.6336259 1.260842e-79
## cor306 PDCD1LG2 TYROBP 0.6043151 1.038850e-70
## cor378 PDCD1LG2 SLC11A1 0.6605481 1.042615e-88
DEGfilter <- unique(resCor$gene)
6. 富集分析(GO/KEGG)
对格式化识别出的差异基因进行富集分析,包括GO和KEGG,如下:
GO富集分析
library(clusterProfiler)
library(org.Hs.eg.db)
library(enrichplot)
eg <- bitr(DEGfilter, fromType = "SYMBOL", toType = c("ENTREZID", "ENSEMBL", "SYMBOL"),
OrgDb = "org.Hs.eg.db")
dim(eg)
## [1] 426 3
dim(unique(eg))
## [1] 426 3
go <- enrichGO(eg$SYMBOL, OrgDb = org.Hs.eg.db, ont = "ALL", keyType = "SYMBOL",
pAdjustMethod = "BH", pvalueCutoff = 0.01, qvalueCutoff = 0.05)
dim(go)
## [1] 602 10
go@result[1:5, 1:5]
## ONTOLOGY ID Description
## GO:0001819 BP GO:0001819 positive regulation of cytokine production
## GO:0042110 BP GO:0042110 T cell activation
## GO:0002274 BP GO:0002274 myeloid leukocyte activation
## GO:0007159 BP GO:0007159 leukocyte cell-cell adhesion
## GO:1903037 BP GO:1903037 regulation of leukocyte cell-cell adhesion
## GeneRatio BgRatio
## GO:0001819 53/291 467/18722
## GO:0042110 53/291 487/18722
## GO:0002274 38/291 223/18722
## GO:0007159 45/291 371/18722
## GO:1903037 42/291 336/18722
dotplot(go, showCategory = 10, font.size = 10, split = "ONTOLOGY", color = "pvalue") +
facet_grid(ONTOLOGY ~ ., scale = "free") + scale_y_discrete(labels = function(x) stringr::str_wrap(x,
width = 60))
KEGG富集分析
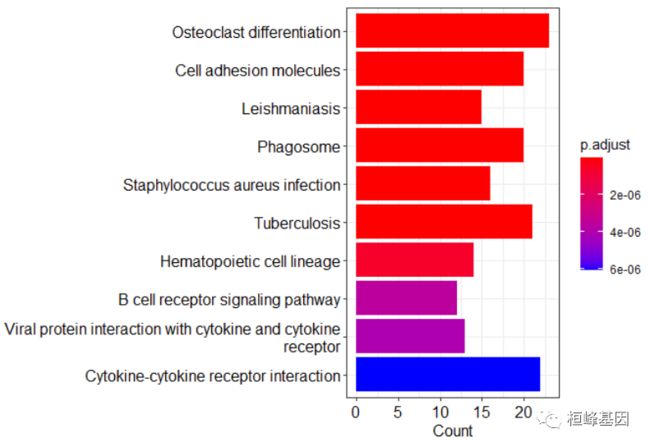
绘制条形图,如下:
### KEGG
kegg <- enrichKEGG(eg$ENTREZID, organism = "hsa", keyType = "kegg", pvalueCutoff = 0.05,
pAdjustMethod = "BH", minGSSize = 1, maxGSSize = 500, qvalueCutoff = 0.05, use_internal_data = FALSE)
head(kegg)
## ID Description GeneRatio BgRatio
## hsa04380 hsa04380 Osteoclast differentiation 23/176 128/8145
## hsa04514 hsa04514 Cell adhesion molecules 20/176 149/8145
## hsa05140 hsa05140 Leishmaniasis 15/176 77/8145
## hsa04145 hsa04145 Phagosome 20/176 152/8145
## hsa05150 hsa05150 Staphylococcus aureus infection 16/176 96/8145
## hsa05152 hsa05152 Tuberculosis 21/176 180/8145
## pvalue p.adjust qvalue
## hsa04380 2.303004e-15 4.214498e-13 3.078753e-13
## hsa04514 4.727064e-11 3.132132e-09 2.288069e-09
## hsa05140 6.320320e-11 3.132132e-09 2.288069e-09
## hsa04145 6.846190e-11 3.132132e-09 2.288069e-09
## hsa05150 1.679971e-10 6.148694e-09 4.491712e-09
## hsa05152 2.223882e-10 6.782840e-09 4.954965e-09
## geneID
## hsa04380 695/7305/3937/6688/2213/1540/10326/10859/11024/3554/4688/10288/2212/2209/2215/1436/11006/353514/2214/11025/102725035/11027/79168
## hsa04514 920/1462/5788/3383/942/3676/29126/941/3685/10666/3689/7412/29851/3684/940/201633/6402/3117/3122/3109
## hsa05140 3717/3676/4688/7097/2212/2209/3689/2215/1536/3684/3117/1378/2214/3122/3109
## hsa04145 4481/2213/4688/1514/7097/3685/2212/2209/3689/2215/1536/3684/64581/4973/10333/3117/2214/3122/3109/4360
## hsa05150 2213/3383/2212/2209/3689/2215/5724/3684/2357/719/2359/3117/728/2214/3122/3109
## hsa05152 2213/3717/3587/7097/3687/2212/2209/2207/3689/2215/26253/3684/64581/7096/10333/3117/1378/2214/3122/3109/4360
## Count
## hsa04380 23
## hsa04514 20
## hsa05140 15
## hsa04145 20
## hsa05150 16
## hsa05152 21
dim(kegg)
## [1] 45 9
barplot(kegg, showCategory = 10) + scale_y_discrete(labels = function(x) stringr::str_wrap(x,
width = 60))
7. 基因集富集分析(GESA)
数据处理
在我们做基因集富集分析时,首先需要对数据处理,由于GESA的数据输入要求是对表达数据降序排列后的向量,因此我们转换格式,如下:
geneList order ranked geneList
library(dbplyr)
library(tidygraph)
library(tibble)
gene_info <- DEGs[DEGfilter, ] %>%
rownames_to_column(var = "SYMBOL") %>%
inner_join(., eg[, 1:2], by = "SYMBOL") %>%
# 必须降序
arrange(desc(logFC))
# 构造输入数据格式
gene_info <- unique(gene_info)
geneList <- gene_info$logFC
names(geneList) <- as.character(gene_info$ENTREZID)
head(geneList)
## 3122 5552 10631 7805 7305 6362
## 1354.95132 187.13722 152.89445 136.33062 107.90073 91.09393
差异基因分析
_1000_购买
GSEA富集分析
我们需要gmt文件,http://www.gsea-msigdb.org/gsea/downloads.jsp 路径下载,选择合适的,我们这里是结直肠癌中免疫基因集做差异比较分析,所以我们需要找到免疫基因集,选择 C7: immunologic signature gene sets 即 c7.all.v7.5.1.entrez.gmt 文件,然后进行 GSEA 富集分析,由于基因集富集的结果并不理想,所以这里就没有绘图,我们看到根据阈值并没有筛选出来通路,如下:
library(stats)
kegg_gmt <- read.gmt("c7.all.v7.5.1.entrez.gmt") #读gmt文件
gsea <- GSEA(geneList, TERM2GENE = kegg_gmt, pvalueCutoff = 0.02) #GSEA分析
head(gsea)
## [1] ID Description setSize enrichmentScore
## [5] NES pvalue p.adjust qvalues
## <0 行> (或0-长度的row.names)
GO富集分析
由于基因集GO富集的结果我们通过参数pvalueCutoff = 0.2进行筛选,并绘制气泡图,如下:
gseGO <- gseGO(
geneList, #gene_fc
ont = "ALL",# "BP"、"MF"和"CC"或"ALL"
OrgDb = org.Hs.eg.db,#人类注释基因
keyType = "ENTREZID",
pvalueCutoff = 0.2,
pAdjustMethod = "BH",#p值校正方法
)
sortGO<-gseGO[order(gseGO$enrichmentScore, decreasing = T),]#按照enrichment score从高到低排序
head(sortGO[,1:4])
## ONTOLOGY ID
## GO:0005575 CC GO:0005575
## GO:0110165 CC GO:0110165
## GO:0046634 BP GO:0046634
## GO:0045582 BP GO:0045582
## GO:0045580 BP GO:0045580
## GO:0045621 BP GO:0045621
## Description setSize
## GO:0005575 cellular_component 249
## GO:0110165 cellular anatomical entity 249
## GO:0046634 regulation of alpha-beta T cell activation 11
## GO:0045582 positive regulation of T cell differentiation 11
## GO:0045580 regulation of T cell differentiation 12
## GO:0045621 positive regulation of lymphocyte differentiation 12
绘制GSEA富集图
go <- gseGO$ID[1:4]
gseaplot2(gseGO, go, pvalue_table = TRUE, color = colorspace::rainbow_hcl(4), base_size = 10)
KEGG富集分析
由于基因集KEGG富集的结果我们通过参数pvalueCutoff = 0.2进行筛选,并绘制气泡图,如下:
gseKEGG <- gseKEGG(geneList, organism = "hsa", keyType = "kegg", exponent = 1, minGSSize = 5,
maxGSSize = 500, eps = 1e-10, pvalueCutoff = 0.2, pAdjustMethod = "BH", verbose = TRUE,
use_internal_data = FALSE, seed = FALSE, by = "fgsea")
sortKEGG <- gseKEGG[order(gseKEGG$enrichmentScore, decreasing = T), ] #按照enrichment score从高到低排序
head(sortKEGG)
## ID Description setSize
## hsa05321 hsa05321 Inflammatory bowel disease 5
## hsa04672 hsa04672 Intestinal immune network for IgA production 7
## hsa04658 hsa04658 Th1 and Th2 cell differentiation 6
## hsa05169 hsa05169 Epstein-Barr virus infection 7
## hsa05145 hsa05145 Toxoplasmosis 10
## hsa05168 hsa05168 Herpes simplex virus 1 infection 7
## enrichmentScore NES pvalue p.adjust qvalues rank
## hsa05321 0.9471354 1.610002 0.02054321 0.18468 0.1458 1
## hsa04672 0.9371687 1.677422 0.01904623 0.18468 0.1458 1
## hsa04658 0.9262431 1.627275 0.02269057 0.18468 0.1458 1
## hsa05169 0.9211088 1.648676 0.02140588 0.18468 0.1458 1
## hsa05145 0.9194856 1.684449 0.02997859 0.18468 0.1458 1
## hsa05168 0.9189549 1.644821 0.02140588 0.18468 0.1458 1
## leading_edge core_enrichment
## hsa05321 tags=20%, list=0%, signal=20% 3122
## hsa04672 tags=14%, list=0%, signal=15% 3122
## hsa04658 tags=17%, list=0%, signal=17% 3122
## hsa05169 tags=14%, list=0%, signal=15% 3122
## hsa05145 tags=10%, list=0%, signal=10% 3122
## hsa05168 tags=14%, list=0%, signal=15% 3122
绘制GSEA富集图
paths <- gseKEGG$ID[1:4]
gseaplot2(gseKEGG, paths, pvalue_table = FALSE, color = colorspace::rainbow_hcl(4),
base_size = 20)
结果解读
我们看到通过一般的GO 和 KEGG 注释就可以得到我们需要的结果,我们通过PD-L2表达量将样本分为两组,发现相关的基因,并进行功能上的分析,可以找到共性,这样筛选之后,我们就可以做后续的生存分析,构建临床预后模型等一系列与疾病,用药相关的分析了!
References:
-
Cui X, Zhang X, Liu M, et al. A pan-cancer analysis of the oncogenic role of staphylococcal nuclease domain-containing protein 1 (SND1) in human tumors. Genomics. 2020;112(6):3958-3967. doi:10.1016/j.ygeno.2020.06.044
-
Wang S, Chen S, Ying Y, et al. Comprehensive Analysis of Ferroptosis Regulators With Regard to PD-L1 and Immune Infiltration in Clear Cell Renal Cell Carcinoma. Front Cell Dev Biol. 2021;9:676142. Published 2021 Jul 5. doi:10.3389/fcell.2021.676142