Tangram Tutorial 2:配合 squidy 使用 Tangram
「写在前面」
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:使用 Tangram 进行空间解析单细胞转录组的深度学习和比对 教程篇:
1.Tangram Tutorial 1:映射小鼠皮层 MOp 的 snRNAseq 数据到 Slide-seq2 数据
目录
- 1 安装
- 2 导入数据
- 3 预处理
- 4 寻找对齐
- 5 细胞类型映射
- 6 通过对齐的单细胞创建新的空间组数据
- 7 通过对齐进行反卷积
官网教程:
https://tangram-sc.readthedocs.io/en/latest/tutorial_sq_link.html
1 安装
Tangram 基于 pytorch、scanpy 和(可选但强烈推荐)squidpy - 本教程旨在与 squidy 配合使用。在与 squidpy 整合之前,您还可以查看本教程。
- 要在本地运行笔记本,请使用我们的 YAML 文件创建一个 conda 环境:
conda env create -f tangram_environment.yml。 - 该笔记本基于 squidpy v1.1.0。
import scanpy as sc
import squidpy as sq
import numpy as np
import pandas as pd
from anndata import AnnData
import pathlib
import matplotlib.pyplot as plt
import matplotlib as mpl
import skimage
import seaborn as sns
import tangram as tg
import anndata as ad
sc.logging.print_header()
print(f"squidpy=={sq.__version__}")
%load_ext autoreload
%autoreload 2
%matplotlib inline
2 导入数据
官网提供的导入数据函数可能会报错,推测是因为网络的问题,因此需要自己手动下载导入,下面提供了各个函数对应的数据下载链接:
# 数据下载地址:
# sq.datasets.visium_fluo_adata_crop()
https://figshare.com/articles/dataset/Brain_Coronal_Fluorescent_Adata_Crop/13604168/1
# sq.datasets.visium_fluo_image_crop()
https://figshare.com/articles/dataset/Brain_Coronal_Fluorescent_Image_Crop/13604165/1
# sq.datasets.sc_mouse_cortex()
https://figshare.com/articles/dataset/sc_mouse_cortex/13677259/1
导入数据:
adata_st = ad.read_h5ad('data/visium_fluo_crop.h5ad')
adata_st = adata_st[
adata_st.obs.cluster.isin([f"Cortex_{i}" for i in np.arange(1, 5)])
].copy()
img = sq.im.ImageContainer('data/visium_fluo_crop.tiff')
adata_sc = ad.read_h5ad('data/sc_mouse_cortex.h5ad')
我们对小鼠大脑进行子集化,使其仅包含大脑皮层 clusters。预处理的单细胞数据集取自 tasic2018shared 并使用标准 scanpy 函数进行预处理。
让我们可视化空间数据集和单细胞数据集。
adata_st.shape
## (324, 16562)
adata_st.obs
adata_sc.shape
## (21697, 36826)
adata_sc.obs
fig, axs = plt.subplots(1, 2, figsize=(20, 5))
sc.pl.spatial(
adata_st, color="cluster", alpha=0.7, frameon=False, show=False, ax=axs[0]
)
sc.pl.umap(
adata_sc, color="cell_subclass", size=10, frameon=False, show=False, ax=axs[1]
)
plt.tight_layout()

Tangram 通过查看用户指定的基因子集(称为训练基因)来学习单细胞数据的空间对齐。训练基因需要承载有趣的信号并进行高质量的测量。通常,我们选择的训练基因是 100-1000 个差异表达基因,跨细胞类型分层。有时,我们还使用整个转录组,或使用不同的训练基因组执行不同的映射,以查看结果变化有多大。
Tangram 使用基于余弦相似性的自定义损失函数在空间上拟合 scRNA-seq profiles。该方法总结如下:
3 预处理
对于本例,我们使用 1401 个 marker genes 作为训练基因。
sc.tl.rank_genes_groups(adata_sc, groupby="cell_subclass", use_raw=False)
markers_df = pd.DataFrame(adata_sc.uns["rank_genes_groups"]["names"]).iloc[0:100, :]
markers = list(np.unique(markers_df.melt().value.values))
len(markers)
## 1401
我们使用 pp_adatas 准备数据,它执行以下操作:
- 通过
genes参数从用户获取基因列表。这些基因用作训练基因。 - 在每个 AnnData 的
uns字典中的training_genes字段下注释训练基因。 - 确保数据集中的基因顺序一致(Tangram 要求每个矩阵中的 j-th 列对应于相同的基因)。
- 如果某个基因的计数在其中一个数据集中全部为零,则该基因将从训练基因中删除。
- 如果两个数据集中都不存在基因,则从训练基因中删除该基因。
- 在 pp_adatas 函数中,基因名称被转换为小写,以消除不一致的大写。如果不需要,可以设置参数
gene_to_lowercase=False
tg.pp_adatas(adata_sc, adata_st, genes=markers)
两个数据集包含最初提供的 1401 个 training genes 中的 1280 个,因为一些训练基因已经被删除。
4 寻找对齐
为了找到 scRNA-seq profiles 的最佳空间比对,我们使用 map_cells_To_space 函数:
- 函数按照
num_epochs的指定进行迭代映射。我们通常会在分数趋于平稳后中断映射。 - 该分数衡量了映射细胞的基因表达与训练基因的空间数据之间的相似性。
- 默认映射模式为
mode='cells',建议在 GPU 上运行。 - 或者,可以指定
mode='clusters',它对属于同一 cluster 的单个细胞进行平均(通过cluster_label传递注释)。当 scRNAseq 和空间数据来自不同样本时,这更快,是我们的选择。 - 如果您希望使用 GPU 运行 Tangram,请设置
device=cuda:0,否则使用设置device=cpu。 -
density_prior指定每个空间体素内的细胞密度。如果空间体素处于单细胞分辨率(即 MERFISH),请使用uniform。默认值rna_count_based假定细胞密度与 RNA 分子的数量成正比。
ad_map = tg.map_cells_to_space(
adata_sc, adata_st,
mode="cells",
# mode="clusters",
# cluster_label='cell_subclass', # .obs field w cell types
density_prior='rna_count_based',
num_epochs=500,
# device="cuda:0",
device='cpu',
)
映射结果存储在返回的 AnnData 结构中,保存为 ad_map,结构如下:
- cell-by-spot
X包含 celli位于 spotj的概率。 -
obsdataframe 包含单个 cell 的 metadata。 -
vardataframe 包含空间数据的 metadata。 -
unsdictionary 包含一个 dataframe,其中包含有关训练基因的各种信息(保存为train_genes_df)。
5 细胞类型映射
为了可视化空间中的细胞类型,我们调用 project_cell_annotation 将 annotation 从映射传输到空间。然后我们可以调用 plot_cell_annotation 来可视化它。您可以设置 perc 参数来设置 colormap 的范围,这将有助于删除异常值。
ad_map
## AnnData object with n_obs × n_vars = 21697 × 324
## obs: 'sample_name', 'organism', 'donor_sex', 'cell_class', 'cell_subclass', 'cell_cluster', 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes', 'total_counts_mt', 'log1p_total_counts_mt', 'pct_counts_mt', 'n_counts'
## var: 'in_tissue', 'array_row', 'array_col', 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes', 'total_counts_MT', 'log1p_total_counts_MT', 'pct_counts_MT', 'n_counts', 'leiden', 'cluster', 'uniform_density', 'rna_count_based_density'
## uns: 'train_genes_df', 'training_history'
tg.project_cell_annotations(ad_map, adata_st, annotation="cell_subclass")
annotation_list = list(pd.unique(adata_sc.obs['cell_subclass']))
tg.plot_cell_annotation_sc(adata_st, annotation_list, perc=0.02)
了解映射是否成功的第一个方法是寻找已知的细胞类型模式。为了获得更深入的了解,我们可以使用帮助程序 plot_training_scores,它为我们提供了四个面板:
tg.plot_training_scores(ad_map, bins=20, alpha=.5)

- 第一个面板是每个训练基因的相似性分数的直方图。
- 在第二张图中,每个点都是一个训练基因,我们可以观察每个基因的训练得分(y 轴)和 scRNA-seq 数据(x 轴)中的稀疏性。
- 第三个面板与第二个面板类似,但包含空间数据的基因稀疏性。空间数据通常比单细胞数据更加稀疏,这种差异通常是造成低质量地图的原因。
- 在最后一个面板中,我们将训练分数显示为数据集之间稀疏度差异的函数。对于具有相当稀疏性的基因,映射的基因表达与空间数据中的基因表达非常相似。然而,如果某个基因在一个数据集中(通常是空间数据)非常稀疏,但在其他数据集中则不然,则映射分数会较低。出现这种情况是因为 Tangram 无法正确匹配基因模式,因为数据集之间的丢失数量不一致。
尽管上图为我们提供了单基因水平的分数摘要,但我们需要知道哪些基因的分数较低。这些信息存储在 dataframe .uns['train_genes_df'] 中;这是用于构建上面四个图的 dataframe。
ad_map.uns['train_genes_df']
6 通过对齐的单细胞创建新的空间组数据
如果映射模式是'cells',我们现在可以使用映射的单个细胞生成“新空间数据”:这是通过 project_genes 完成的。该函数接受映射 (adata_map) 和相应的单细胞数据 (adata_sc) 作为输入。结果是逐个基因的体素 AnnData,形式上类似于 adata_st,但包含来自映射的单细胞数据而不是 Visium 的基因表达。对于下游分析,我们总是将 adata_st 替换为相应的 ad_ge。
ad_ge = tg.project_genes(adata_map=ad_map, adata_sc=adata_sc)
ad_ge
## AnnData object with n_obs × n_vars = 324 × 36826
## obs: 'in_tissue', 'array_row', 'array_col', 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes', 'total_counts_MT', 'log1p_total_counts_MT', 'pct_counts_MT', 'n_counts', 'leiden', 'cluster', 'uniform_density', 'rna_count_based_density'
## var: 'mt', 'n_cells_by_counts', 'mean_counts', 'log1p_mean_counts', 'pct_dropout_by_counts', 'total_counts', 'log1p_total_counts', 'n_cells', 'highly_variable', 'highly_variable_rank', 'means', 'variances', 'variances_norm', 'sparsity', 'is_training'
## uns: 'cell_class_colors', 'cell_subclass_colors', 'hvg', 'neighbors', 'pca', 'umap', 'rank_genes_groups', 'training_genes', 'overlap_genes'
让我们选择一些得分较低的训练基因,来尝试理解其中的原因。
genes = ['rragb', 'trim17', 'eno1b']
ad_map.uns['train_genes_df'].loc[genes]
## train_score sparsity_sc sparsity_sp sparsity_diff
## rragb 0.358792 0.079919 0.867284 0.787365
## trim17 0.203691 0.069641 0.959877 0.890236
## eno1b 0.343277 0.022492 0.885802 0.863311
为了可视化基因模式,我们使用辅助程序 plot_genes。该函数接受两个逐基因体素的 AnnData:实际空间数据 (adata_measured) 和 Tangram 空间预测 (adata_predicted)。该函数从基因 genes 上的两个空间 AnnData 返回基因表达图。
tg.plot_genes_sc(genes, adata_measured=adata_st, adata_predicted=ad_ge, perc=0.02)
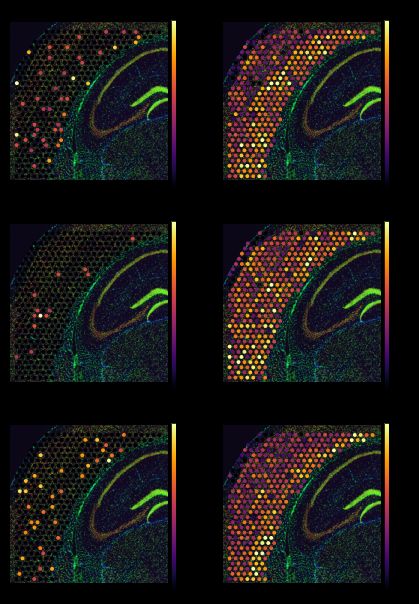
上图解释了训练分数低的情况。检测到的一些基因具有非常不同的稀疏程度 - 通常它们在空间数据中比在 scRNAseq 中稀疏得多。这是因为像 Visium 这样的技术更容易出现技术中断。因此,由于基线信号缺失,Tangram 无法找到这些基因的良好空间对齐。然而,只要大多数训练基因得到高质量的测量,我们就可以信任映射并使用 Tangram 预测来纠正基因表达。这是一种与概率模型完全不同的前提的 imputation 方法。
另一个应用是通过检查在空间数据中未检测到但在单细胞数据中检测到的基因来发现。它们在使用 pp_adatas 函数训练之前被删除,但 Tangram 可以预测它们的表达。
genes=['loc102633833', 'gm5700', 'gm8292']
tg.plot_genes_sc(genes, adata_measured=adata_st, adata_predicted=ad_ge, perc=0.02)

- 到目前为止,我们只检查了用于对齐数据的基因(训练基因),但映射的单细胞数据
ad_ge包含整个转录组。其中包括超过 35k 个测试基因。
(ad_ge.var.is_training == False).sum()
## 35546
我们也可以使用 plot_genes 来检查测试基因的基因表达。检查测试转录组对于验证映射至关重要。同时,我们需要注意,由于技术的下降,某些预测可能与空间数据不一致。
计算所有基因的相似度分数很方便,可以通过 compare_spatial_geneexp 来完成。该函数接受两个空间 AnnData(即逐个基因的体素),并返回一个包含所有基因相似度分数的 dataframe。训练基因由布尔字段 is_training 标记。如果我们还将单细胞 AnnData 传递给 Compare_spatial_geneexp 函数,如下所示,则会返回带有附加稀疏列的 dataframe - sparsity_sc(单细胞数据稀疏度)和 sparsity_diff(空间数据稀疏度 - 单细胞数据稀疏度)。如果我们想稍后使用从 compare_spatial_geneexp 函数返回的 datafrme 调用 plot_test_scores 函数,这是必需的。
df_all_genes = tg.compare_spatial_geneexp(ad_ge, adata_st, adata_sc)
df_all_genes
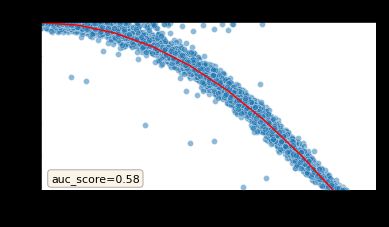
对测试基因的预测可以使用 plot_auc 以图形方式可视化:
# sns.scatterplot(data=df_all_genes, x='score', y='sparsity_sp', hue='is_training', alpha=.5); # for legacy
tg.plot_auc(df_all_genes);

上图是 Tangram 中最重要的验证图。每个点代表一个基因;x 轴表示得分,y 轴表示该基因在空间数据中的稀疏度。毫不奇怪,低分预测的基因代表空间数据中非常稀疏的基因,这表明 Tangram 预测正确地表达了这些基因。请注意,上面观察到的曲线是 Tangram 映射的典型曲线:该曲线下的面积是我们用来评估映射的最可靠的度量。
让我们检查一些预测。其中一些基因在生物学上是稀疏的,但得到了很好的预测:
genes=['tfap2b', 'zic4']
tg.plot_genes_sc(genes, adata_measured=adata_st, adata_predicted=ad_ge, perc=0.02)
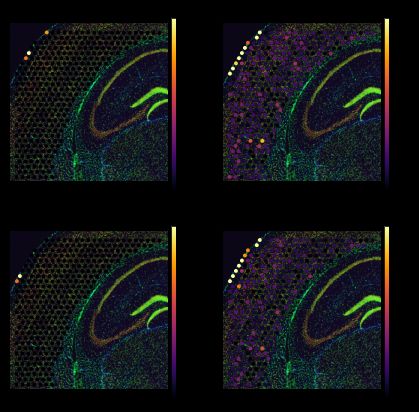
一些非稀疏基因呈现出 Tangram 强调的图案:
genes = ['cd34', 'rasal1']
tg.plot_genes_sc(genes, adata_measured=adata_st, adata_predicted=ad_ge, perc=0.02)
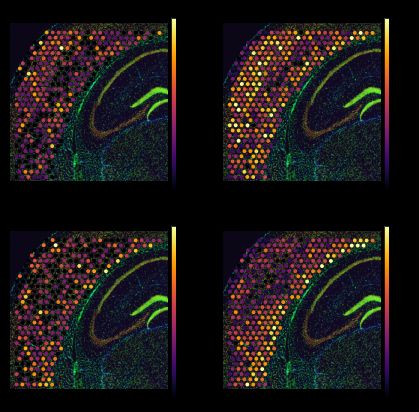
最后,一些未注释的基因具有未知的功能。这些基因通常很难在空间数据中检测到,但 Tangram 可以提供预测:
genes = ['gm33027', 'gm5431']
tg.plot_genes_sc(genes[:5], adata_measured=adata_st, adata_predicted=ad_ge, perc=0.02)

对于非目标空间技术,如 Visium 和 Slide-seq,一个空间体素可能包含多个细胞。在这些情况下,将基因表达分解到单个细胞中可能会很有用,这一过程称为反卷积。反卷积是一个必需的特征,并且也很难通过计算方法准确获得。如果您的目标是研究细胞类型的共定位,我们建议您使用空间细胞类型图。如果您的目标是发现细胞间通信机制,我们建议您计算基因程序,然后使用 project_cell_annotations 在空间上可视化程序的使用情况。无论如何,要继续进行反卷积,请参见下文。
为了对细胞进行反卷积,Tangram 需要知道每个体素中存在多少个细胞。这是通过对相应组织学上的细胞进行分割来实现的,squidpy 通过两行代码实现了这一点:
-
squidpy.im.process应用平滑作为预处理步骤。 -
squidpy.im.segment使用分水岭算法计算分割掩码。
请注意,某些技术(例如 Slide-seq)目前不允许对已分析基因的同一组织载玻片进行染色。对于这些数据,您仍然可以通过粗略估计细胞密度来尝试反卷积 - 通常我们只是传递统一的先验。最后,请注意,反卷积很难验证,因为我们没有 ground truth 的空间分辨单细胞。
sq.im.process(img=img, layer="image", method="smooth")
sq.im.segment(
img=img,
layer="image_smooth",
method="watershed",
channel=0,
)
让我们可视化插图的分割结果
inset_y = 1500
inset_x = 1700
inset_sy = 400
inset_sx = 500
fig, axs = plt.subplots(1, 3, figsize=(30, 10))
sc.pl.spatial(
adata_st, color="cluster", alpha=0.7, frameon=False, show=False, ax=axs[0], title=""
)
axs[0].set_title("Clusters", fontdict={"fontsize": 20})
sf = adata_st.uns["spatial"]["V1_Adult_Mouse_Brain_Coronal_Section_2"]["scalefactors"][
"tissue_hires_scalef"
]
rect = mpl.patches.Rectangle(
(inset_y * sf, inset_x * sf),
width=inset_sx * sf,
height=inset_sy * sf,
ec="yellow",
lw=4,
fill=False,
)
axs[0].add_patch(rect)
axs[0].axes.xaxis.label.set_visible(False)
axs[0].axes.yaxis.label.set_visible(False)
axs[1].imshow(
img["image"][inset_y : inset_y + inset_sy, inset_x : inset_x + inset_sx, 0, 0]
/ 65536,
interpolation="none",
)
axs[1].grid(False)
axs[1].set_xticks([])
axs[1].set_yticks([])
axs[1].set_title("DAPI", fontdict={"fontsize": 20})
crop = img["segmented_watershed"][
inset_y : inset_y + inset_sy, inset_x : inset_x + inset_sx
].values.squeeze(-1)
crop = skimage.segmentation.relabel_sequential(crop)[0]
cmap = plt.cm.plasma
cmap.set_under(color="black")
axs[2].imshow(crop, interpolation="none", cmap=cmap, vmin=0.001)
axs[2].grid(False)
axs[2].set_xticks([])
axs[2].set_yticks([])
axs[2].set_title("Nucleous segmentation", fontdict={"fontsize": 20});

DAPI 和 mask 之间的比较证实了分割的质量。然后,我们需要提取一些对下游反卷积任务有用的图像特征。具体来说:
- 每个点下的独特分割对象(即细胞核)的数量。
- 分割对象的质心坐标。
# define image layer to use for segmentation
features_kwargs = {
"segmentation": {
"label_layer": "segmented_watershed",
"props": ["label", "centroid"],
"channels": [1, 2],
}
}
# calculate segmentation features
sq.im.calculate_image_features(
adata_st,
img,
layer="image",
key_added="image_features",
features_kwargs=features_kwargs,
features="segmentation",
mask_circle=True,
)
我们可以使用 scanpy 可视化每个点下的物体总数。
adata_st.obs["cell_count"] = adata_st.obsm["image_features"]["segmentation_label"]
sc.pl.spatial(adata_st, color=["cluster", "cell_count"], frameon=False)

7 通过对齐进行反卷积
使用 Tangram 去卷积的基本原理是限制映射的单细胞轮廓的数量。这与大多数反卷积方法不同。具体来说,我们将它们设置为等于组织学中分段细胞的数量,如下所示:
- 我们传递
mode='constrained'。这会向损失函数添加一个过滤项和一个布尔正则化器。 - 我们将
target_count设置为等于分段细胞的总数。Tangram 将寻找最佳的target_count细胞以在空间中对齐。 - 我们传递一个
density_prior,其中包含每个体素的细胞分数。
ad_map = tg.map_cells_to_space(
adata_sc,
adata_st,
mode="constrained",
target_count=adata_st.obs.cell_count.sum(),
density_prior=np.array(adata_st.obs.cell_count) / adata_st.obs.cell_count.sum(),
num_epochs=1000,
device="cuda:0",
# device='cpu',
)
像以前一样,我们可以绘制细胞类型图:
tg.project_cell_annotations(ad_map, adata_st, annotation="cell_subclass")
annotation_list = list(pd.unique(adata_sc.obs['cell_subclass']))
tg.plot_cell_annotation_sc(adata_st, annotation_list, perc=0.02)
我们通过检查测试转录组来验证映射:
ad_ge = tg.project_genes(adata_map=ad_map, adata_sc=adata_sc)
df_all_genes = tg.compare_spatial_geneexp(ad_ge, adata_st, adata_sc)
tg.plot_auc(df_all_genes);
这是关键部分,我们将使用前面反卷积步骤的结果。之前,我们计算了每个点下唯一分割对象的绝对数量及其质心。让我们以对 Tangram 有用的正确格式提取它们。在生成的数据框中,每行代表一个分割对象(一个细胞)。我们还有图像坐标以及唯一的质心 ID,它是一个包含点 ID 和数字索引的字符串。Tangram 提供了一个方便的功能,可以将点 ID 和分段 ID 之间的映射导出到 adata.uns。
tg.create_segment_cell_df(adata_st)
adata_st.uns["tangram_cell_segmentation"].head()
## spot_idx y x centroids
## 0 AAATGGCATGTCTTGT-1 5304.000000 731.000000 AAATGGCATGTCTTGT-1_0
## 1 AAATGGCATGTCTTGT-1 5320.947519 721.331554 AAATGGCATGTCTTGT-1_1
## 2 AAATGGCATGTCTTGT-1 5332.942342 717.447904 AAATGGCATGTCTTGT-1_2
## 3 AAATGGCATGTCTTGT-1 5348.865384 558.924248 AAATGGCATGTCTTGT-1_3
## 4 AAATGGCATGTCTTGT-1 5342.124989 567.208502 AAATGGCATGTCTTGT-1_4
我们可以使用 tangram.count_cell_annotation() 将反卷积步骤结果的细胞类型映射到假定的分割 ID。
tg.count_cell_annotations(
ad_map,
adata_sc,
adata_st,
annotation="cell_subclass",
)
adata_st.obsm["tangram_ct_count"].head()
最后将结果导出到新的 AnnData 对象中。
adata_segment = tg.deconvolve_cell_annotations(adata_st)
adata_segment.obs.head()
请注意,AnnData 对象不包含计数,而仅包含细胞类型注释,作为 Tangram 映射的结果。尽管如此,为了可视化目的创建这样的 AnnData 对象还是很方便的。下面您可以欣赏到每个点现在不再是 Visium 点,而是具有映射的细胞类型的单个唯一分割对象。
fig, ax = plt.subplots(1, 1, figsize=(20, 20))
sc.pl.spatial(
adata_segment,
color="cluster",
size=0.4,
show=False,
frameon=False,
alpha_img=0.2,
legend_fontsize=20,
ax=ax,
)

本文由 mdnice 多平台发布